SVAR PÅ SPØRGSMÅL OM DNA, GENER OG GENTEKNOLOGI.
Brug venstrespalten for at finde spørgsmål. (Eller søg med Ctrl-F/Ctrl-B).
Hvis du har kommentarer til denne side, så send en email til: bionyt@gmail.com
/DNA/Modelorganismer/Kolibakterie/Navn/Det korte svar er: En tysk forsker
Den tyske forsker Theodor Escherich, som isolerede bakterier fra bleer af raske babyer og bemærkede den stavformede mikrobe, som han beskrev i 1995. Andre forskere gav siden hen bakterien navnet Escherichia coli. Bog: Microcosm: E. coli and the new science of life - af Carl Zimmer, William Heinemann, London 2008 (lån:FB).
Test dig selv
Du kan tegne abonnement på BioNyt: Videnskabens verden her!
/Celler/Cellekerne/Kromosomer/Det korte svar er: 46 kromosomer
Menneskets har 46 kromosomer. Hvert menneske har et dobbelt antal af nr. 1-22 af kromosomerne (nemlig et kromosomnummer fra hver forældre) samt to kønskromosomer (enten X+X [kvinder], eller X+Y [mænd]). Kvinder har altså to X-kromosomer, mens mænd kun har ét X-kromosom. Det har betydning for gener, som findes på X-kromosomet. Da kvinder har to X-kromosomer, har de altså to af hver af de gener, som sidder på X-kromosomet. Mænd har kun én udgave af disse gener, og hvis det ikke fungerer har mænd altså ikke en ekstra kopi.
Test dig selv
Du kan tegne abonnement på BioNyt: Videnskabens verden her!
font size="1">/Celler/Cellekerne/Kromosomer/Det korte svar er: F.eks. er Kromosom nr.1 på 246.127.941 baseenheder medens Kromosom Y har 50.286.555 baseenheder
Størrelsen af kromosomerne hos mennesket vises nedenfor - Her vises antallet af baseenheder i hver af menneskets kromosomer:
Kromosom nr.: Antal baseenheder.
Kromosom nr.1: 246.127.941 baseenheder
Kromosom nr.2: 243.615.958 baseenheder
Kromosom nr.3: 199.344.050 baseenheder
Kromosom nr.4: 191.731.959 baseenheder
Kromosom nr.5: 181.034.922 baseenheder
Kromosom nr.6: 170.914.576 baseenheder
Kromosom nr.7: 158.545.518 baseenheder
Kromosom nr.8: 146.308.819 baseenheder
Kromosom nr.9: 136.372.045 baseenheder
Kromosom nr.10: 135.037.215 baseenheder
Kromosom nr.11: 134.482.954 baseenheder
Kromosom nr.12: 132.078.379 baseenheder
Kromosom nr.13: 113.042.980 baseenheder
Kromosom nr.14: 105.311.216 baseenheder
Kromosom nr.15: 100.256.656 baseenheder
Kromosom nr.16: 90.041.932 baseenheder
Kromosom nr.17: 81.860.266 baseenheder
Kromosom nr.18: 76.115.139 baseenheder
Kromosom nr.19: 63.811.651 baseenheder
Kromosom nr.20: 63.741.868 baseenheder
Kromosom nr.21: 46.976.097 baseenheder
Kromosom nr.22: 49.396.972 baseenheder
Kromosom X: 153.692.391 baseenheder
Kromosom Y: 50.286.555 baseenheder.
Test dig selv
Du kan tegne abonnement på BioNyt: Videnskabens verden her!
DNA/Det korte svar er: DeoxyRibonukleinAcid(syre) (DNA) er det arvemateriale, som findes i alle celler
DeoxyRibonukleinAcid(syre) (DNA) er det arvemateriale, som findes i alle celler og i de samme mængder i alle celler i samme individ (undtaget sædceller og ægceller, som er haploide celler med halv portion DNA). DNA er en dobbeltstreng af nukleinsyrer og påsat en backbone (rygrad) af et sukker (deoxyribose) og en phosphat. På deoxyribosen er der påsat en af de fire baser Adenin, Thymin, Cytosin eller Guanin. Baserne binder til hinanden parvis, A binder til T og C binder til G. De bindinger som er mellem baserne kaldes for hydrogen-bindinger. Mellem T og C er der to hydrogenbindinger, mens der mellem G og C er tre hydrogenbindinger.
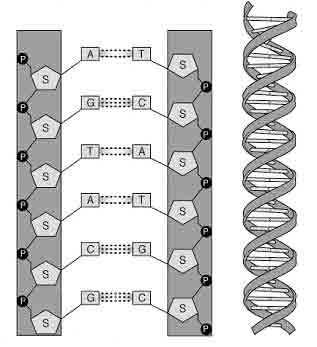
Test dig selv
Du kan tegne abonnement på BioNyt: Videnskabens verden her!
DNA/Det korte svar er: Bl.a. forskel i ribosen
Bindingerne mellem enhederne er ens i DNA og RNA (se her); da RNA har en OH-gruppe, hvor DNA har en H-gruppe (i 2'-stillingen - idet 1'-stillingen er defineret som det sted, hvor basen sidder) kan RNA ændres kunstigt dette sted, hvilket DNA ikke kan.

(Forskellen mellem proteiner og nukleinsyrer (dvs. DNA og RNA) er beskrevet her).
Test dig selv
Du kan tegne abonnement på BioNyt: Videnskabens verden her!
DNA/Det korte svar er: Ved "DNA-smeltepunktet", hvor de hydrogenbindinger, som holder de to antiparallelle strenge samlet, brydes
De hydrogenbindinger, som holder de to antiparallelle strenge samlet, kan brydes. Hvis det sker, bliver DNA´et denatureret. Hydrogen-bindingerne kan enten brydes ved varmepåvirkning eller ved kemisk påvirkning (ved at sænke eller hæve pH-værdien). Det er defineret sådan, at når halvdelen af hele DNA-strengen er denatureret, kaldes det for smeltepunktet (Tm : melting temperature). Smeltetemperaturen er forskellig for forskellige stykker DNA, da Tm er afhængig af antallet af C-G og A-T bindinger. Jo flere C-G bindinger, der er, desto højere er Tm. Det skyldes, at det er tre bindinger, som holder C-G sammen, hvorimod det kun er to bindinger, som holder A-T sammen.
Test dig selv
Du kan tegne abonnement på BioNyt: Videnskabens verden her!
DNA/Det korte svar er: Deoxynukleotid, som mangler en 3´-hydroxylgruppe, og bruges i DNA-studier
En dideoxynekleotid (didN) er en deoxynukleotid, som mangler en 3´-hydroxylgruppe, og som dermed ikke kan danne en 3`-5` phosphodiester binding, som er nødvendig for at forlænge en eksisterende kæde. Dideoxynukleotider benyttes i DNA-sekventering (Sanger´s dideoxy-metode og i behandling af virale sygdomme.
Test dig selv
Du kan tegne abonnement på BioNyt: Videnskabens verden her!
DNA/Det korte svar er: Et nukleosid med en phosphatgruppe betegnes et "nukleotid"
Et nukleosid er en byggeklods i DNA eller RNA, som består af en nitrogen-holdig base og en fem-carbon sukker. Et nukleosid med en phosphatgruppe betegnes et "nukleotid". Tilsammen udgør nukleotiderne alle generne og tildels også en væsentlig del af kromosomerne (som også indeholder proteiner).
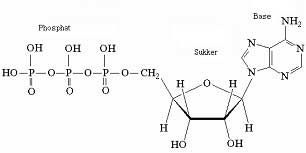
Test dig selv
Du kan tegne abonnement på BioNyt: Videnskabens verden her!
DNA/Det korte svar er: Der er gjort mange uventede fund, bl.a. introns, mobile områder osv.
Det var et uventet fund at gener indeholder intron-områder som skal fjernes for at genet kan fungere. Det var et uventet fund, at de proteinkodende områder kun udgør en mindre del af DNA-strengen. Det var et uventet fund, at der findes et enzym (revers-transkriptase), som kan lave RNA om til DNA. Det var et uventet fund, at der findes mobile områder i DNA-strengen. Det var et uventet fund, at der findes aktivator-områder (enhancers) langt fra det gen, som de påvirker. Fundet af DNA-områder, der koder for små RNA-stykker med forskellige funktioner, var også et uventet fund. Mange andre uventede fund er gjort eller vil blive gjort. (9785s.1) .
Test dig selv
Du kan tegne abonnement på BioNyt: Videnskabens verden her!
DNA/Det korte svar er: Af “baser” forbundet med phosphatgruppe-broer
Hver af de to strenge i en dobbeltstrenget DNA-streng er opbygget af kæder af baserne A, T, C og G, der holdes sammen med phosphatgruppe-broer (mellem 5' carbonatomet i den ene deoxyribosegruppe og 3' carbonatomet i den næste deoxyribosegruppe) (9785s.9)
Test dig selv
Du kan tegne abonnement på BioNyt: Videnskabens verden her!
/DNA/Alder/Bakterier/Det korte svar er: Svaret synes at være "over 500.000 år", omend det ikke nødvendigvis er den samme bakterie
Svaret synes at være "over 500.000 år". Eske Willerslevs gruppe fra Københavns Universitet har fundet liv, der betegnes som verdens ældste levende organismer - bakterier i permafrost - som kan siges stadig at være i live efter en halv million år, idet de har kunnet holde deres DNA intakt i al den tid. Man lavede forskellige test af bakterien, som bl.a. kunne siges at trække vejret ved at danne CO2. Man kunne desuden finde meget lange DNA-stykker, og det ville man ikke forvente, hvis den var død. Bakteriens stofskifte er gået så langsomt i permafrost, at den har været levende i dette kolossale tidsrum. Denne opdagelse satte begrebet “alder” i perspektiv. Det er formentlig ikke til at påvise, om det er den enkelte bakterie, som kan siges at have denne høje alder, men omvendt - hvis alderen ikke kan tilskrives de enkelte bakterier - vil det jo så betyde, at bakteriearterne har kunnet formere sig i denne lange tid.
Test dig selv
Du kan tegne abonnement på BioNyt: Videnskabens verden her!
/DNA/Alder/Bakterier/Det korte svar er: Permafrost på Mars er koldere end på Jorden, og der er påvist bakterier i permafrost på Jorden
Man har i permafrost på Jorden påvist bakterier, som stadig er levende efter 500.000 år i permafrost. Så kan man måske også finde noget på Mars, hvis der er eller har været liv der. Permafrost på Mars er koldere end på Jorden. (ref.9548) .
Test dig selv
Du kan tegne abonnement på BioNyt: Videnskabens verden her!
/DNA/Arv/Det korte svar er: Et barn får både DNA fra moderen og fra faderen, præcist 50 % fra hver. Faderens kromosomer bestemmer kønnet
Et barn får både DNA fra moderen og fra faderen, præcist 50 % fra hver. Så man kan ikke sige, at grunden til, at et barn f.eks. ligner sin mor mest, er fordi barnet har arvet flere gener af moderen.
Det er de såkaldte Mendelske love, som ligger til grund for nedarvningsteorien. Kort fortalt siger Mendels love i en moderne udgave, at generne nedarves som en tro kopi af forældrenes DNA. Undervejs i denne proces kan der forekomme mutationer, som afspejles i afkommet. Dog nedarves alt DNA, som ikke er i cellekernen, direkte fra moderen (nemlig i form af DNA i mitokondrierne). Det er udelukkende faderen, som bestemmer barnets køn, nemlig afhængig af om sædcellen leverer et X- eller et Y-kromosom.
Test dig selv
Du kan tegne abonnement på BioNyt: Videnskabens verden her!
/DNA/Arv/Mutation/Det korte svar er: Mutationer er ændringer i DNA
Mutationer er ændringer i DNA. Der findes mange former for mutationer, og de kan være mere eller mindre alvorlige for organismen. Stille eller tavse eller synonyme mutationer er mutationer, hvor der udskiftes en enkel base, men hvor den aminosyre, som den pågældende codon koder for, ikke bliver anderledes (bemærk, at der er 20 aminosyrer, hvorimod der er 64 forskellige codoner).
Substitutionsmutationer, hvor en base bliver udskiftet med en anden, er den mest hyppige mutation. To former for substitution er mulige: en transition, hvor en pyrimidin-base bliver udskiftet med en pyrimidin, og en transversion, hvor en pyrimidin bliver udskiftet med en purin eller omvendt.
Der findes også deletions-mutationer, hvor en eller flere baser slettes, så hele genet forskubbes. Ydermere findes der en mutationstype, som kaldes insertion, og hvor der indsættes en eller flere baser.
Der findes også en række mere sjældne mutationer, som f.eks. mutagene tautomerer. Dette er når en base, som f.eks. Adenin, bliver omdannet til en imino-tautomer, som gør den i stand til at baseparre med en cytosin.
Test dig selv
Du kan tegne abonnement på BioNyt: Videnskabens verden her!
/DNA/Arv/Mutation/Det korte svar er: Bl.a. kemisk og ved UV-belysning
Der findes en lang række kemikalier, som er mutagene, (dvs. at de kan ændre vores DNA), og mange af kemikalier omgiver vi os dagligt med, som f.eks. i tobaksrøg osv.
Nogle baseanaloger, f.eks. 5-bromuracil og 2-aminopurin, som kan blive inkorporeret i DNA, og disse kan erstatte henholdsvis thymin og en af purinerne.
Der findes også kemiske mutagener, som modificerer de allerede eksisterende baser. F.eks. reagerer salpetersyrling (HNO2) med baser, som indeholder aminogrupper.
Ultraviolet lys kan danne en pyrimidin-dimer. Hvis der er to thymin-baser ved siden af hinanden på den samme DNA-streng, kan disse to danne indbyrdes kemiske forbindelser. I så fald vil al transskription af genet blive blokeret, indtil mutationen er repareret.
Test dig selv
Du kan tegne abonnement på BioNyt: Videnskabens verden her!
/DNA/Arv/Mutation/Det korte svar er: Ved direkte reparation, base-udskiftningsreparation eller nukleotid-udskiftningsreparation
Der findes en række reparationsmekanismer af mutationer. Tre typer af reparation er mulige: en direkte reparation, en base-udskiftningsreparation og en nukleotid-udskiftningsreparation. Under en base-udskiftningsreparation er backbone intakt, idet det kun er basen, som udskiftes. Men under en nukleotid-udskiftningsreparation udskiftes hele nukleotidet (dvs. base + backbone).
Link
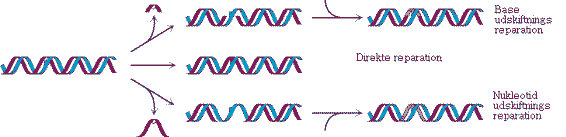
Et eksempel på direkte reparation kunne være en fotokemisk kløvning af en pyrimidin-dimer. Næsten alle celler indeholder et fotoreaktiverende enzym, som kaldes DNA-photolyase.
Test dig selv
Du kan tegne abonnement på BioNyt: Videnskabens verden her!
/DNA/Arv/Mutation/Det korte svar er: Et forsøg, som kan vise, at der opstår mutationer i bakterier
En fluktuationstest er et forsøg, som kan vise, at der opstår mutationer i bakterier. Forsøget går ud på, at man lader en bakterie-kultur vokse op til den stationære fase.
Denne bakterie-opløsning fordeles i to forskellige reagensglas, som yderligere fordeles i henholdsvis 50 små reagensglas og en stor. Efter yderligere dyrkning, fordeles disse bakterie-opløsninger ud på forskellige petriskåle, som podes med en bakterievirus, dvs. en virus, som angriber bakterier. Resultatet viser, at de bakteriekulturer, som blev samlet i et stort reagensglas, alle består af 3-7 kolonier, mens de kulturer, som blev fordelt på forskellige reagensglas, alle har mellem 0 og 100 kolonier. Dvs. at nogle af disse bakterier er muteret til at være modstandsdygtige overfor den bestemte virus.
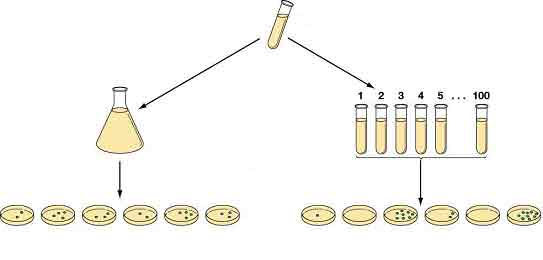
Test dig selv
Du kan tegne abonnement på BioNyt: Videnskabens verden her!
/DNA/Arv/Mutation/Det korte svar er: Ames-testen er en metode til undersøgelse af et stofs mutagene virkning
Ames-testen er en metode til undersøgelse af et stofs mutagene virkning. Ved testen anvendes bakteriemutanter af tyfus-stammer (Salmonella typhimurium), der under påvirkning af mutagene stoffer undergår mutationer, som man kan iagttage ved dyrkning af bakterierne.
Disse tyfus-bakterier har en mutation, så de ikke kan vokse uden at få tilført aminosyren histidin. Påvirkes disse bakterier med et stof, der mistænkes for at være mutagent, er hypotesen, at nogle af bakterierne vil tilbagemutere, så de igen er i stand til at leve uden histidin.
Grunden til, at bakterier er så velegnede som testorganismer, er, at de kun har et kromosom, dvs. at de kun har én udgave af hvert gen. En mutation i et gen vil derfor ikke kunne skjules af et andet allel. En anden grund til at de er velegnede som testorganismer, er deres lille størrelse: Bakterier er så små, at man kan måle meget små mutationshyppigheder, da der i én dråbe næringsbouillon med bakterier findes flere millioner bakterier.
Ames-testen kan udføres på flere forskellige måder, og der kan også fås forskellige former for mutanter, velegnet til hver sin specielle stofpåvirkning. Selv om Salmonella-bakterien er velegnet til undersøgelse af mutagene stoffer, er en bakterie og f.eks. en menneskecelle opbygget meget forskelligt. Mennesket har mange enzymer, der ikke findes hos bakterier, og man kan derfor ikke slutte, at et stof, der ikke virker mutagent på bakterier, nødvendigvis vil være harmløst for mennesker.
I en af variationerne af Ames-testen prøver man at gøre miljøet omkring bakterierne mere lig det miljø, en pattedyrscelle er udsat for. Det gør man ved at blande Salmonella-bakterien med en ekstrakt af rotteleverceller (S9-mix) tillige med det mistænkte stof. Hypotesen er da, at promutagene stoffer af rotteleverenzymerne omdannes til mutagene stoffer, der så påvirker Salmonella-bakterierne, så de muterer.
Læs mere her
Test dig selv
Du kan tegne abonnement på BioNyt: Videnskabens verden her!
/DNA/Arv/Mutation/Det korte svar er: En pr. 100 millioner DNA-byggesten pr. generation (men hurtigere ændringer kan ske i cellerne ved omflytning af DNA-stykker)
Mutationshastigheden er blevet bestemt til at være ca. 1 pr. 100.000.000 nukleotider (DNA-byggesten) pr. generation. Dette er en meget lav mutationshastighed. Denne lave mutationshastighed står i modsætning til at organismerne har mulighed for hurtige ændringer ved at flytte rundt på visse dele af DNA-strengen (mobilt DNA) (9785s.1)
Test dig selv
Du kan tegne abonnement på BioNyt: Videnskabens verden her!
/DNA/Celle/Det korte svar er: 3% af tørstoffet
Rent faktisk udgør DNA ikke en særlig stor procentdel af en celles tørstof, kun ca. 3 %. Der er store mængder RNA i forhold til DNA. Procenten af DNA er den samme i hver celle (undtaget i de haploide celler), mens RNA-niveauet kan variere lidt (en stamcelle i hvile har f.eks. ikke så meget RNA som en celle, der har høj aktivitet).
Fordelingen af tørstof i en celle kan f.eks. være:
Protein = 55 %
rRNA = 16.7 %
tRNA = 3 %
mRNA = 0,9 %
DNA = 3.1 %
Lipider = 9.1 %
Lipopolysaccharider = 3.4 %
Peptidoglycan = 2.5 %: findes kun i bakterier
Vitaminer = 2.9 %
Uorganiske ioner = 1.0 % .
Test dig selv
Du kan tegne abonnement på BioNyt: Videnskabens verden her!
/DNA/Celle/Det korte svar er: B-DNA helix, samt A- og Z-DNA
Der eksisterer tre former for DNA. Opdagerne af DNA, Watson og Crick, fandt den model, som i dag kaldes B-DNA helix. Røntgenkrystallografi har senere afsløret to andre former for DNA, kaldet A- og Z-DNA. A-DNA helix-formen optræder kun, når den relative fugtighed reduceres til under ca. 75 %. A-DNA helix er nemlig en form for dehydreret DNA, dvs. DNA, som ikke er i vandig opløsning.
A- og B-DNA er højre-hånds snoede, antiparallelle strenge, som holdes sammen med hydrogenbindinger. A-helixen er bredere og kortere end B-helixen, og B-helixens baser har en hældning i forhold til helix-aksen, sammenlignet med A-helix, hvis baser er vinkelrette på helix-aksen.
Z-DNA er en venstre-hånds drejet Z-DNA helix, som er opbygget af korte DNA-oligonukleotider af vekslende puriner og pyrimidiner. Denne form findes kun under høje salt-koncentrationer, som tvinger den elektrostatiske frastødning mellem phosphaterne i rygraden tættere sammen.
Under fysiologiske forhold er det meste DNA som B-form.
Her er et link, som opsummerer de tre DNA-former.
Test dig selv
Du kan tegne abonnement på BioNyt: Videnskabens verden her!
/DNA/Celle/Det korte svar er: I ”supercoiling”,
Det DNA, som skal pakkes, vil svare til at pakke 50 meter spindelvæv i en ært, uden at det går i stykker eller bliver filtret. Derudover skal man hele tiden have styr på, hvor de forskellige gener befinder sig. Dette klarer cellerne ved at lave den såkaldte ”supercoiling”, hvilket er det samme som, når en snoet telefonledning snor om sig selv. Derefter kan denne supercoilede DNA vikles rundt om nogle proteiner, som kaldes histoner. Disse basiske proteiner består af fire proteiner, som minder meget om hinanden i den tertiære struktur. Pakning af DNA er af meget stor relevans for regulering af gen-transkriptionen, da polymeraser ikke har adgang til pakket DNA.
Forskellige teknikker til at udforske DNA:
Blotting-teknikker: De teknikker, som kaldes Southern blot og Northern blot, bruges til at separere og karakterisere henholdsvis DNA og RNA. En anden af blottingteknikkerne er Western blot, som bruges til, ved hjælp af antistoffer, at karakterisere proteiner.
3) DNA-sekventering: Den præcise nukletidsekvens af DNA kan bestemmes. Sekventering har været brugt til at skaffe store mængder af information om geners arkitektur, cellens kontrol med genudtrykkelse (expressionen), og proteiners struktur.
4) Solid-phase syntese af nukleinsyrer: Præcise sekvenser af nukleinsyrer kan fremstilles helt fra bunden, eller de kan bruges til at genkende specifikke gen-sekvenser.
5) Polymerase Kæde Reaktion (PCR): Polymerase-kædereaktionen kan fremstille tusindvis af ens DNA-molekyler ud fra et stykke DNA. Et molekyle kan mangfoldiggøres til så store mængder, at de kan bruges til manipulation og karakterisering. Denne PCR-teknik er god til at fremstille meget DNA ud fra materiale, hvor der kun er begrænsede mængder af DNA, f.eks. i fossiler eller vævprøver i kriminalsager (PCR-metoden er beskrevet i BioNyt nr.72).
Test dig selv
Du kan tegne abonnement på BioNyt: Videnskabens verden her!
/DNA/Celle/Det korte svar er: Telomeren er DNA-enden. Et enzym, telomerasen, kan gendanne enden ved syntetisere en seks-baser lang sekvens
Da eukaryoters kromosomer ikke er cirkulære, som i bakterier, så har kromosomerne hver 2 ender. Disse ender består af hundredvis gentagelser af en DNA-sekvens på ca. seks baser. Den ene af strengene består af mange G-baser i 3´-enden, og er lidt længere end den anden streng (link). Hos mennesker er denne streng AGGGTT.
Når denne streng gendannes, sker det ved hjælp af et enzym telomerase, som genkender primer-sekvensen GGTT, og påsætter sekvensen AGGGTT, idet telomerasen syntetiserer denne seks-baser lange sekvens.
- Så efter en cyklus ser det sådan ud: GGTTAGGGTT og efter endnu en cyklus ser det således ud:
GGTTAGGGTTAGGGTT, dvs. at den samme sekvens (AGGGTT) bliver sat på mange gange.
Hele fidusen med telomerase-enzymet ligger i, at den har sin egen "template"-streng med.
Dvs. den syntetiserer GGTTAGGGTT og igen GGTTAGGGTTAGGGTT osv.
Ifølge en teori afsluttes telomeren, dvs. DNA-enden, som vist på denne figur.
Test dig selv
Du kan tegne abonnement på BioNyt: Videnskabens verden her!
/DNA/Celle/Det korte svar er: cDNA er et enkeltstrenget DNA-molekyle, som er dannet ved hjælp af enzymet "revers transkriptase"
cDNA er et enkeltstrenget DNA-molekyle, som er dannet ved hjælp af enzymet "revers transkriptase". Dette enzym er i stand til at lave en DNA-streng ud fra en RNA-streng.
cDNA kan også fremstilles syntetisk ud fra nukleotider.
Test dig selv
Du kan tegne abonnement på BioNyt: Videnskabens verden her!
/DNA/Celle/Det korte svar er: En samling af én organismes DNA-fragmenter
Et DNA-bibliotek er en samling af én organismes DNA-fragmenter, som alle er indsat i en værtsorganisme for på den måde at lagre eller opbevare DNA-fragmenterne i et slags arkiv. Hvis man eksempelvis ønsker at lave et human DNA-bibliotek, tager man det humane DNA og kløver det med restriktions-enzymer i tusindvis af småstykker. Alle disse stykker bliver inkorporeret i forskellige bakteriers plasmider (selvformerende kromosomer), og så har man en samling gener i hver sin egen værtsorganisme, som man i princippet vil kunne styre til at udtrykke genet, hvis man vil.
Læs mere her
Test dig selv
Du kan tegne abonnement på BioNyt: Videnskabens verden her!
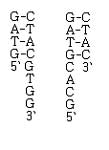
/DNA/Celle/Det korte svar er: En hairpin er en intern, dobbeltstrenget region i en nukleinsyrestreng
En hairpin er en intern, dobbeltstrenget region. Denne region kan kun dannes, hvis der er de rigtige baser ved siden af hinanden. Eksempelvis kan sekvenserne:br>
5`GTAGCTACGTGG-3` og
5`GCACGTAGCTAC-3`
danne interne hairpins på fire baser.
Test dig selv
Du kan tegne abonnement på BioNyt: Videnskabens verden her!
/DNA/Celle/Det korte svar er: Helikase bryder hydrogenbindinger i dobbeltbindingen af DNA, men også andre enzymer medvirker
Udvalgte proteiners funktion
Helikase (dnaB protein): Afvikler dobbelthelixen (bryder hydrogenbindinger)
Primase: Syntetiserer RNA primere
SSB (single strand binding proteins): Sørger for at afviklede strenge ikke danner nye hydrogenbindinger.
DNA gyrase (Topoisomerase II): Indleder negativ supercoiling
DNA polymerase III holoenzym *: Syntetiserer DNA
DNA polymerase I: Fjerner primere og udfylder de tomme pladser
DNA ligase: Klistrer DNA enderne sammen.
(*Holoenzym betyder, at det er et enzym-kompleks, dvs. flere forskellige enzymer i et kompleks).
Test dig selv
Du kan tegne abonnement på BioNyt: Videnskabens verden her!
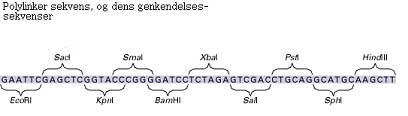
/DNA/Celle/Det korte svar er: En kort DNA sekvens, som består af adskillige restriktions-enzym- genkendelsessekvenser
En polylinker er en kort DNA sekvens, som består af adskillige restriktions-enzyms genkendelsessekvenser, som befinder sig i klonings-vektorer.
Test dig selv
Du kan tegne abonnement på BioNyt: Videnskabens verden her!
/DNA/Celle/Det korte svar er: Udveksling af DNA-sekvenser mellem kromatider
Overkrydsning er udveksling af DNA-sekvenser mellem kromatider i homologe kromosomer under meiosen.
Test dig selv
Du kan tegne abonnement på BioNyt: Videnskabens verden her!
/DNA/Celle/Kerne/Det korte svar er: Korte DNA-stykker med gentagelser
Mikrosatellitter er korte DNA-sekvenser, der består af rækker af gentagelser af typisk 2-4 basepar (Short Tandem Repeats, STR). Det kan f.eks være AATGAATGAATGAATG. Ofte er der mellem 5 og 25 gentagelsesgange. Mikrosatellitter forekommer både i og mellem generne. De bruges som markører for kromosomregioner eller gener. De påvises med PCR-teknik og bruges til at studere evolution, kortlægge gener, påvise genetiske sygdomme og ved retsgenetiske og antropologiske undersøgelser. Mikrosatellit- metoden bliver sjældent brugt på materiale, som er mere end et par årtier gammelt, fordi denne teknologi kan være problematisk, når den anvendes, hvor der kun er lidt DNA-materiale til stede (såkaldt LCNDNA, dvs. lavt kopiantal DNA).
Test dig selv
Du kan tegne abonnement på BioNyt: Videnskabens verden her!
/DNA/Celle/Kerne/Det korte svar er: Kerne-DNA
Det er cellekerne-DNA (nukleært DNA).
Test dig selv
Du kan tegne abonnement på BioNyt: Videnskabens verden her!
/DNA/Celle/Kerne/Det korte svar er: Kernen er med til at holde DNA på et samlet sted, og dette forhindrer store molekylkomplekser og organeller i at komme ind til DNA´et
Der har ofte været diskussioner om, hvad formålet med en kerne er. Kernens membran har porer, som gør det muligt for de fleste makromolekyler frit at bevæge sig ind og ud af kernen. Kernen kan derfor ikke udgøre nogen særlig god beskyttelse imod kemiske stoffer. Da kernemembranen heller ikke er meget tykkere end en phospholipid-membran, er kernen heller ikke nogen god beskyttelse imod stråling osv. Men hvad er så formålet med en kerne egentlig?
Vi ved, at prokaryoter har overlevet uden kerner i meget længere tid end organismer med cellekerne (eukaryoterne) har eksisteret. Det er jo heller ikke sådan, at prokaryoterne har ”bukket under” for eukaryoterne, - begge grupper lever side om side og har stor nytte af hinanden. Er det så en fordel for flercellede organismer at have en kerne? Kan man overhovedet forestille sig flercellede prokaryote organismer?
Kernen er med til at holde DNA på et samlet sted, og dette forhindrer store molekylkomplekser og organeller i at komme ind til DNA´et.
Test dig selv
Du kan tegne abonnement på BioNyt: Videnskabens verden her!
/DNA/Død/Det korte svar er: Normale celler kan ikke som kræftceller fortsætte med at dele sig i det uendelige, idet cellen har en mekanisme til at forhindre uendelig vækst
Alle ved, at organismer, som f.eks. mennesker, er dødelige. Biologer har i lang tid troet, at selv om den enkelte celle dør, vil en cellelinie kunne dele sig i det uendelige. I 1960´erne udførte Leonard Hayflick et forsøg, som viste, at cellelinier af mennesker ikke er udødelige. Disse cellelinier kan kun leve i en afgrænset periode på ca. 50 generationer, hvorefter de alle dør. Grænsen for, hvor lang tid en cellelinie kan overleve, kaldes for Hayflick-grænsen. Men kræftceller (cancerceller) har ikke en sådan grænse, - de vil fortsætte med at dele sig i det uendelige.
Forskere har opdaget, at forskellen mellem almindelige celler og cancerceller er, at de sidstnævnte kan blive ved med at dele sig fordi cancerceller har enzymet telomerase, hvorimod almindelige celler generelt ikke har dette enzym. (Stamceller har dog dette enzym, da det beskytter enderne på det DNA, som de skal give videre)
Dermed kan man se, at cancerceller kan reparere deres telomerer efter hver replikation, hvorimod de fleste almindelige celler ikke kan. Derfor vil almindelige cellers kromosomer bliver kortere og kortere for hver replikation, hvorimod cancerceller vil kunne reparere deres telomerer. Før eller siden vil de almindelige celler miste deres telomerer helt, og deres kromosomer vil så nedbrydes, hvilket vil medføre, at cellen dør. Dette sker ikke for cancerceller, da telomerasen vil redde dem fra den skæbne.
(Kilde: R.F. Weaver Molecular Biology 3.udg., 2005)
Test dig selv
Du kan tegne abonnement på BioNyt: Videnskabens verden her!
/DNA/Forældre/Mitokondrie/Det korte svar er: 1 mutation pr. 1000 år
Som tommelfingerregel opsamles 1 mutation pr. tusind år i mitokondrie-DNA’et, så de menneskeindivider, der vandrer den ene vej, vil opsamle 30 mutationer over 30.000 år, - og de individer, der vandrer den anden vej, vil opsamle 30 andre mutationer over de 30.000 år. Hvis man sammenligner de to grupper efter 30.000 år, vil der være en afvigelse på 60 mutationer i mitokondrie-DNA'et. Ud fra dette kan man finde frem til, hvordan grundstammen må have set ud.
Test dig selv
Du kan tegne abonnement på BioNyt: Videnskabens verden her!
/DNA/Gener/Epigenetik/Det korte svar er: Effekter, der går over flere generationer, antages at skyldes kemiske ændringer af DNA, såsom påsættelse af methylgrupper. Sådanne epigenetiske ændringer troede man tidligere blev nulstillet ved hver generation. Men en undersøgelse viste, at mænd, som begyndte at ryge før puberteten, havde sønner, som i en alder af 9 år var signifikant federe end gennemsnittet. Der var ingen lignende virkning for døtrene. I en anden undersøgelse var bedstefædrenes kost kun koblet til livslængden af sønnerne i anden generation og bedstemødrenes kost var kun koblet til livslængden af døtrene i 2. generation.
Ernæring og rygevaner i begyndelsen af livet kan påvirke sundheden af mænds sønner og sønnesønner viser en ny undersøgelse. Disse overraskende arvelige virkninger mener man skyldes små kemiske ændringer i DNA, som betegnes epigenetiske modifikationer. De kan have store virkninger på befolkningens sundhed, for eksempel kan adfærden hos nutidens børn have problematiske følger for fremtidige generationer. Marcus Pembrey, en kemi-genetiker ved University College London og kolleger i Umeå Universitet i Sverige har på to forskellige måder fundet tegn på, at sundhedsvirkninger kan passere ned ad den mandlige arvelinie. For det første studerede forskerne resultaterne fra en undersøgelse, 'Avon Longitudinal Study of Parents and Children', som er en undersøgelse af britiske par, som fik børn i begyndelsen af 1990’erne. Over 5.000 fædre i undersøgelsen havde været rygere. Af disse mænd begyndte 166 at ryge, før de var 11 år gamle, - det vil sige i en periode før deres pubertet, hvor kroppen er særligt følsom over for miljømæssige stressfaktorer. Den britiske forskergruppe filtrerede dataene for at fjerne indflydelsen af faktorer, såsom socioøkonomisk status og om fædrene var fortsat med at ryge, indtil deres børn blev født. De mænd, som begyndte at ryge før puberteten, havde sønner, som i en alder af 9 år var signifikant federe end gennemsnittet. Der var ingen lignende virkning for døtrene.
Forskerne så også på de historiske data fra et fjernt område i det nordlige Sverige, hvor man fulgte familier tre generationer tilbage og vurderede deres kost ud fra data om høstudbytterne i området. Mennesker, hvis bedsteforældre havde haft begrænset tilgang til føde i en alder mellem 9 år og 12 år, så ud til at leve længere, og denne virkning var kønsspecifik. Bedstefædrenes kost var kun koblet til livslængden af sønnerne i anden generation og bedstemødrenes kost var kun koblet til livslængden af døtrene i 2. generation. Lignende virkninger, som går over flere generationer, er før blevet påvist hos kvinder, men dette er det første tegn på, at sådanne ændringer passerer ned gennem den mandlige arvelinie. Bekymringen er, at dette kan have meget større indflydelse på befolkningens sundhed, end man tidligere har forstået. Dette er et nyt paradigme for medicin for lægevidenskaben, og det forklarer, hvordan vores miljø måske har kunnet påvirke vores sundhed, og også måske vil gøre det i generationer ud i fremtiden, siger Mike Skinner, som er forplantningsbiolog ved Washington State University i Seattle, og som har studeret lignende virkninger i dyr. Effekter, der går over flere generationer, antages at skyldes kemiske ændringer af DNA, såsom påsættelse af methylgrupper. Sådanne epigenetiske ændringer troede man tidligere blev nulstillet ved hver generation, men hen over genomet synes der at være områder, hvor epigenetiske ændringer modstår at blive fjernet eller visket ud. Virkninger, som nedarves i den mandlige linie, kan skyldes epigenetiske ændringer på Y-kromosomet, siger Marcus Pembrey. Når epigenetiske ændringer er nedarvet, kan de tænkes at vare ved i meget lang tid.
Mike Skinner tror, at nedarvede epigenetiske ændringer sandsynligvis vil være en vigtig årsag til sygdom. Flere studier tyder på, at børn i mange lande drikker og ryger i en tidligere alder, således at konsekvenserne kunne gå hen og blive store, hvis virkningerne føres videre til næste generation. Stigningen i fedme og diabetes kan måske også forklares ved nedarvede epigenetiske ændringer. Dette ville være en farlig spis-og-se-virkningen-i-fremtiden-mekanisme, siger Rob Waterland, som studerer ernæring og epigenetik ved Baylor College of Medicine i Houston i Texas. (kilde: New Scientist nr.2533, 6 jan.2006, s.10 ).
Test dig selv
Du kan tegne abonnement på BioNyt: Videnskabens verden her!
/DNA/Gen/Intron/Det korte svar er: DNA, som ikke indeholder gener (men som kan indvirke på aflæsningen af generne)
Vores (menneskers) celler indeholder ca. 1000 gange mere DNA end en E.coli kolibakterie, men det er ikke ensbetydende med, at vi har 1000 gange flere gener. I forhold til bakterier har de fleste komplekse eukaryote organismer store mængder ikke-kodende DNA. Det formodes, at omkring 97 % af alt DNA i en eukaryot organisme ikke koder for proteiner eller for aktive RNA-molekyler. Junk-DNA er en betegnelse for alt det ikke-kodende DNA.
(Kilde: R.F. Weaver Molecular Biology 3.udg., 2005)
Test dig selv
Du kan tegne abonnement på BioNyt: Videnskabens verden her!
/DNA/Gen/Intron/Det korte svar er: Områder i DNA'ets gen-områder, som ikke bruges til aflæsning af gen-produktet (proteinet)
De fleste gener er opbrudt i afsnit, sektioner, af DNA-strengen, og disse afsnit af DNA-strengen kaldes intron-områder (introns). Disse områder skal fjernes på RNA-trinnet for at generne kan blive funktionsdygtige. Stykkerne, som bevares i genet, kaldes exon-områder (exons). Intron-områderne blev opdaget i slutningen af 1970'erne. (9785s.1-2)
Test dig selv
Du kan tegne abonnement på BioNyt: Videnskabens verden her!
/DNA/Gen/Intron/Det korte svar er: 30%
Intron-områderne udgør ca. 30% af genomet hos mennesker og pattedyr. Exon-områderne udgør kun 1-2 % af menneskets genom. (9785s.2)
Test dig selv
Du kan tegne abonnement på BioNyt: Videnskabens verden her!
/DNA/Gen/Intron/Det korte svar er: 98% (nyere forskning har dog modificeret denne opfattelse)
98% af menneskets genom er junk-DNA ifølge den tidligere opfattelse af junk-DNA. Senere har man dog opdaget, at der i dette såkaldte junk-DNA (spild-DNA) findes områder, som har betydning. Man ved nu at mindst 50% af menneskets genom-DNA består af mobile elementer (transposable elementer) som er rester efter hoppende DNA gennem hundreder af millioner år. Kun reletivt få af disse er stadig mobile elementer. Imidlertid har ny forskning vist at både de stadig mobile elementer og mange af de ikke mere mobile elementer stadig har en funktion. Denne funktion er dog kun til stede på den måde, at disse områder kan influere de aktive gener over evolutionær tid - men det er ikke blevet demonstreret, at disse områder har betydning for det enkelte individ. Desuden findes der i dette “junk-DNA” også områder, som koder for små RNA-stykker af forskellige typer og med forskellige funktioner. Der er f.eks. fundet aktiverende områder (enhancers) langt fra de gen, som de påvirker. (9785s.2)
Test dig selv
Du kan tegne abonnement på BioNyt: Videnskabens verden her!
/DNA/Gen/Intron/Exon/Det korte svar er: 1-2%
Exon-områderne udgør kun 1-2 % af menneskets genom. Intron-områderne udgør ca. 30% af genomet hos mennesker og pattedyr. (9785s.2)
Test dig selv
Du kan tegne abonnement på BioNyt: Videnskabens verden her!
/DNA/Gen/Kræftgen/Det korte svar er: p53 er et tumorrepressor-gen, som spiller en stor rolle inden for kræftforskningen
p53 er et gen, som spiller en stor rolle inden for kræftforskningen. p53-genet er et såkaldt tumorsuppressor-gen, dvs. et gen, som når det er aktivt stopper for tumorudvikling. Hvis en person kun har én kopi af p53-genet, vil der være meget stor sandsynlighed for, at personen i løbet af sit voksne liv udvikler tumorer et eller flere steder i kroppen. Denne tilstand er sjælden og kaldes "Li-Fraumeni syndromet". Læs mere her.
p53-proteinet binder til DNA, og derved stimuleres et andet gen til at producere et p23-protein. Dette p23-protein indgår i en kompleksdannelse med endnu et protein, som kaldes "protein cell division stimulating protein" (cdk2). Når denne kompleksdannelse har fundet sted, kan cellen ikke gå til den næste fase af celledelingen. En mutation i p53-genet medfører, at p23-genet ikke bliver udtrykt, og dermed bliver der ikke dannet et kompleks. I det tilfælde vil celledelingen være uden for kontrol, og der vil kunne dannes en tumor.
Test dig selv
Du kan tegne abonnement på BioNyt: Videnskabens verden her!
/DNA/Genom/Det korte svar er: 3200 millioner
Det humane genom er opbygget af 3,2 milliarder baser. Langt det meste af vores arvemasse (genomet) findes i cellekernens kromosomer, og cellekerne-genomet kaldes også "det nukleære genom". I menneskets celler er der 23 par kromosomer, altså i alt 46 kromosomer. Da ét par er X og Y (de to forskellige kønskromosomer) består menneskets kernegenom af 24 forskellige, lineære DNA-molekyler, der tilsammen er opbygget af 3,2 milliarder baser.
Test dig selv
Du kan tegne abonnement på BioNyt: Videnskabens verden her!
/DNA/Genom/Det korte svar er: Kerne-DNA
Det er cellekerne-DNA (nukleært DNA).
Test dig selv
Du kan tegne abonnement på BioNyt: Videnskabens verden her!
/DNA/Genom/Det korte svar er: Få individer i evolutionære flaskehals-perioder
Årsagen forklares med gentagne "flaskehalse" i menneskets evolution: De gentagne "flaskehalse", som kendetegner det moderne menneskes 150.000 årige historie har medført, at selv vidt adskilte befolkningsgrupper ligner hinanden genetisk. Faktisk findes det meste af den genetiske variation inden for den enkelte population, og det meste af denne variation er opstået inden opsplitningen i Afrika for 60.000-70.000 år siden (ref.9726).
Test dig selv
Du kan tegne abonnement på BioNyt: Videnskabens verden her!
/DNA/Genom/Det korte svar er: Et genom er den samlede arvemasse i en celle (f.eks. chimpanse-genomet, menneske-genomet)
Et genom er den samlede arvemasse i en celle. Dvs. det kan være alt det DNA, som findes i kromosomerne, samt det DNA, som findes i eventuelle organeller (mitokondrier, grønkorn (planter), osv.).
Test dig selv
Du kan tegne abonnement på BioNyt: Videnskabens verden her!
/DNA/Genom/Det korte svar er: Den procentvise mængde af et givet allel
Med et gens genfrekvens menes den procentvise mængde af et givet allel, i en bestemt population.
Test dig selv
Du kan tegne abonnement på BioNyt: Videnskabens verden her!
/DNA/Genom/Det korte svar er: Den tilfældige variation i gen-frekvensen fra mellem generationerne
Med "genetisk drift" menes den tilfældige variation i gen-frekvensen fra en generation til en senere generation.
Test dig selv
Du kan tegne abonnement på BioNyt: Videnskabens verden her!
/DNA/Genom/Det korte svar er: Det varierer, f.eks. fra 10-3000 millioner enheder.
Bagegær har et genom på 12 Mb (12 mill. enheder), svampedyret Dictyostelium har 34 Mb, rundormen Caenorhabditis 100 Mb, Gåsemadplanten Arabidopsis thaliana 125 Mb, bananfluen Drosophila 180 Mb, malariamyggen Anopheles 278 Mb, pufferfish Takifugu 400 Mb, ris 400 Mb, mus 2500 Mb, majs 3200 Mb, mennesket 3000 Mb. (9785s.7)
Test dig selv
Du kan tegne abonnement på BioNyt: Videnskabens verden her!
/DNA/Genom/Allel/Det korte svar er: DNA-område i et gen der varierer i sekvens
Et gen eller en bestemt DNA-sekvens kan hos forskellige individer optræder lidt forskelligt, og man taler da om forskellige "alleler". Et gen kan f.eks. have 5 eller 10 forskellige alleler. Diploide organismer (med en far og en mor) har ofte arvet to forskellige alleler af et gen.
Test dig selv
Du kan tegne abonnement på BioNyt: Videnskabens verden her!
/DNA/Genom/Allel/Det korte svar er: Manglende evne til at påvise alle allelvarianter i en test
Ved DNA-opformering er “allel-tab” (“allelic dropout”) en bekymring for forskerne. Hvis DNA kun findes i lavt kopiantal, vil den tilfældige fordeling af DNA i de første opformeringscyklusser i PCR-reaktionen let forårsage, at en af de to alleler “oversvømmer” PCR-reaktionen, og helt undertrykker opformeringen af den anden allel. Resultatet vil være, at den genetiske profil ligner en homozygot, uanset den oprindelige genotype er heterozygot. Omfanget af allel-tab kan variere betydeligt fra prøve til prøve, samt mellem forskellige primere og mellem alleler for samme mikrosatellit-markør. Problemets omfang på afgøres fra sag til sag. Man bestemmer, hvor mange PCR-opformeringer, det er nødvendigt at lave, for at genotypen kan vurderes med sikkerhed. Det kaldes mange-rørs-metode (“multiple tubes approach”).
Test dig selv
Du kan tegne abonnement på BioNyt: Videnskabens verden her!
/DNA/Genom/Hest/Det korte svar er: Ja, for flere hesteracer tilmed
Eske Willerslevs afdeling har sekvensbestemt hele genomet fra en 700.000 år gammel hest, samt hele genomet fra en 13.000 år gammel hest. Desuden har de sekvensbestemt hele genomet hos æslet samt hos 5- 7 hesteracer. Det giver oplysninger om hestens evolution - men også om tæmningen af hesten (domesticeringen) - og kan belyse spørgsmål som: Hvilke egenskaber ved de forskellige hesteracer blev der selekteret for? Hvilke egenskaber er oprindelige og hvilke er nyere?
Test dig selv
Du kan tegne abonnement på BioNyt: Videnskabens verden her!
/DNA/Genom/Menneske/Nutids/Indianere/Sitting_bull/Det korte svar er: Lederen af sioux-folket
Eske Willerslev har fået tilbudt at lave DNA-analyse af hår fra Sitting Bull, der er et ikon for indianerne i Nordamerika (ref.9556). Der er i øvrigt flere familier i USA, der påstår, at de er den rigtige familie nedstammende fra Sitting Bull, lederen af sioux-folket. Der har været retssager - og den familie, der vant retssagen, har Eske Willerslev fået mundswabs fra - bl.a. af oldebørn. I princippet kan han vise, om retssagen endte retfærdigt( Sitting Bull, 1882. Et symbol på kampen mod de hvide, fordi han nægtede at underskrive en fredsaftale. I 1876 ved Little Bighorn-floden i Montana blev et indianermøde angrebet af oberstløjtnant Custers soldater, som blev udslettet. Sitting Bull flygtede med sin stamme til Canada, men sult tvang stammen til i 1881 at vende tilbage til USA og overgive sig. Han medvirkede i Wild West Show, bl.a. på en turné til Europa. I 1890 opstod en åndedansbevægelse, som de hvide gjorde ham ansvarlig før, skønt han ikke var en del af bevægelsen. Under en anholdelse satte han sig til modværge og blev skudt og dræbt.
Test dig selv
Du kan tegne abonnement på BioNyt: Videnskabens verden her!
/DNA/Genom/Majs/Det korte svar er: Studiet er endnu ikke afsluttet
Tom Gilbert har planlagt studier af, hvordan majs har ændret sig genetisk og proteinmæssigt i løbet af den tid, planten er blevet brugt som afgrøde, hvor den gennem 10.000 år er ændret fra vilde former til en i dag i sammenligning totalt uigenkendelig majsafgrøde (ref.9588). Ved direkte at observere forandringerne i indsamlet DNA og proteiner fra forhistorisk materiale kan man få indsigt i processen. Projektet kræver samarbejde med genetikere og arkæologer i USA og Mexico - områder, som er majsplantens hjemsted. Måske vil man kunne genindføre variationer i landsorterne og dermed overføre modstand mod sygdomme, siger han. Det vil være verdens første forskningsprogram med direkte fokus på at udforske majsarternes udvikling gennem en kombineret fossil DNA/protein-tilgang.
Test dig selv
Du kan tegne abonnement på BioNyt: Videnskabens verden her!
/DNA/Genom/Mammut/Det korte svar er: Man har sekvensbestemt aminosyrerne i proteiner i en 43.000 år gammel uldhåret mammut
Det er ikke altid DNA, der studeres: Det er lykkedes forskerne på Eske Willerslevs afdeling at sekvensbestemme aminosyrerne i proteiner i en 43.000 år gammel uldhåret mammut, og bl.a. påvise, at dens blod indeholdt albumin, som var 2 aminosyrer forskelligt fra albuminet hos den nulevende afrikanske og indiske elefant. Dette åbner for en helt ny forskningsgren: paleoproteomics - studiet af gammelt protein (ref.9580).
Test dig selv
Du kan tegne abonnement på BioNyt: Videnskabens verden her!
/DNA/Genom/Mammut/Det korte svar er: 3700 år siden
Den uldhårede mammut levede på Wrangel-øen nord for østsibirien indtil for 3700 år siden, dvs. flere århundreder efter at Ægyptens pyramider blev bygget (ref.9747).
Test dig selv
Du kan tegne abonnement på BioNyt: Videnskabens verden her!
/DNA/Genom/Menneske/Det korte svar er: Man har genombestemt et 4000 år gammelt menneske
Eske Willerslevs gruppe fra Københavns Universitet på Københavns Universitet har (som de første) sekvensbestemt genomet af forhistorisk menneske, nemlig genomet af en mand, der levede i Grønland for 4000 år siden og som tilhørte en ny uddød befolkningsgruppe.
Test dig selv
Du kan tegne abonnement på BioNyt: Videnskabens verden her!
/DNA/Genom/Menneske/Det korte svar er: 46
Langt det meste af vores arvemasse (genomet) findes i cellekernens kromosomer. Cellekerne-genomet kaldes det nukleære genom. I menneskets celler er der 23 par kromosomer, altså i alt 46 kromosomer. Da ét par er X og Y (de to forskellige kønskromosomer) består menneskets kernegenom af 24 forskellige, lineære DNA-molekyler, der tilsammen er opbygget af 3,2 milliarder baser.
Test dig selv
Du kan tegne abonnement på BioNyt: Videnskabens verden her!
/DNA/Genom/Menneske/Det korte svar er: 24 (nemlig 22 autosomer, 2 kønskromosomer)
Da ét par af menneskets kromosompar er X og Y (de to forskellige kønskromosomer) består menneskets kernegenom af 24 forskellige, lineære DNA-molekyler, der tilsammen er opbygget af 3,2 milliarder baser. Langt det meste af vores arvemasse (genomet) findes i cellekernens kromosomer. Cellekerne-genomet kaldes det nukleære genom. I menneskets celler er der 23 par kromosomer, altså i alt 46 kromosomer. 22 forskellige autosomer i par, samt 2 kønskromosomer.
Test dig selv
Du kan tegne abonnement på BioNyt: Videnskabens verden her!
/DNA/Genom/Menneske/Det korte svar er: 3,2 milliarder
Det humane genom er opbygget af 3,2 milliarder baser. Langt det meste af vores arvemasse (genomet) findes i cellekernens kromosomer, og cellekerne-genomet kaldes også "det nukleære genom". I menneskets celler er der 23 par kromosomer, altså i alt 46 kromosomer. Da ét par er X og Y (de to forskellige kønskromosomer) består menneskets kernegenom af 24 forskellige, lineære DNA-molekyler, der tilsammen er opbygget af 3,2 milliarder baser.
Test dig selv
Du kan tegne abonnement på BioNyt: Videnskabens verden her!
/DNA/Genom/Menneske/Det korte svar er: Omkring 25.000 – 30.000 gener i mennesket
For måske bare 10-15 år siden troede man, at der fandtes over 1 million forskellige gener, men efter at det humane genom er blevet kortlagt (projekt HUGO) er man kommet frem til, at der findes ca. 25.000 – 30.000 gener i mennesket.
Test dig selv
Du kan tegne abonnement på BioNyt: Videnskabens verden her!
/DNA/Genom/Menneske/Det korte svar er: Nogle områder har mange gener, andre områder er nærmest tomme for gener
Internettets genom-browser viser et udsnit af genomet. Når der er markeringer, er det tegn på at der er fundet gener. Nogle områder har mange gener, andre områder er nærmest tomme for gener.
Test dig selv
Du kan tegne abonnement på BioNyt: Videnskabens verden her!
/DNA/Genom/Menneske/Det korte svar er: De er opstået ved gentagne duplikeringer
Det er tydeligt ud fra genernes opbygning, at tæt-beliggende gener er opstået ved gentagne duplikeringer, fordi deres størrelse og opbygning ligner hinanden. Nogle af de største af disse duplikerede genfamilier er hox-generne, som styrer kropsbygningen, f.eks. hvor ben og arme dannes på kroppen. Genfamilien for lugtesansen er også meget stor.
Test dig selv
Du kan tegne abonnement på BioNyt: Videnskabens verden her!
/DNA/Genom/Menneske/Det korte svar er: I juni 2004 var der fundet 2.843.433.602 sekvensbestemte baser i menneskets genom, svarende til ca. 90%
Humangenomet, som kan ses på den omtalte side på Internettet, dækker 99% af de genholdige områder af genomet, ialt var der i juni 2004 fundet 2.843.433.602 sekvensbestemte baser. Der er under 1 fejl pr. 10.000 baser; dvs. at præcisionen er 99,99%.
Test dig selv
Du kan tegne abonnement på BioNyt: Videnskabens verden her!
/DNA/Genom/Databaser/Det korte svar er: Database med angivelse af gener
Internettet har gjort det muligt at dele data med resten af verden på meget kort tid. Derfor er der på Internettet i dag store databaser, hvor det er muligt for forskere hurtigt at finde et gen i en "genbank". I genbanker kan man finde information om et bestemt gen, oplysninger om dets eventuelle proteinprodukt, data om på hvilket kromosom genet sidder, og en liste over alle artikler, som er skrevet om det pågældende gen.
En af de største genbank-databaser findes på hjemmesiden under National Center for Biotechnology Information, NCBI: www.ncbi.nlm.nih.gov/entrez/query.fcgi?db=Nucleotide. I juni 2005 indeholdt genbanken over 89.000.000.000 baser (89 milliarder).
Test dig selv
Du kan tegne abonnement på BioNyt: Videnskabens verden her!
/DNA/Genom/Databaser/Det korte svar er: I genbanker kan man finde Internetbaseret information om et bestemt gen
Internettet har gjort det muligt at dele data med resten af verden på meget kort tid. Derfor er der på Internettet i dag store databaser, hvor det er muligt for forskere hurtigt at finde et gen i en "genbank". I genbanker kan man finde information om et bestemt gen, oplysninger om dets eventuelle proteinprodukt, data om på hvilket kromosom genet sidder, og en liste over alle artikler, som er skrevet om det pågældende gen.
En af de største genbank-databaser findes på hjemmesiden under National Center for Biotechnology Information, NCBI: www.ncbi.nlm.nih.gov/entrez/query.fcgi?db=Nucleotide. I juni 2005 indeholdt genbanken over 89.000.000.000 baser (89 milliarder).
Test dig selv
Du kan tegne abonnement på BioNyt: Videnskabens verden her!
/DNA/Genom/Databaser/Det korte svar er: I størrelsesordenen 200 arter
I 2012 kendte man genomerne hos mindst 190 arter. Heraf også mange pattedyr såsom mennesket, chimpanse, hund, ko, mus, rotte, næbdyr. (9785s.14)
Test dig selv
Du kan tegne abonnement på BioNyt: Videnskabens verden her!
/DNA/Genom/Databaser/Det korte svar er: Kortlægningen af forskellige arters genom kan følges på Internettet
Kortlægningen af menneskets genom kan følges på Internettet. Blandt andet på denne adresse. (se Internetkilderne nedenfor). Denne særlig anbefalede Internetside blev oprindelig lavet af Jim Kent som lavede animationsprogrammer til computere og var interesseret i computerkunst. Han blev forsker ved University of California - Santa Cruz (UCSC), og fik en PhD i fra The Zahler Lab ved UCSC.
Test dig selv
Du kan tegne abonnement på BioNyt: Videnskabens verden her!
/DNA/Genom/Databaser/Det korte svar er: Kortlægningen af menneskets genom kan følges på Internettet
På samme Internet-adresse ses generne hos chimpansen, hønen, musen, rotten, Fugu-fisken, bananfluen, to arter af rundorm, gær og SARS-virusset. Man kan i databasen se, hvor generne findes på kromosomerne. Udviklingen i kortlægningen kan altså følges på Internettet, bl.a. på adressen: www.genome.ucsc.edu. På denne hjemmeside kan du selv foretage sammenligninger mellem menneskets genom og genomet hos chimpansen, mus (mouse), rotten (rat), høns (chicken), Fugu-fisken, Drosophila-bananfluen, en rundorm (C. briggsae = Caenorhabditis briggsae) og gærsvamp (yeast). Du kan selv finde gener, som er fælles, og gener som er specielle. Bl.a. kan du forsøge at finde gener, som kun findes hos mennesket og chimpansen, eller måske kan du være heldig at finde gener, som kun findes hos mennesket:
Test dig selv
Du kan tegne abonnement på BioNyt: Videnskabens verden her!
/DNA/Genom/Databaser/Det korte svar er: På http://genome.ucsc.edu/cgi-bin/hgBlat
Søgning i databasen efter en bestemt sekvens, f.eks. "AACTGAA": sker ved at søge på : denne internetadresse [skriv teksten i feltet UPLOAD SEQUENCE].
Test dig selv
Du kan tegne abonnement på BioNyt: Videnskabens verden her!
/DNA/Genom/Databaser/Det korte svar er: Man kan under søgningen skjule nogle oplysninger for at skærmbilledet ikke bliver for overvældende med oplysninger
Under søgningen i databasen kan man foretage forskellige valg, f.eks. skjule nogle oplysninger, for at skærmbilledet ikke bliver for overvældende med oplysninger, eller f.eks. kan man bede om at få vist, hvor f.eks. humangenomet adskiller sig fra chimpansens genom. Man havde i juni 2004 fundet 28.889.041 baseforskelle mellem mennesket og chimpansen blandt menneskets 2.843.433.602 baseenheder (Disse 28.889.041 forskelle svarede altså til godt 1%).
UCSC's genom-browser giver hurtige og pålidelige oplysninger om de dele af genomet, som man ønsker oplysning om. Samtidig gives bemærkninger, "annotation tracks", om det pågældende område af genomet. Det kan f.eks. være hvilke kendte gener, som findes i området, forudsigelser om gener (ud fra fundne gener hos andre dyrearter), gensonder fra området - de såkaldte "EST" eller "expressed sequence tags".
Test dig selv
Du kan tegne abonnement på BioNyt: Videnskabens verden her!
/DNA/Genom/Databaser/Det korte svar er: Det er korte cDNA-fragmenter
EST er korte cDNA-fragmenter, som oversættes til mRNA, som er grundlaget for dannelse af proteiner [mRNA indeholder koden for aminosyrerækkefølgen i proteiner]. EST-fragmenterne er altså relevante, fordi de genkender dele af genomet, som faktisk aflæses til brugbart mRNA (dvs. fra aktive gener), og de er dermed også markører for de kodende områder af genomet, dvs. at de viser vejen til gener.
Test dig selv
Du kan tegne abonnement på BioNyt: Videnskabens verden her!
/DNA/Genom/Databaser/Det korte svar er: Hvis man finder en EST-sekvens i et gen kan man lokalisere genet.
Hvis man finder en EST-sekvens i et gen kan man lokalisere genet. Men dermed ved man stadig ikke, hvilken funktion genet har.
Test dig selv
Du kan tegne abonnement på BioNyt: Videnskabens verden her!
/DNA/Genom/Databaser/Det korte svar er: Det er øer af DNA-strengen, som er rige på C [cytosin] og G [guanin]
Databasen kan give oplysninger om de såkaldte "CpG islands" (islands er det engelske ord for "øer"), hvilket er slang for C-plus-G, dvs. C+G; altså øer af DNA-strengen, som er rige på C [cytosin] og G [guanin]. C og G er baser, og DNA-strengen er opbygget af de fire baser A [adenin], T [thymin] samt . C [cytosin] og G [guanin]. C kan kun binde sig til G (og A kan kun binde sig til T). A og T kan altså ikke binde sig til C eller G. DNA'et er dobbeltstrenget, og når der er et A på den ene streng vil der altså være et T på den anden. Tilsvarende - når der er C på den ene streng, vil der være G på den anden; deraf betegnelsen C+G øer.
Test dig selv
Du kan tegne abonnement på BioNyt: Videnskabens verden her!
/DNA/Genom/Databaser/Det korte svar er: Områder med særlig mange C'er og G'er anses , f.eks. GG eller GGG, altså to-tre G-baser lige efter hinanden, menes at kunne tiltrække huller efter elektroner, som er slået bort, og derved skadet DNA-strengen
Når der i et område er særlig mange C'er og G'er anses dette for en vigtig oplysning, fordi GG eller GGG, altså to-tre G-baser lige efter hinanden menes at kunne tiltrække huller efter elektroner, som er slået bort, og derved skadet DNA-strengen. Hullet kan flytte sig til nabobasen, og videre igen, indtil det når frem til et GG eller GGG, som lettere accepterer hullet [og som derved får et mutation - og ifølge en teori er dette en måde, hvorpå mutationer bortledes væk fra genet på]. CpG-øer findes ofte i 5'-regionen af generne hos hvirveldyrs gener, og derfor kan C+G bruges til at pege på mulige gener i genomsekvensen. C+G-områderne er modstandsdygtige mod methylering, og har tendens til at være forbundet med genet, som hyppigt er aktive i cellen, dvs. som afkodes til dannelse af mRNA - og derefter dannelse af det kode-tilsvarende protein.
Test dig selv
Du kan tegne abonnement på BioNyt: Videnskabens verden her!
/DNA/Genom/Databaser/Det korte svar er: I genom-browseren kan man indsætte sige egne "tracks" (spor), f.eks. for at bruge det i forskning
Når man bruger genom-browseren kan man indsætte sige egne "tracks" (spor), f.eks. for at bruge det i undervisning, eller for at bruge det til forskning.
Test dig selv
Du kan tegne abonnement på BioNyt: Videnskabens verden her!
/DNA/Genom/Databaser/Det korte svar er: I genomdatabasen kan man bl.a. zoome ind på et bestemt gen for at se hvordan det spaltes af spaltningsenzymer
Genom-browseren fra Santa Cruz viser også, hvor der er de såkaldte kromosombånd, og om der er lignende gensekvenser hos mus og andre dyr. Som det fremgår videre af vejledningen til genombrowseren i Santa Cruz, kan brugeren se på et helt kromosom for at få en fornemmelse for gentætheden, åbne et specifikt cytogenetisk bånd for at se en genkandidat for en sygdom, hvis man mener at kende eller gætter på genets position, eller man kan zoome ind på et bestemt gen for at se hvordan det spaltes af spaltningsenzymer. Klik på dele af kromosomet åbner op for information om stedet.
Humangenomprojektet er hovedsagelig udført af følgende institutioner:
The University of Oklahoma, USA.
Chromosome 21 Consortium
Gesellschaft für Biotechnologische Forschung (GBF), Braunschweig, Germany
Institute of Molecular Biology (IMB), Jena, Germany
Washington University School of Medicine Genome Sequencing Center, St Louis, USA
RIKEN Human Genome Research Group, Japan
Stanford Human Genome Center, Palo Alto, CA, USA
Broad Institute of MIT i samarbejde med Harvard University, USA
Beijing Human Genome Center, Beijing, China
Keio University, Tokyo, Japan
University of Washington Genome Center, Seattle, WA, USA
Genome Therapeutics Corporation (GTC), Waltham, MA, USA
Genoscope National Sequencing Centre, Evry, France
Baylor College of Medicine Human Genome Sequencing Center, Houston, TX, USA
Joint Genome Institute (JGI), U.S. Department of Energy, Walnut Creek, CA, USA
Max Planck Institute for Molecular Genetics, Berlin, Germany
The Sanger Institute, Cambridge, UK.
Institute for Systems Biology (ISB), Seattle, WA
The Institute for Genomic Research (TIGR), Rockville, MD, USA
Internetkilder: 3264 (UCSC Genome Bioinformatics)
Gå direkte til genom-søgeren; vælg genom (menneske, chimpanse, mus, rotte, høns, fisk, flue, 2 orme, gær, SARS-virus - samt bavian, kat, hund, ko, svin, zebrafisk)
Gå direkte til menneskets genom (vælg position [f.eks. "target1:1-1,000,000"] og bredde [f.eks. "900"], der skal vises)
Andre kilder: 3242: New Scientist 15/3-2004 s.41.
3243: Science bd.296 s.1276
3244: New Scientist 31. jan. 2004 s.42
3245: New Scientist 21 feb. 2004 s.36-39.
Test dig selv
Du kan tegne abonnement på BioNyt: Videnskabens verden her!
/DNA/Genom/Moa/Det korte svar er: Vingeløs kæmpefugle, uddøde
Moa-fugleslægten omfattede 9 arter af store planteædende skovfugle på 15- 250 kg. De levede over det meste af New Zealand, men uddøde 200 år efter polynesiernes ankomst sidst i 1200- tallet. (Den ældste menneske-boplads på New Zealand er Wairau Bar, som er dateret til år 1290).
Test dig selv
Du kan tegne abonnement på BioNyt: Videnskabens verden her!
/DNA/Genom/Moa/Det korte svar er: Deres slægtskab, kønsforskelle, arter
For ca. 500 år siden uddøde alle de usædvanlige og kæmpestore moa-arter i New Zealand. Moa'erne mindede om strudse, men manglede helt vinger. Der var forskellige arter (mindst 9 arter), og individerne af Dinornis-arter af moa-gruppen var mest spektakulære. De adskilte sig markant i størrelse. Der kunne være 1-2 m højdeforskel og vægten varierede fra 34 til 242 kg. Det førte til, at man troede at der var flere arter end der var. Studier af mitokondrie-DNAsekvenser viste imidlertid, at der kun var en Dinornis-art på Nordøen, og en anden Dinornis-art på sydøen. Desuden fandt man overraskende ud af, at en “lille art” (som man havde kaldt Dinornis struthoides) faktisk blot var hannerne af de to nævnte arter. Det lykkedes nemlig en forskergruppe, hvori Eske Willerslev deltog, at påvise cellekerne-DNA-sekvenser, som viser kønnet på individerne. Man kunne dermed kønsbestemme de fossile knogler.
Kønsbestemmelse ved DNA havde man aldrig tidligere kunnet gøre på fossile knogler. (Det har vist sig, at cellekerne-DNA fra moa, såsom kønsbestemmelses-markører og mikrosatellit-DNA, kan opformeres med stor succesrate). Dette viste, at man var kommet til at navngive hanner og hunner som forskellige arter, fordi størrelsesforskellen på kønnene var enorm og usædvanlig (oftest er hanner størst).
De største hunner var omkring 280% tungere end hannen og 150% højere end hannen. Dette er uden fortilfælde blandt fugle og terrestriske pattedyr. Desuden var der enorme forskelle på størrelsen afhængig af levestedet. Vægten af hunlige Dinornis fra sydog nordøen varierede fra 76 til 242 kg. På steder med tæt vegetation og stor nedbør blev fuglene mindre (ref.9774). Sidst i 2011 publicerede Eske Willerslevs afdeling for første gang et sæt mikrosatellit-markører, der var udviklet udelukkende for en uddød gruppe af organismer (ref.9531).
I denne undersøgelse publiceredes for første gang et sæt mikrosatellit-markører, der var udviklet specielt og udelukkende for at bruges til at studere en uddød gruppe af organismer (ref.9531). Eske Willerslev indgik i det internationale team, som anført af Richard Holdaway, Mike Bunce og Morten Allentoft udførte et genetisk studie af de uddøde moakæmpefugle. (Morten Allentoft var dengang PhD-studerende i New Zealand, og er nu PostDoc i Eskes Willerslevs gruppe). Men det er lykkedes at lave primere for mikrosatellitmarkører (Se: "Hvad er mikrosatelliter?"), rettet mod studier af de uddøde moa-fugle (Aves: Dinornithiformes). Man kan dermed gøre sig fri af afhængigheden af mitokondrie- DNA ved studier af gammelt DNA. Denne uddøde fuglegruppe har dermed fået en metode, som svarer til SNiP-teknologien (enkelt nukleotid polymorfi (SNP; s="single") ).
DNA-markørerne fandt man i moaknogler og moa-æggeskaller. 6 polymorfe DNA-områder (loci) anvendtes til at vurdere den genetiske diversitet (forskellige fra individ til individ). Langt flere end de 6 udvalgte markører blev forsøgt anvendt, men blev opgivet, f.eks. hvis de ikke var specifikke nok. Mikrosatellit-datasættet repræsenterede fossiler, der var 600 - 5000 år gamle. Eftersom mikrosatellitter har en høj mutationshastighed og nedarves fra begge forældre, kan de give mere detaljerede populationsgenetiske oplysninger end mitokondrie-DNA (mtDNA), der kun arves af moderen. Proceduren for fastlæggelse af mikrosatellitter er veletableret, så udfordringen er at identificere tilstrækkeligt mange loci, som er polymorfe og informative. Ofte er datakvaliteten dog problematisk.
Mikrosatellit-DNA er cellekerne-DNA, og kerne-DNA finder man meget sjældnere end mitokondrie- DNA, og det kræver omhyggelige indsamlingsmetoder.
Man har fundet knogler, ekskrementer, fjer og æggeskaller af moa-fugle. Analyser af gammelt DNA fra hundredvis af moa-individer, fundet nær hinanden såvel geografisk som tidsmæssigt, kan afdække populationsdynamiske forhold næsten svarende til forskning på nulevende populationer.
Moa-studiet krævede 4 succesfulde PCR-resultater (og det kunne være nødvendigt at udføre en PCR-analyse 6-10 gange for at opnå dette). Hvis man - som man ellers normalt gør - kun havde testet markøren i én PCR-analyse, ville man have troet, at 53% af de heterozygote gener var homozygote (idet man så kun havde fundet én allel-type i disse tilfælde). Man konstaterede allel-tab i halvdelen af 900 PCR-analyser. Allel-tabet varierede fra 36% til 70% fra markør til markør. Årsagen er det lave antal skabeloner (templates), som er typisk for gammelt DNA. I nogle tilfælde gik især de længere alleler tabt. De forskellige loci kunne have 5 - 17 forskellige alleler.
Allel-tabet kunne konstateres, hvis begge alleler blev fundet, når man lavede mange analyser: Et heterozygot individ, der i analyserne var AA, BB, BB og AB i 4 forskellige PCR-analyser, havde 75% allel-tab (da kun hver 4. analyse (25%) fremviste begge alleler, altså AB). Et heterozygot individ, der var AB og igen AB i to PCR-analyser havde intet allel-tab (0% allel-tab). Man screenede DNA fra 88 fund af sydøens kæmpemoa (Dinornis robustus). Der blev udarbejdet retningslinier for at undgå fejl (i form af færre heterozygote individer i forhold til det forventede ifølge Hardy-Weinberg lovens forholdstal for populationsgenetik). De seks anvendte markørers sandsynlige position på kromosomerne af moafuglen forsøgte man at vurdere ved at sammenligne med de to total-kortlagte fugle-genomer: Hønse-genomet (Gallus gallus) og zebrafinke-genomet (Taeniopygia guttata). Disse genomer kan ses på Internettet (www.blast.ncbi. nlm.nih.gov/Blast.cgi).
Høns og zebrafinker er evolutionsmæssigt adskilt fra moa-fuglene med over 100 mill. år. Det forklarer, hvorfor sammenligningen ikke gav noget resultat. Et enkelt sekvensområde havde dog over 80% identitet med en sekvens på 83 basepar på kromosom- 1 hos zebrafinken samt 96% identitet med en sekvens på 26 basepar på kromosom- 1 hos høns: Formentlig fand- )tes denne markør derfor på kromosom 1 hos moa-fugle. Når man engang bliver færdig med at opbygge genombiblioteket for ratite-fugle (ikke-flyvende fugle som strudse, kiwier mv.), bliver det lettere at lave sammenligninger. Genetikere har studeret museumseksemplarer af udstoppede dyr mv. for at se, om der er sket et tab af genetisk biodiversitet på følge af de seneste årtiers miljøforandringer. Sådanne undersøgelser vil altså kunne udføres på langt ældre materiale. For at finde de 6 brugbare mikrosatellit- primere måtte man analysere over 600.000 sekvenser, hvorved man fandt 1800 STR-sekvenser og endte med at teste 89 primere. Kun 6 var egnede.
De fleste æggeskaller har bevaret DNA-materialet bedre end knoglerne. Æggeskallerne har været bedre beskyttet mod bakterier. En æggeskal af Anomalopteryx leverede 4 af de 6 mikrosatellit- primere. De 6 primere, som blev brugt til DNA-studiet af kæmpemoa Dinornis robustus, stammede fra tre andre moa-slægter. De 6 primere kan derfor også bruges til analyser af andre moa-slægter, og kunne anvendes på moa-slægterne Emeus og Pachyornis.
Isoleringen af moa-DNA'et skete på følgende måde: Analyserne startede med, at man nedbrød proteiner (med enzymet “proteinase K”) i prøver med 200 mg knoglepulver. Derefter udtrak man DNA (ved en ultra-filtrering) og fjernede PCR-hæmmere (ved en siliciumoxid-rensningsmetode). Portioner af 1/1000 milliliter DNA blev opformeret ved 50 cyklusser af PCR-metoden. Afhængigt af den anvendte primer brugtes forskellige temperaturer for det trin, hvor DNA-strengene samles (annealing). Analyserne udførtes på 88 individer af moafugle: Man brugte 88 venstre-tibiotarsi-knogler (en sammenvokset skinneben/fod-knogle hos fugle).
For at undgå kryds-forurening (ved at komme til at flytte DNA fra et individ over til prøven fra et andet individ) lod man være med at bruge den ellers almindelige teknik med multi-pipettering under opsætning af PCR-analyserne. Hvert rør med DNA blev åbnet for sig og pipetteret enkeltvis over i hvert PCRrør. Man benyttede ikke de velkendte plader med 96-brønde, som giver øget risiko for kryds-forurening. Derved undgik man kryds-forurening helt.
Eftersom mikrosatellitter har en høj mutationshastighed og nedarves fra begge forældre, kan de give mere de-taljerede populationsgenetiske oplys-ninger end mitokondrie-DNA (mtDNA), som kun nedarves fra moderen.
Proceduren for fastlæggelse af mikro-satellitter fra gammelt DNA er vel-etableret, så hvis datakvaliteten er ok, er udfordringen at identificere tilstrækkeligt mange loci, som er polymorfe og informative, hvilket kun et spørgsmål om at have tid og ressourcer nok til at identificere, designe og gennemgå tilstrækkeligt mange primere.
Men ofte er netop datakvaliteten problematisk.
Mikrosatellit-DNA er cellekerne-DNA, og det finder man derfor meget sjældnere end mitokondrie-DNA. For at kunne bruge mikrosatellitter som markører, kræves derfor en langt mere omhyggelige metoder for at sikre data-pålideligheden.
Diploide organismer (med en far og en mor) har ofte arvet to forskellige alleler af et gen. Den største bekymring ved DNA-opformering er derfor “allel-tab” (“allelic dropout”).
Hvis DNA kun findes i lavt kopiantal, vil den tilfældige fordeling af DNA i de første op-formeringscyklusser i PCR-reaktionen let kunne forårsage, at en af de to alleler “oversvømmer” PCR-reaktionen, og helt undertrykker opformeringen af den anden allel. Resultatet vil være en fejlslagen PCR-reaktion, hvor den genetiske profil ligner en homozygot, uanset den oprindelige genotype. Allel-tab er velkendt i PCR-reaktioner på DNA fra bl.a. fæces, hår, fjer og tand.
Omfanget af allel-tabet kan variere betydeligt fra prøve til prøve, samt mellem forskellige primere og mellem alleler for samme mikrosatellit-markør, så det er nødvendigt at fastslå problemets omfang fra sag til sag.
Vigtigst i denne sammenhæng er det at bestemme, hvor mange selv-stændige PCR-opformeringer, det er nødvendigt at lave, for at genotypen af det tilsyneladende homozygote individ kan vurderes med sikkerhed. Dette er kendt som ‘de mange rørs metode (“multiple tubes approach”).
Moa-studiet krævede 4 succesfulde PCR-resultater (og det kunne være nødvendigt at udføre en PCR-analyse 6-10 gange for at opnå dette). Hvis man - som man ellers normalt gør - kun havde testet markøren i én PCR-analyse, ville man fejlagtigt have troet, at 53% af de heterozygote gener var homozygote (idet man så kun havde fundet én allel-type i disse tilfælde).
Man konstaterede allel-tab i halvdelen af 900 PCR-analyser. Allel-tabet varierede fra 36% til 70% fra markør til markør. I nogle tilfælde gik især længere alleler tabt. Ved test af 75 individer ville man i 5 individer finde en (falsk) homozygot for en af de 6 loci. De nævnte 6 loci var i øvrigt polymorfe: Hver med 5 - 17 forskellige alleler.
Man undersøgte for de 6 markører i 1500 PCR-reaktioner. Allel-tabet kunne konstateres, hvis begge alleler blev fundet, når man lavede mange analyser: Et heterozygot individ, der i analyserne var AA, BB, BB og AB i 4 forskellige PCR-analyser, havde 75% allel-tab (da kun hver 4. analyse (25%) fremviste begge alleler, altså AB).
Et heterozygot individ, der var AB og igen AB i to PCR-analyser havde intet allel-tab (0% allel-tab).
Man screenede DNA fra 88 fund af sydøens kæmpe-moa (Dinornis robustus) fra fossilsteder i North Canterbury for at karakterisere allel-tabet for hver markør. Der blev udarbejdet retningslinier for at undgå fejl (i form af færre heterozygote individer i forhold til det forventede ifølge Hardy-Weinberg lovens forholdstal for populationsgenetik).
Langt flere end de 6 markører blev forsøgt anvendt, men opgivet (f.eks. hvis de ikke var specifikke nok). De seks anvendte markørers sandsynlige position på kromosomerne af moa-fuglen forsøgte man at vurdere ved at sammenligne med de to total-kort-lagte fuglegenomer: Hønse-genomet (Gallus gallus) og zebrafinke-genomet (Taeniopygia guttata). Disse genomer kan ses på Internettet (www.blast.ncbi.nlm.nih.gov/Blast.cgi).
Disse fugle er dog evolutionsmæssigt adskilt med over 100 mill. år fra moa-fuglene, hvilket forklarer, hvorfor sammenligningen ikke gav noget resultat - bortset fra, at et sekvensområde havde over 80% identitet med en sekvens på 83 basepar på kromosom-1 hos zebrafinken og 96% identitet med en sekvens på 26 basepar på kromosom-1 hos høns: Formentlig fandtes markøren på kromosom 1 hos moa.
Når man er færdig med at opbygge genombiblioteket for ratite-fugle (ikke-flyvende fugle som strudse, kiwier mv.), bliver det lettere at lave sammenligninger med fund hos de beslægtede, uddøde moa-fugle.
De 6 mikrosatellit-primere, som blev brugt til DNA-studiet af kæmpe-moa Dinornis robustus, var primere, som stammede fra tre andre moa-slægter. De 6 primere kan derfor formentlig bruges til analyser af andre moa-slægter også. (Det blev bekræftet, da man anvendte primerne på moa-slægterne: Euryapteryx, Emeus og Pachyornis).
Analyserne startede med, at man nedbrød proteiner (med enzymet “proteinase K”) i prøver med 200 mg knoglepulver. Derefter udtrak man DNA (ved ultra-filtrering) og fjernede PCR-hæmmere (ved siliciumoxid-rensningsmetode).
Portioner af kun 1/1000 milliliter DNA blev opformeret ved 50 cyklusser af PCR-metoden. Afhængigt af den anvendte primer brugtes forskellige temperaturer for det trin, hvor DNA-strengene samles (annealing).
Analyserne udførtes på 88 individer af moa-fugle: Man brugte 88 venstre-tibiotarsi-knogler (en sammenvokset skinneben/fod-knogle hos fugle).
Genetikere har studeret museums-eksemplarer af udstoppede dyr mv. for at se, om der er sket et tab af genetisk biodiversitet på følge af de seneste årtiers miljøforandringer. Dette studie viser, at undersøgelser også vil kunne udføres på langt ældre materiale.
Det kræver dog et omfattende forarbejde. For at finde de 6 brugbare mi-krosatellit-primere måtte man analysere over 600.000 sekvenser, hvorved man fandt 1800 STR-sekvenser og endte med at teste 89 primere, men kun 6 viste sig egnede.
De fleste æggeskaller har bedre bevaret DNA-materiale end knoglerne. Det skyldes måske, at æggeskallerne har været bedre beskyttet mod bakterier. En æggeskal af Anomalopteryx leverede 4 af de 6 mikrosatellit-primere.
Hos nulevende dyr forekommer gentagelser i kerne-DNA'et med forskellig hyppighed i sekvenserne - ifølge nogle undersøgelser således i 1,3%, 1,8%, 2,4% og 11,3%. Hos moa-fuglen så man gennemsnitligt kun gentagelser hos 0,3% af sekvenserne. Det kan skyldes, at gentagelser var sjældne i moa-fuglens DNA (noget tilsvarende er foreslået for andre fugle), men det kan også skyldes, at DNA’et kun indeholdt lidt cellekerne-DNA, eller at der var relativt meget bakterie-DNA. Det kan også være, at tallet 0,3% ikke er det korrekte tal.
For at undgå kryds-forurening (ved at komme til at flytte DNA fra et individ over til prøven fra et andet individ) lod man være med at bruge den ellers almindelige teknik med multi-pipettering under opsætning af PCR-analyserne. Hvert rør med DNA blev åbnet for sig, og pipetteret enkeltvis over i hvert PCR-rør. Man benyttede ikke de velkendte plader med 96-brønde, som giver øget risiko for kryds-forurening. Derved undgik man kryds-forurening.
Test dig selv
Du kan tegne abonnement på BioNyt: Videnskabens verden her!
/DNA/Genom/Moa/Det korte svar er: Utvivlsomt
Maorierne vil gerne undgå skyld ved at påstå, at moa'erne var på retur, men moa-populationen var talrig og livskraftig (meget lille genetisk drift, ifølge DNA-studier, tyder på kæmpestor population). De blev stegt i jordgryder med hovedet af den døde moa over jorden, og retten var færdig, når der kom røg ud af næseborene.
Moa betyder på polynesisk »spiselig fugl«. Indtil omkring år 1500 levede 9 moaarter på New Zealand. Indtil omkring år 1000 havde New Zealand ikke kendt til pattedyr. På et tidspunkt før år 1300 nåede maorier fra Polynesien frem til New Zealand. Menneskene, og pattedyr, der blev bragt med, udryddede alle moa-arter. De nulevende kiwier er dog beslægtede. Moa-skeletter findes over hele New Zealand, bl.a. i sumpe. I huler på sydøen er uheldige fugle faldet gennem huller eller svage punkter i hulernes loft. De største moa-arter blev op til 3 meter høje og vejede omkring 250 kilo. Den beslægtede elefantfugl fra Madagaskar er lidt tungere, men blev ikke helt så høj. Moa'erne var strudsefugle og kunne ikke flyve.
Stamformen gav ophav til "ratites"- arter på de forskellige kontinenter:
New Zealand: Kiwier, Moa'er (uddøde).
Sydamerika: Nanduer, Tinamuer.
Australien: Emu'er, Kasuar'er
New Guinea: Kasuar'er
Madagaskar: Elefantfugle (uddøde)
Afrika: Struds (ref.9773)
Test dig selv
Du kan tegne abonnement på BioNyt: Videnskabens verden her!
/DNA/Genom/Moa/Det korte svar er: De uddøde moa-fugle
Moa-fuglene i New Zealand mangler vinger (de er nu uddøde). I rarites-gruppen er tab af flyveevne sket flere gange. I modsætning til andre "rarites"-fugle havde moa'erne helt mistet vingerne. Hunnerne var meget større end hannerne. De havde forsinket modning (K-strategi), hvilket gjorde dem ekstremt følsomme, da mennesker begyndte at jage dem og afbrænde skovene. Kiwi'erne på New Zealand er udviklet fra en anden kolonisering med ratitefugle end den, der gav ophav til moa'erne (ref.). Moa'erne udvikledes fra for 18 mill. år siden, især for 4-10 mill. år siden. De nærmeste slægtninge til de uddøde moa-kæmpefugle er nogle fugle i Sydamerika, som er relativt små, som kan flyve, og som man slet ikke opfattede som rarites-fugle! (ref.9773).
Test dig selv
Du kan tegne abonnement på BioNyt: Videnskabens verden her!
/DNA/Genom/Hvaler/Det korte svar er: At der er adfærdstyper, og måske forskellige arter
Man har ofte anset fysisk isolation for at være en hovedårsag til opdeling imellem populationer. Forskning viser imidlertid, at adskilte populationer kan opstå, selv om der (som i havet) ikke er fysiske barrierer. Populationerne kan f.eks. opstå ved specialisering i fødevalget, klimatiske faktorer, varierede havområder og trofasthed overfor bestemte yngleområder.
Spækhuggerne i det nordlige Atlanterhav kan opdeles i tre adskilte populationer. Dette blev påvist af en forskergruppe, hvori Eske Willerslev deltog. Man studerede dels 17 mikrosatellit loci, dels hyppigheden af mitokondriehaplotyper og for nogle af prøverne sammenlignede man fuldstændige sekvenser af mitokondrie-genomet. En af spækhugger-populationerne er knyttet til sildebestanden i Nordatlanten, Island og Norge. En anden spækhugger-population er knyttet til makrelbestanden i det nordøstlige Atlanterhav. En tredie spækhugger- population er delvis knyttet til tun i Atlanterhavet (ref.9549). Faktisk er der måske allerede tale om flere forskellige arter af spækhuggere, selv om de umiddelbart ser ens ud. Andre studier kunne påvise forskellig morfologi hos 4 typer omkring Antarktis. De har forskellig adfærd: De spækhuggere, der lever af sæler, svømmer stille i små grupper og dykker langt for at overraske sælerne. De spækhuggere, der lever af fisk, er mere sociale og afgiver flere lyde, for fisk hører dårligt ved høje frekvenser. Ud for England er der mindst to økotyper, men om de parrer sig med hinanden er endnu uvist (da man kun har studeret mitokondrie- DNA, dvs. moderlinierne). For at teste om DNA udveksles på tværs af moderlinierne må fader-DNA også testes. Ud for Skotland fandt man spækhuggere, som lever af småhvaler. Tom Gilberts studier af spækhuggerskeletter i museer har vist, at nogle havde meget nedslidte tænder, måske slid efter indtagelse af store mængder små fisk, og disse dyr viste stor variation i isotoperne, hvilket tyder på at kosten var varieret. Andre (større individer) havde ikke nedslidte tænder, måske fordi de spiste større pattedyr, og disse dyr viste ingen variation i isotoperne, hvilket tyder på at kosten var ensartet. DNA-sekventering af ca. 100 år gamle museums-hvalskeletter har vist, at de store spækhuggerarter adskilte sig genetisk fra de mindre spækhuggerarter. DNA-studiet viste, at nordatlantens Vågehval-ædende spækhuggere er beslægtet med Vågehval- ædende spækhuggere omkring Antarktis. Studierne giver indsigt i de økologiske processer, der danner grundlag for udvikling af nye arter i evolutionen (ref.9587).
Test dig selv
Du kan tegne abonnement på BioNyt: Videnskabens verden her!
/DNA/Gen/Operon/Det korte svar er: Et gen er et stykke DNA, som koder for et protein eller et aktivt RNA
Et gen er et stykke DNA, som koder for et protein eller et aktivt RNA
Test dig selv
Du kan tegne abonnement på BioNyt: Videnskabens verden her!
/DNA/Gen/Operon/Det korte svar er: DNA-molekylet områder, hvor beslægtede gener er samlet, og typisk styres samlet
Ofte findes der på DNA-molekylet områder, hvor beslægtede gener er samlet, og ofte sådan, at de sidder lige efter hinanden på kromosomet. Disse gener styres af en fælles reguleringsmekanisme. Lige inden genet, sidder der en såkaldt operator, som er det stykke DNA, hvortil en eventuel repressor skal binde (se næste spørgsmål). Lige før (dvs. opstrøms for) operatoren findes en såkaldt promotor, som er det sted på DNA, hvor RNA-polymerasen binder lige før transkriptionen af det efterfølgende gen går i gang. En ”operon” er alle de tre ting tilsammen, dvs. en promotor, en operator og et (eller flere beslægtede) gen(er).
Test dig selv
Du kan tegne abonnement på BioNyt: Videnskabens verden her!
/DNA/Gen/Operon/Det korte svar er: Regulatorisk gen, hvis genprodukt binder sig (hvis den er aktiveret) til operatoren
Opstrøms for operonet findes et såkaldt regulatorisk gen. Produktet af dette gen er et protein, som kaldes en repressor. Denne repressor binder sig (hvis den er aktiveret) til operatoren. Man kan så spørge, hvad det er, som aktiverer/deaktiverer repressoren? Svaret herpå er: De gener, som findes i operatoren, har en bestemt funktion, f.eks. at de kan kode for de enzymer, som nedbryder lactose. I dette tilfælde vil tilstedeværelsen af lactose være med til at inaktivere repressoren. Dvs. at transkriptionen af de gener, som koder for enzymer, som nedbryder lactose, bliver reguleret af lactoseproduktet selv.
Test dig selv
Du kan tegne abonnement på BioNyt: Videnskabens verden her!
/DNA/Gen/Operon/Det korte svar er: En (aktiverende) del af de såkaldte transkriptionsfaktorer
Aktivator-proteiner er en del af de såkaldte transkriptionsfaktorer. Disse aktivatorproteiner binder til en enhancer, som laver en loop-formation til det pågældende promotor-område, som skal transkriberes. Dermed er aktivatoren også med til at regulere gen-ekspressionen.
Test dig selv
Du kan tegne abonnement på BioNyt: Videnskabens verden her!
/DNA/Gen/Operon/Det korte svar er: En (hæmmende) del af de såkaldte transkriptionsfaktorer
Repressor-proteiner kan binde til DNA-sekvenser, som kaldes silencer, og disse er med til at inhibere, altså hæmme, transkriptionen.
Test dig selv
Du kan tegne abonnement på BioNyt: Videnskabens verden her!
/DNA/Gentek/Det korte svar er: DNA, som er blevet overført fra en art til en anden art
Rekombinant DNA kaldes også for hybrid-DNA. Det er DNA, som er blevet overført fra en art til en anden art. Denne overførsel kan ske ved f.eks. gensplejsning.
Test dig selv
Du kan tegne abonnement på BioNyt: Videnskabens verden her!
/DNA/Gentek/Det korte svar er: En vektor er f.eks. et plasmid, som man kan indsætte nye gener i
Inden for bioteknologi omtaler man "vektorer" i forbindelse med gensplejsning. En vektor er f.eks. et plasmid, som man kan indsætte nye gener i. Plasmidet bliver kløvet med et restriktionsenzym, hvorefter man indsætter et nyt gen. Dette nye gen ”klistres” på plads med et ligase-enzym. Disse gener er oftest påsat et markør gen så det er muligt at frasortere de plasmider, som ikke har fået genet inkorporeret.
Test dig selv
Du kan tegne abonnement på BioNyt: Videnskabens verden her!
/DNA/Gentek/Det korte svar er: En metode til at skelne mellem organismer ved at analysere kløvede DNA fragmenter fra dem
Restriction Fragment Length Polymorphisms (RFLP) er en metode til at differentiere organismer ved analyse af kløvede DNA fragmenter fra dem. Hvis to organismer har forskellige afstande mellem de valgte kløvningssteder, (når man bruger en bestemt restriktions-endonuklease), vil de frakløvede fragmenter have forskellige længder. En sammenligning af disse fragmenter vil kunne bruges til at adskille to arter, og til at adskille de forskellige stammer af en art.
Test dig selv
Du kan tegne abonnement på BioNyt: Videnskabens verden her!
/DNA/Gentek/Det korte svar er: Metoder, hvormed man kan ændre på gener
Genteknologi er i sig selv mange ting, og kan defineres på mange forskellige måder. I bund og grund handler det om at lave om på gener ved hjælp af forskellige teknikker, som forskellige mikroorganismer har gjort naturligt brug af.
F.eks. har bakterier egne enzymer, som kan klippe i bestemte DNA-sekvenser, uden at bakterien ødelægger sit eget DNA. Et eksempel på et enzym, som klipper i DNA, er Eco RI (stammer fra Escherichia coli bakterien). Genkendelsesstedet for enzymet ses på figuren. Kløvningen sker ved de røde pile, mens den grønne prik angiver symmetriaksen.
 .
.
Bemærk at sekvensen er palindromisk (hvilket betyder, at det kan staves ens forfra og bagfra, som f.eks. ordene ”radar” og ”regninger”). Man kan spørge: Hvordan undgår bakterien at klippe i sit eget DNA? Sandsynligheden for at sekvensen (3´CTTAAG 5`) findes i genomet er: ¼ x ¼ x ¼ x ¼ x ¼ x ¼ = (¼)6 = 0,000244 = 1/4096 dvs. at for hver ca. 4000 basepar vil man kunne finde denne sekvens. Da en bakteries genom er større end det, skal den finde andre muligheder at beskytte sig med.
Bakterier kan lave en såkaldt methylering af deres DNA. Enzymet, som står for det, kaldes DNA-methyltransferase. Denne methylering medfører, at restriktionsenzymerne ikke kan genkende en bestemt sekvens. Methyleringen har også visse bivirkninger. (Methyleret DNA kan replikeres til to hemimethylerende DNA-strenge, hvorefter methylasen påsætter de manglende methylgrupper, se her).
Andre former for genteknologi er Rekombinant genteknologi, som f.eks. bruges i medicinalindustrien af Novo Nordisk A/S til fremstilling af peptidhormonet insulin. Her anvender man den mulighed, at man kender DNA-sekvensen for insulin, og at man derfor kan indsætte disse gener i gærceller, hvormed man kan få dem til at producere insulin hurtigt og billigt.
Test dig selv
Du kan tegne abonnement på BioNyt: Videnskabens verden her!
/DNA/Gentek/Det korte svar er: Sammensætning af gener på nye måder ved hjælp af genteknologi
Når man kender til restriktionsenzymer, og kender forskellige gener og deres egenskaber, vil man også være i stand til at klippe et gen ud og sætte det ind i en anden organisme, og evt. derved give den nye organisme en ny egenskab. Lovene om industriel anvendelse af gensplejsning er skrappe, og der er mange etiske hensyn som skal tages til efterretning, før man må begynde at gensplejse.
Det er dog ikke muligt at lave mennesker med f.eks. blade fra planter, så vi slipper for at spise sukker. Visse fysiologiske begrænsninger vil altid være i vejen for sådanne ”sjove” opfindelser.
Test dig selv
Du kan tegne abonnement på BioNyt: Videnskabens verden her!
/DNA/Gentek/Det korte svar er: Gen-knockout er en bioteknologisk metode, hvormed man kan inaktivere et eller flere gener i et kromosom
Gen-knockout er en bioteknologisk metode, hvormed man kan inaktivere et eller flere gener i et kromosom. Formålet er at undersøge et gen, som man har sekvensbestemt, men som man også ønsker at kende funktionen af.
Læs mere her
Test dig selv
Du kan tegne abonnement på BioNyt: Videnskabens verden her!
/DNA/Gentek/Bakterier/Det korte svar er: Bakterier kan udveksle gener på flere måder, kaldet transformation, transduktion og konjugation
Bakterier har den enestående mulighed, at de kan udveksle gener. De kan udveksle flere gener eller hele plasmider (dvs. selvformerende kromosomstykker). Denne udveksling af gener fremskynder bakteriernes evolutionsprocesser, fordi generne kan give apatogene (ikke-sygdomsfremkaldende) bakterier nye egenskaber, som kan gøre dem patogene. Udvekslingen af gener kan ske på tre forskellige måder:
1) Transformation: Når der i en bakteriekoloni dør bakterier vil de døde bakteriers DNA flyde rundt i den væske, som bakterierne befinder sig i. Dette DNA kan optages af andre bakterier, som derefter kan gøre brug af det nye DNA.
 Kilde her
Kilde her
2) Transduktion: Når en bakterie bliver inficeret af et virus, kan denne virus medbringe DNA-fragmenter fra en tidligere værtscelle (transduktionens donor-bakterie). Dette DNA bliver måske (afhængig af hvilken type virus det er) inkorporeret i den nye bakteries DNA, og kan dermed give modtager-bakterien nye egenskaber.
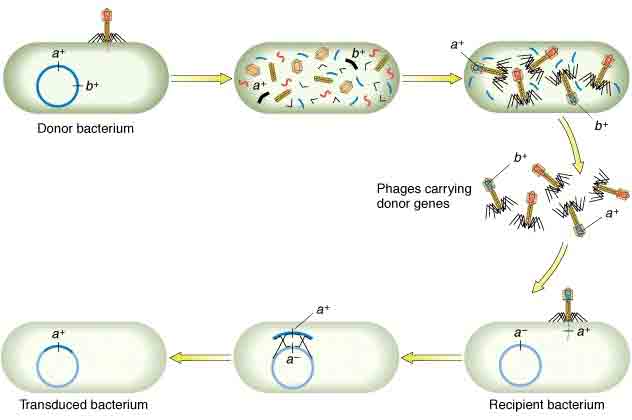 Kilde her
Kilde her
3) Konjugation: Her danner to bakterier bro mellem hinanden. Gennem denne bro kan to bakterier udveksle DNA. Den ene bakterie (kaldet +) har et plasmid, som låner en enkel-strenget kopi til den modtagende bakterie (kaldet -). - bakterien laver så en tilsvarende plasmid til sig selv ud fra den lånte kopi. Nu har begge bakterier et plasmid hver. 
width="400">Kilde her
Se flere billeder her
Test dig selv
Du kan tegne abonnement på BioNyt: Videnskabens verden her!
/DNA/Gentek/Dyr/Det korte svar er: To forskere mødtes i 1984 og lavede de en brainstorm om, hvordan man kunne fremstille menneskeproteiner til at bekæmpe blodpropper. De nåede frem til, at det måtte være muligt at indføre menneskegener i DNA'et hos dyr, og senere høste proteinet fra dyrets mælk
Historien om geden og musen begyndte i 1984 i Biogen Inc. i Cambridge, Massachusetts, hvor Harry M. Meade og Nils Lonberg mødtes for 20 år siden, da de var henholdsvis 39 og 29 år.
Harry M. Meade var vokset op som barn på en gård med malkekvæg nær Pittsburgh og var vant til landmandslivet og til at malke kvæg. Han fik et liv med geder. Nils Lonberg fik et liv med mus. Han voksede op i Arlington i Virginia, hvor han fik to ørkenrotter fra sin bedstemor, og siden avlede sig frem til en koloni på 56 dyr. Senere mødte han sin kommende kone i muselaboratoriet ved Memorial Sloan-Kettering Cancer Center i New York.
Da de to forskere mødtes i Biogen i 1984, lavede de en brainstorm om, hvordan man kunne fremstille menneskeproteiner til at bekæmpe blodpropper. De nåede frem til, at det måtte være muligt at indføre menneskegener i DNA'et hos dyr, og senere høste proteinet fra dyrets mælk.
De syntes selv, at dette var en fantastisk idé, men kun få af deres forskerkolleger var enige. Da de dagen efter fortalte ideen til Biogen's ledelse, bl.a. direktøren Walter Gilbert, blev de to forskere nærmest leet ud af lokalet. Nils Lonberg husker, at Walter Gilbert så på ham, og sagde "hvorfor ikke gøre det i gåseæg - en gås, som lægger guldæg? Det er netop, hvad man nu gør, som æghistorien i dette blad viser.
Nils Lonberg fik efter en uddannelse i biokemi og molekylærbiologi ved Harvard Univ. stilling i Biogen indtil 1985. Harry M. Meade besøgte ham ofte og hjalp med at ændre musenes DNA, så de fik menneskeproteiner i deres mælk.
De to forskere lejede en kvinde-brystpumpe fra et apotek i New York og justerede apparatets brystvortepipetter, så det passede til hunmusenes små brystvorter.
Da Harry M. Meade et år senere ville aflevere den lånte brystpumpe tilbage, blev apotekeren så chokeret over at høre, hvad den havde været brugt til, at han nægtede at tage den tilbage.
Harry M. Meade og Nils Lonberg supplerede hinanden og udarbejdede, hvad der blev til det første patent (i 1989) på at udvinde lægemidler fra mælk. Samme år deltog Nils Lonberg i en konference, hvor en forsker fortalte om en ny knock-out teknik, hvormed man kunne inaktivere udvalgte gener i mus. Dette udnyttede Nils Lonberg til at frembringe mus, som er bærere af menneskets komplekse immunsystem, og som kan producere antistoffer i sine celler. Nils Lonberg blev videnskabelig leder af Medarex Inc., som er et førende firma inden for kommerciel anvendelse af gensplejsede mus.
Et halvt hundrede biotekfirmaer og medicinalfirmaer anvender mus fra Medarex for at finde behandlinger for sygdomme, fra ondartet hudkræft til lymfekræft.
Når Nils Lonberg holder foredrag, viser han ofte to røntgenbilleder af en patient før og efter behandling med antistoffer fra hans mus. Patientens 31 lungetumorer var forsvundet på det andet røntgenbillede.
Harry M. Meade blev en af de ledende forskere, som skabte de gensplejsede geder hos GTC, og han kan fortælle om mennesker, der vil kunne redde livet med antitrombin, som hidtil kun har kunnet skaffes fra bloddonorblod, men som nu altså er godkendt i Europa, produceret i gedemælk.
Endnu har Nils Lonberg og Harry M. Meade ikke lavet et lægemiddel sammen, men de håber, at det vil lykkes engang, - som for at besegle det gode samarbejde gennem årene. Ideen er først at udvikle et lægemiddel i Nils Lonberg's mus, og derefter masseproducere det i Harry M. Meade's Saanen-geder, Alpin-geder eller Toggenburg-geder.
Det kunne være en passende afslutning på 20 års forskning i gensplejsede dyr til bekæmpelse af sygdomme hos mennesker (4974).
Test dig selv
Du kan tegne abonnement på BioNyt: Videnskabens verden her!
/DNA/Gentek/Dyr/Monoklonale_antistoffer/Det korte svar er: Man antager, at ca. 200 firmaer verden over arbejder med udvikling af ca. 500 produkter af monoklonale antistoffer
Monoklonale antistoffer er antistoffer fra samme klon af en celle ("monoklonalt"). De har været anvendt i 30 år, og man antager, at ca. 200 firmaer verden over arbejder med udvikling af ca. 500 produkter af monoklonale antistoffer (4978). Der er udviklet og godkendt talrige monoklonale antistoffer til diagnose, altså påvisning af sygdom, og i USA er mindst 18 monoklonale antistoffer blevet markedsført til sygdomsbehandling. Flere har været store kommercielle successer. F.eks. Mabthera (rituximab, der i USA markedsføres under navnet Rituxan). Det er det mest solgte af alle lægemidler mod kræft, nemlig mod den lymfekræft, som kaldes "ikke-Hodgkin's lymfom". Stoffet er desuden i 2006 blevet godkendt i USA til den trediedel af leddegigt-patienter, som ikke kan behandles med anti-TNF (også kaldt "anti-TNF" eller alfa-TumorNekroseFaktor inhibitor) (4971). (Mabthera virker selektivt ind på immunsystemets B-celler, som i leddegigt angriber knogler og ledbrusk. Antistoffet virker ikke på de celler, som danner nye B-celler, og forhindrer derfor heldigvis ikke, at immunsystemet kan virke på nye infektioner).
Det antages, at verdensmarkedsværdien for terapeutiske antistoffer vil være ca. 150 milliarder kr. i 2010, medens markedsværdien for antistoffer til diagnoser vil være under 1 milliard kr. (4978)
Typisk indvirker et lægemiddel på kontakten mellem et lille molekyle og et stort molekyle. Lægemidlet er ofte et tilsvarende lille molekyle, som indvirker på det store molekyle, som f.eks. kan være et enzym, der har betydning for stofskiftet eller virker som ion-transportører.
Opdagelsen af sådanne lægemidler er ofte sket ved at studere, hvordan celler og organismer reagerer fysiologisk, når de udsættes for forskellige små molekyler. Nu er man imidlertid blevet bedre til også at finde målsteder, som reagerer på store molekyler. Dermed kan man lede direkte efter målstederne selv, idet man kan studere, hvilken virkning en genetisk ændring har på hele organismens fysiologi.
Hvis man f.eks. bruger mus som en modelorganisme, kan man ændre dens gener og iagttage virkningen. Eller man kan studere, hvilken betydning genetiske forskelle i individuelle menneskers gener har på forekomsten af sygdomme. På den måde kan man finde frem til nye målmolekyler og lægemidler, som indvirker på disse målsteder.
Man har derved fundet ud af, at mange processer i vores krop sker ved, at store molekyler indvirker på andre store molekyler. Det kan f.eks. være en celles receptor, et hormon eller en vækstfaktor, et cytokin eller et kemokin. Sådanne samvirkninger mellem store molekyler kan ikke let stoppes af små molekyler. De store er simpelthen for stabile molekylkomplekser. Det er baggrunden for, at antistoffer er så vigtige, når der skal udvikles nye lægemidler.
Antistoffer er store molekyler. De kan derfor lettere stille sig i vejen for store molekylers samvirken med hinanden. F.eks. forhindrer lægemidlet Remicade (infliximab) fra firmaet Centocor, at alfa-TNF-protein sætter sig på dets receptor. (Lægemidlet bruges mod leddegigt, psoriasis, colitis ulcerosa, Crohn's sygdom og Morbus Bechterew) (4933).
Antistoffer er proteiner. Det er i princippet muligt at bruge andre store molekyler end proteiner, men det har vist sig vanskeligt at lave store molekyler, som ikke er proteiner. Man havde f.eks. en overgang stor interesse for kulhydrater som mulige lægemidler. Men det viste sig ekstremt vanskeligt at få kulhydrat-lægemidler til at være specifikke og effektive.
Det største problem med proteiner er, at vores immunsystem er udviklet til at opdage fremmede proteiner. Humane antistoffer er en undtagelse i denne sammenhæng, idet de er den eneste type af terapeutiske proteiner, som både kan være meget forskelligt opbyggede, men samtidig er meget lidt aktiverende på immunsystemet, og humane antistoffer er praktisk talt aldrig giftige for kroppen.
Antistoffer har yderligere to vigtige egenskaber, som gør dem til gode lægemidler. De nedbrydes kun meget langsomt i kroppen, og de er i stand til at få andre komponenter i det naturlige immunsystem til at angribe målcellerne eller til at indvirke gavnligt på cellernes signalreceptorer.
Denne forstærker-funktion hos antistofferne fremkaldes via antistofmolekylets "tunge kæde", også kaldet fc-delen af antistofmolekylet. Der findes forskellige underklasser af antistoffer med forskellige fc-strukturer. Dette gør det muligt at finde lægemiddelkandidater med meget forskellige egenskaber. F.eks. kan forskellige antistoffer virke på samme målsted, men med modsatte virkninger på cellens biologi. Det ene antistof kan f.eks. virke fremmende på en receptors aktivitet, medens det andet antistof omvendt hæmmer receptoren (4933).
Problemet ved lægemidler, som er monoklonale antistoffer, er at de er store proteiner, der er beklædt med sidegrene af kulhydrater. De kan fremstilles ved at dyrke gensplejsede pattedyrceller, men det er ofte vanskeligt, og dyrkningsmedierne er kostbare, - og desuden er oprensningen af antistofferne besværlig og kvalitetskontrollen skal være meget avanceret.
Anvendelsen af antistof-lægemidler kræver, at de indsprøjtes i en blodåre eller under huden. Det kræver et nålestik. Det er langt lettere (og langt billigere) blot at skulle tage en pille. Derfor bruges antistofbaserede lægemidler kun til alvorlige sygdomme, hvor man ikke har andre behandlinger.
Det første lægemiddel, der var baseret på et monoklonalt antistof, blev godkendt allerede i 1980'erne. Siden begyndelsen af 1980'erne havde forskere nemlig forsøgt at producere menneskeproteiner i mus. Disse proteiner indeholdt typisk små dele af museprotein, og det viste sig, at det medførte bivirkninger, hvis disse antistoffer blev givet til mennesker, og udviklingen af antistof-lægemidler stoppede helt i begyndelsen af 1990'erne, fordi der opstod dette HAMA-problem: "Human anti-mus antistof"-reaktion. Musenes antistoffer gav problemer, når de blev indgivet i mennesker.
Med ny teknologi, kaldet protein-manipulation eller protein-engineering, lærte man at nedsætte HAMA-problemet, så immunsystemet hos mennesker reagerede mindre på museantistofferne, idet man erstattede noget af musesekvenserne med menneske-sekvenser.
Nils Lonberg blev ansat som seniorforsker ved GenPharm i 1990. I GenPharm udviklede han to forskellige slags gensplejsede mus. Den ene havde et immunsystem, der ikke kunne producere antistoffer. Den anden mus indeholdt generne, som er ansvarlige for at lave menneskets antistoffer (4931).
Afkommet mellem disse to mus var mere menneskelignende end muselignende med hensyn til deres immunsystem. Hvis en sådan mus fik en menneskesygdom, f.eks. ved at blive påført nogle svulstceller fra et menneske, producerede musecellerne antistoffer, som ville kunne bruges i et menneske. Når disse antistoffer indsprøjtes i mennesker, reagerer menneskets immunsystem, som om det var kroppen selv, som havde dannet disse antistoffer. Antistofferne bliver altså ikke mere opfattet som fremmede musestoffer.
Det er ved disse teknikker ikke nødvendigt at tilpasse antistofferne til hvert enkelt individuelle menneske. I stedet kan man anvende en transgen mus, som selv laver menneske-antistofmolekyler, der uden problemer kan gives til alle mennesker.
Man kan sikre sig, at antistofferne virker optimalt ved at udsætte musene for fremmedstoffet, indtil de laver rigtig gode antistoffer mod det.
Antistofferne kan gøres mere effektive ved at de bindes til små giftmolekyler, som gør dem i stand til direkte at ødelægge deres målsted, f.eks. en kræftcelle.
Nils Lonberg fortalte om sine mus på en konference i 1993. Det medførte imidlertid, at et andet firma, Cell Genesys, anklagede GenPharm for at have stjålet firmahemmeligheder. GenPharm svarede igen ved at hævde, at Cell Genesys havde overtrådt GenPharm's patentrettigheder. Retssagerne kostede så meget, at GenPharm måtte afskedige 103 af dets 110 ansatte. Det udfordrende firma Cell Genesys skiftede navn til Abgenix og endte med at droppe anklagerne om tyveri, og betalte GenPharm 40 mill. dollar for patentrettighederne (4931).
Et andet firma, Medarex, købte GenPharm for 62 millioner dollar i 1997, og kunne 3 år senere rejse 388 millioner dollar ved at udstede aktier. Medarex har siden udviklet 27 eksperimentelle lægemidler, som er produceret i mus med humane gener. Af disse har firmaet indledt kliniske forsøg med 18 lægemidler, hvoraf 7 nu afprøves på mennesker. Det er bl.a. stoffet MDX-010, som er en ny lægemiddeltype mod hudkræft, men som også vil kunne bruges mod andre kræftformer. Tre af firmaets kommende lægemidler er udviklet sammen med firmaet Johnson & Johnson, to afprøves af firmaet Novartis, og fire er blevet udviklet af det danske firma Genmab, som Medarex er delvis ejer af (4933).
Genmab i København indgik i 2001 en samarbejdsaftale med Medarex og dets datterselskab Genpharm International (samt med firmaet deCode Genetics i Island) om at samarbejde om forskning, udvikling og salg af menneske-antistoffer mod medicinske målsteder.
Medarex samarbejder desuden med det store firma Bristol-Myers Squibb om at udvikle et lægemiddel til behandling af metastatisk hudkræft. Firmaet Pfizer Inc. har også interesseret sig for Medarex og betalte 80 millioner dollar for rettighederne til 50 fremtidige antistoffer, som måtte blive udviklet over det næste tiår. Kæmpefirmaet Amgen udvikler tre nye lægemidler i et samarbejde med Medarex's mus, og måske vil Medarex blive opslugt af Amgen, ligesom Amgen opslugte Medarex'' konkurrerende firma Abgenix.
Abgenix har 13 eksperimentelle lægemidler, som er blevet udviklet fra transgene mus. Blandt andet har Abgenix rapporteret om en kræftbehandling, som er blevet udviklet sammen med kæmpefirmaet Amgen, og som fik tyktarmskræft i senstadie til at svækkes med 46%. Det fik Amgen til at opkøbe Abgenix for 2,2 milliarder dollar.
Der er altså nogen som mener, at der er rigtig mange penge i transgene mus, som kan lave nye antistoffer.
Figurtekst: Det monoklonale antistof Catumaxomab bruges mod visse kræftformer. Det er specielt ved at den ene arm af antistofmolekylet bindes til kræftceller, som har specielle epithelcelle-klæbemolekyler, og at den anden arm af antistofmolekylet bindes til T-immunceller, som har CD3-molekyler på overfladen (4983).
Den såkaldte "accessory"-celle (f.eks. en makrofag-celle) har betydning for T-celle-immunsvaret, nemlig dels (ved anvendelse af MHC-molekyler) at "præsentere" fremmedstoffet ("antigenet"), og dels ved at cellen har en "sensibiliserende" funktion, som medfører dannelse af specielle celletyper, som udskiller immunhormoner (4984).
Man har fremstillet langt over 25.000 monoklonale antistoffer (4980). De fleste er lavet i små portioner på under 0,1 gram til forskningsformål. Kommercielle produktioner betegnes som middelstore, hvis de er over 10 gram, og store hvis de er over 100 gram. Antistofferne bruges til diagnose, til behandling og til videre forskning.
Langt de fleste monoklonale antistoffer er dannet i oprindelige museceller. Menneskekroppen reagerer imod museantistoffer, men man har enten kunnet lave antistofferne mere menneskelignende eller produceret antistofferne i SCID-mus. ("Severe Combined Immunodeficiency", hos mennesket kendt som "bubble boy" sygdom, dvs. med alvorlige defekter i både immunforsvarets T- og B-cellesystem). Man kan også producere antistofferne "in vitro", dvs. i laboratoriet og uden brug af mus, men det er kostbart, og bruges især til større produktioner.
Antistoffer dannes af immunsystemets B-celler. Hver B-celle kan kun danne én type antistof. En B-celle fra musen bliver i laboratoriet sammensmeltet med en kræftcelle (en myeloma-celle, dvs. en B-celle, der ikke kan dø, fordi den er en kræftcelle). Ved denne cellefusion dannes en såkaldt hybridoma-celle. Denne har dels kræftcellens evne til at leve evigt (og derved danne en svulst), og dels evnen hos musens B-celle til at producere det pågældende antistof. Derved får man altså "monoklonalt antistof". Omkring 0,2-0,5 ml væske med ca. 100 hybridoma-celler sprøjtes ind i nogle mus, hvorefter cellerne vokser op til en svulst i musens bughule, og svulstcellerne afgiver store mængder af det pågældende antistofprotein til musens bugvæske. (Ophobning af bugvæske kaldes ascites). Musene kan tappes for ascitesvæske i 2-3 omgange inden de må slås ihjel, f.eks. efter 2 uger. Man forsøger at sikre sig, at hybridoma-cellerne ikke er meget invasive i musen, og at de har en høj udskillelse af antistoffer (4980).
Problemerne ved denne produktionsmetode er, at personalet skal være veltrænet, at musene selv danner antistoffer (dog ikke, hvis man bruger mus, som mangler evnen til at lave antistoffer). Desuden må man sikre sig, at musene ikke bliver inficeret af bakterier eller virus.
Når man kun skal bruge mindre mængder antistof, og når det skal gå hurtigt, bruger man mus-bugvæske metoden, fordi produktionstiden kan holdes nede på 1½ måned.
Produktion i ståltanke (in vitro) rummer også problemer, bl.a. risiko for virusinfektioner, dannelse af giftstoffer, rester af DNA fra døde celler og forurening med kalve-antistoffer, fordi cellerne skal have næring fra kalve-blodvæsker. (Man kan undvære blodvæsker i dyrkningsmediet, men så får antistofferne anderledes kulhydrat-sidekæder (4981)). Det er nogenlunde lige besværligt at isolere antistoffer fra musens bugvæske som efter dyrkning af cellerne i ståltanke.
Det kan tage 3-8 år at opnå godkendelse af in vitro produktion af antistoffer, selv om antistoffet tidligere er blevet produceret ved mus-bugvæske metoden.
Nogle lande har begrænset eller forbudt brugen af mus-bugvæske produktionsmetoden af hensyn til dyrevelfærden. Der er dog ikke overbevisende beviser på, at musene lider af smerte eller andet ubehag (4980). Mennesker, som har svulster i bughulen har ikke særlig meget smerte bortset fra, hvis også tarmen er angrebet, og ophobning af væske i bughulen er ikke smertefuldt, men kan dog evt. besværliggøre åndedrættet. Udtapning af bugvæske virker befriende og ikke smertefuldt. Men nogle cellelinier kan, fordi de er mere aggressive, være til tydeligt ubehag for musene, som i så fald får hurtigt åndedræt, nedsat aktivitet, taber sig eller indtager en krum holdning.
Man er gået stadig mere over til in vitro metoder for at undgå brugen af dyrene. Men medens hybridomacellerne vokser villigt i musene, er de ikke sjældent umulige at dyrke in vitro, og hvis der f.eks. kun skal produceres 10 gram antistof, vil man af økonomiske grunde hellere opgive en produktion end udvikle en kostbar in vitro produktion (4980). Artiklen her bragtes i BioNyt nr. 136 s.12-15 (marts 2007).
Test dig selv
Du kan tegne abonnement på BioNyt: Videnskabens verden her!
/DNA/Gentek/Dyr/Omega-3/Det korte svar er: Forskere i USA har klonet en gris, som kan producere omega-3 fedtsyrer
Forskere i USA har klonet en gris, som kan producere omega-3 fedtsyrer. Normalt kan mennesker kun få omega-3 ved at spise bestemte typer fisk (eller fra kosttilskud). Der er rejst tvivl om, hvorvidt fedtsyren fra sådanne grise vil være særlig fordelagtig. Men forskerne bag forsøget siger, at den nye forskning både vil komme landbruget og sundhedsbevidste forbrugere til gode. De forventer dog også stor modstand fra visse grupper, som generelt er imod genetisk modificerede fødevarer (4808).
Test dig selv
Du kan tegne abonnement på BioNyt: Videnskabens verden her!
/DNA/Gentek/Farma/Det korte svar er: Det kaldes på engelsk for ”genetisk pharming” eller blot ”pharming” og spiller derved på de engelske ord “farming” og “pharmaceuticals”. Nogle af disse produkter fremstilles i husdyr, men mange af produkterne forsøger man at producere i afgrødeplanter. Ved farmalandbrug kan produktionen både ske i husdyr og planteafgrøder
Farmalandbrug (som vi her vælger at kalde området) kaldes på engelsk for ”genetisk pharming” eller blot ”pharming”. Det spiller på de engelske ord “farming” og “pharmaceuticals”.
Ved farmalandbrug kan produktionen både ske i husdyr og planteafgrøder. Eksempler på forsøg herpå er produktion af blodproteiner (fx: humanserumalbumin), vacciner (fx: kolera, miltbrand og pest), immunstoffer (fx: interferon), hormoner (fx: insulin), enzymer (fx:trypsin), antistoffer (fx: mod kræft, sædceller, bakterier). Andre eksempler er ”tissue plasminogen activator” mod blodpropper, erythropoietin mod blodmangel, blodstørkningsfaktor VIII og IX til blødere, alfa-1-antitrypsin mod cystisk fibrose. Man laver også forsøg med produktion af proteasehæmmere (enzymer, som forhindrer proteinnedbrydning) og produktion af proteiner som fibrinogen, lactoferrin, kollagen, aprotinin, lysozym, allergener osv. osv. Nogle af disse produkter fremstilles i husdyr, men mange af produkterne forsøger man at producere i afgrødeplanter. Artiklen her bragtes i BioNyt nr. 136 s.6 (marts 2007)
Test dig selv
Du kan tegne abonnement på BioNyt: Videnskabens verden her!
/DNA/Gentek/Farma/Dyr/Det korte svar er: Gensplejsede høns vil kunne producere farmaproteiner i æggehviden og man har opnået en stabil produktion
Der er store perspektiver i produktion af lægemidler i gensplejsede husdyr og afgrøder. Produktionsformen vil kunne bruges til at producere billige lægemidler og være til gavn for udviklingslandene. Dette rejser dog også en række nye problemstillinger for både lægemiddelfirmaet, landmanden og samfundet.
Hønseæg kan bruges til at høste lægemidler, der produceres af gensplejsede høns. Roslin Instituttet i Edinburgh, der klonede fåret Dolly, har vist, at æggene er en realistisk mulighed (4866). Dette forskningsgennembrud kan betyde, at hønseavl i fremtiden vil producere lægemidler af typer, som det ellers kan være vanskeligt at fremstille.
Man har længe været klar over, at gensplejsede høns vil kunne producere farmaproteiner i æggehviden. Men man har ikke haft større held hermed. Roslin Instituttet har nu for første gang opnået en stabil produktion. Dette viser, at metoden kan udnyttes i praksis.
I alle tidligere forsøg faldt produktionen i løbet af nogle generationer af høns, og man konstaterede meget store individuelle forskelle i produktionen, fordi hønsene var fremavlet som kimærer (dvs. at kun nogle af cellerne i hønsene var gensplejsede). Disse problemer har man undgået ved at bruge virus (lentivirus), der med større held frembringer gensplejsningen.
Den nuværende produktion af proteinlægemidler er ofte tidskrævende og kostbar, og man har problemer med at optimere teknologien. I stedet ville man altså kunne bruge hønseæg.
Helt op til 54% af æggehviden i et hønseæg udgøres af ét bestemt protein, kaldet ovalbumin. Helen Sang og medarbejdere fra Roslin Instituttet indsatte human-gener i kyllingefostre i nylagte æg. Generne var designet, så de kun aktiveredes i celler, som havde aktive gener for ovalbumin-produktion, dvs. kun i celler, som producerer æggehvide. Derved kunne man opnå, at æggehviden i æg fra disse høns kom til at indeholde proteinlægemidlet.
Man demonstrerede princippet i to forskellige forsøg: I det ene forsøg indsatte man genet for et monoklonalt mikroantistof, som kaldes miR24. Dette antistof vil kunne bruges mod en ondartet hudkræftform. Man kunne vise, at hudkræft-antistoffet kun blev dannet i de rørformede kirtelceller i hønens æggeleder, og f.eks. ikke i de naboliggende overfladeepithelceller. Desuden kunne man vise, at produktionen af proteinlægemidlet ikke aftog med tiden. Man undersøgte indholdet i hvert tiende æg, som en høne lagde, indtil det 150. æg. Produktionen forblev på samme niveau, og man konstaterede, at produktionsniveauet blev nedarvet til kyllinger af de gensplejsede høns.
Man lavede også et forsøg, hvor man indsatte menneskets gen for beta-1a-interferon, som er et immunforsvarsprotein mod virusangreb, og kunne vise, at interferonet fra æggene var effektivt mod virus.
Æggelederne hos høns har celler, som hver indeholder ca. 100.000 molekyler af mRNA, som er kopier af genet for ovalbumin-proteinet. Hver enkelt af disse mRNA-molekyler kan lave mange molekyler af ovalbumin. Alt i alt bliver det i gennemsnit til 2,2 gram ovalbumin-protein i hvert hønseæg. Desuden kan der dannes proteinlægemidler, f.eks. 1/2000 af ægproteinet. Det vil sige, at en gensplejset høne kan producere ca. 1 mg proteinlægemiddel i hvert æg. Man vil kunne lave en stor kyllingefarm, hvor alle hønsene lægger æg, hvor noget af æggehviden består af et ønsket proteinlægemiddel. Bare for at give en fornemmelse af størrelsesordenen, så vil Hongkongs samlede kyllingepopulation, som til enhver tid er på ca. 1-2 mill. høns, kunne producere 1 kg af et proteinlægemiddel, hver gang hver høne lægger et æg. Og da proteinlægemidler er meget aktive, er dette meget store mængder.
De gensplejsede høns fik indsat de fremmede menneskegener ved hjælp af et lentivirus. Dette virus kan fremkalde anæmi hos heste, men virusset havde fået fjernet alle dets sygdomsfremkaldende gener.
Ud fra en således gensplejset hane og almindelige høns kunne man avle kyllinger, som er bærere af genet. I gennemsnit var hver 25. kylling bærer af det fremmede gen i Helen Sangs forsøg.
Man har længe vidst, at gensplejsede husdyr vil kunne producere farmaceutiske proteiner i modermælken, blodet, urinen mv. Sådanne proteinlægemidler kan f.eks. være de såkaldte monoklonale antistoffer, som bruges til at behandle sygdomme, såsom kræft og leddegigt. Forsøg har tidligere vist, at gensplejsede får, geder, kvæg, kaniner, hamstere og høns kan bruges til produktion af lægemidler på disse måder. Af de nævnte vil høns være de letteste at bruge ved storproduktion. Høns formerer sig hurtigere. Derfor vil man hurtigt kunne opskalere til en stor produktion. Høns er desuden billigere at holde end husdyr. Det er muligt, at æggene endog kan producere proteinlægemidler, som ikke ville kunne produceres af pattedyr, fordi de ville være giftige for pattedyrceller. Høns kan udstyre proteinerne med passende kulhydrat-sidekæder, og måske vil proteiner fra fugle accepteres bedre af immunsystemet hos pattedyr.
Artiklen her bragtes i BioNyt nr. 136 s.4 (marts 2007)
Test dig selv
Du kan tegne abonnement på BioNyt: Videnskabens verden her!
/DNA/Gentek/Farma/Dyr/Det korte svar er: GTC Biotherapeutics fik patent om at malke geder antitrombin-lægemidlet ATryn
GTC Biotherapeutics malker geder for det nu i Europa godkendte antitrombin-lægemiddel ATryn. Firmaet har for nylig fået et patent, som gælder i USA frem til år 2021, og som ifølge firmaet selv dækker produktion af alle terapeutiske proteiner i mælk fra alle transgene pattedyr.
Talrige forsøg har vist, at lægemidler kan produceres i mælken fra gensplejsede husdyr. I teorien vil farma-dyr kunne bruges til at producere insulin til diabetikere, blodstørkningsmidler til blødere og mange andre proteiner.
Det første godkendte lægemiddel, der er produceret af et gensplejset husdyr, er antitrombin-3 (ATIII) med handelsnavnet ATryn. Dette protein udskilles i mælken hos gensplejsede geder, fordi mælkekirtelcellerne indeholder et menneskegen for ATIII, som er vigtigt for menneskets evne til at kunne styre og bremse unødvendig blodkoagulation.
Brandsårsofre og patienter, der gennemgår en hjerteoperation, har brug for lidt mere af dette protein for at blive hurtigere raske. Men ATIII skal især bruges ved arvelig antitrombin-mangel, der giver risiko for blodpropper i dybtliggende blodkar. Tilstanden findes hos 1 ud af 3000-5000 personer, på grund af medfødt fejl i det gen, som danner proteinet antitrombin, så de ikke danner nok af proteinet i deres lever. Disse mennesker får blodfortyndende medicin, såsom Warfarin, men det er farligt under en fødsel eller operation. De får så i stedet antitrombin fra donorblod, hvilket giver risiko for sygdomsoverførsel (som da tusinder af blødere fik HIV, fordi de modtog blodstørkningsfaktorer fra HIV-inficerede bloddonorer). Efter bløderskandalen indførte man i 1980’erne nye “bioreaktorer” i form af celler fra ovarierne af kinesisk hamster. Cellerne blev gensplejset, så de producerede menneskeprotein, og cellerne dyrkedes, så man fik en stor proteinproduktion. Men det er kostbart og vanskeligt for sådanne celler at lave større proteinmolekyler, og ofte har proteinerne ikke de korrekte egenskaber.
I stedet kan man lade geder producere menneske-antitrombin. Det Europæiske Agentur for Lægemiddelvurdering (EMEA) i London afviste i første omgang at godkende ATryn fra geder (fordi firmaet kun havde afprøvet stoffet på ganske få patienter med den sjældne sygdom), men sagen blev genoptaget og produktet godkendtes i sommeren 2006 (bl.a. fordi også resultaterne fra forsøg med gravide patienter blev inddraget). ATryn-proteinet fra gensplejset gedemælk vil i 2007 blive markedsført i Europa.
ATryn-produktet ejes af GTC Biotherapeutics, Inc. (Genzyme Transgenics Corporation Biotherapeutics) i Massachusetts. Dets aktier blev med ét slag 85% mere værd, da EU godkendte produktet, men det var efter at firmaets værdi få måneder før var faldet 40%, da EU i første omgang afviste produktet. Disse værdiudsving var dog intet imod, hvad firmaet tidligere havde oplevet: Da troen på firmaet var højest, var aktieværdien 50 gange højere end i 2006. Det konkurrerende hollandske firma Pharming Group, som nogle år før var tæt på at gå konkurs, blev 15% mere værd, da ATryn blev godkendt (4965).
Pharming Group har sendt en ansøgning til den samme EU-myndighed, som godkendte ATryn. Deres lægemiddel Rhucin virker mod arvelig angioødem (dvs. opsvulmede blodkar) med smertefuld, undertiden dødelig opsvulmen af de bløde væv. Det skyldes en defekt C1-inhibitor (et naturligt humanprotein). Rhucin er en human C1-inhibitor, der er produceret i transgene kaniner (4932). Pharming Group laver også førkliniske forsøg med lactoferrin mod infektion og fibrinogen til lukning af sår.
I Paris er firmaet BioProtein Technologies i gang med at udvikle et rotavirus-protein. Dette firma bruger også kaniner.
Farmalandbrug udførtes også af firmaet PPL Therapeutics, et spinoff fra Roslin-instituttet, der klonede fåret Dolly i 1996. Da PPL Therapeutics ikke skabte kommercielle produkter gik det konkurs med sine 3-4000 transgene får i New Zealand. (Dolly døde iøvrigt af lungesygdom som 6-årig og er nu udstoppet på Edinburgh museum)
GTC Biotherapeutics frembragte deres første gensplejsede geder for 15 år siden. De injicerede befrugtede gede-ægceller med menneskets gen for antitrombin-protein. Genet indsatte sig selv i gedefosteranlæggets DNA, og det blev derefter implanteret i en rugemor-ged.
En af de brunstribede geder, som producerer ATryn, hedder “Sweetheart”. Sweetheart og 60 andre geder bor på en farm i Massachusetts i USA. Disse geder bringer tankerne hen på de gamle grækeres gud Pan, der var delvis menneske og delvis ged, og som forsvarede athenerne mod perserne.
Da GTC Biotherapeutics lavede de første forsøg med at indsætte et menneskegen i geder, blev kun 5% af kiddene bærere af menneskegenet. Efterhånden opnåede man bedre resultater, men gennembruddet kom først, da Roslin-instituttet i Skotland klonede fåret Dolly. På GTC Biotherapeutics anvendte man metoden på de humanprotein-producerende geder og fik pludselig en næsten 100% succesrate.
Det videre forløb så simpelt ud: I stedet for at bygge en biotekfabrik til flere milliarder kr. behøvede man bare at avle flere geder, hvis man havde brug for mere medicin. Investorerne var ellevilde – i en periode. Medicinalindustrien blev nemlig bedre til at håndtere gensplejsede celler i store ståltanke, og investorerne mistede troen og dermed deres investeringer.
Harry M. Meade, der er videnskabelig leder af GTC Biotherapeutics, var dog overbevist om, at ståltanke-cellernes problem ville bestå: De var stadig meget dårlige til at lave meget komplekse proteiner. Nu har GTC Biotherapeutics så fået deres første godkendelse (i Europa) af et humanprotein fra gedemælken, og håber på en FDA-godkendelse i USA i løbet af 2 år. Hvis firmaet får succes, kan de en dag opnå et salg på over ½ milliard dollar om året, idet GTC også arbejder med at producere human-albumin og humant alfa-1 antitrypsin (AAT) i gedemælk. Disse proteiner er ligesom antitrombin komplekse molekyler, som i dag kun kan skaffes fra donorblod. Endvidere forsøger GTC Biotherapeutics at udvikle en vaccine mod malaria samt et CD137-antistof, som kan få kræftknuder til at skrumpe ind, og firmaet samarbejder med Merrimack Pharmaceuticals i Boston om en eksperimentel behandling mod leddegigt. Harry M. Meade håber, at hans geder en dag producerer midler, som kan bruges mod infektionssygdomme som f.eks. HIV.
Der er også en dansk vinkel: Den 3. januar 2007 meddelte GTC Biotherapeutics, at det danske firma LEO Pharma i Ballerup skal stå for markedsføringen af ATryn i hele Europa. De gensplejsede geder bliver dog på GTC Biotherapeutics’ farm i Massachusetts i USA. Kun proteinet sendes til Danmark.
LEO Pharma skal også stå for undersøgelse af ATryn til behandling mod DIC. Det er en komplikation ved alvorlig blodforgiftning. DIC rammer ca. 220.000 patienter i Europa og ca. 250.000 i USA, og halvdelen dør heraf. DIC eller ”Dissemineret intravaskulær koagulering” betyder ”spredt-i-kroppen-forekommende inde-i-blodkarrene størkning-af-blod”. Når kroppen udsættes for en alvorlig bakterieinfektion, så bakterierne begynder at formere sig i blodkarrene, kan de blive spredt i hele kroppen. Bakterierne afgiver forskellige giftstoffer, som kan medføre, at kroppens koagulationssystem kommer ud af balance. Der sker bl.a. det, at der herved forbruges antitrombin-3, der som omtalt modvirker blodpropper. Manglen på dette stof medfører, at der rundt i kroppen dannes mange bittesmå blodpropper inde i blodkarrene. Det kan være livsfarligt, hvis sådanne små blodpropper samler sig på uheldige steder. Med ATryn-behandling vil man formentlig kunne hjælpe kroppen med at få genopbygget lageret af antitrombin-3. I første omgang skal Leo Pharma lave et fase-2 forsøg med deltagelse af 200 patienter for at fastlægge den optimale dosis af ATryn. Senere skal der laves et fase-3 forsøg med flere patienter.
Ifølge GTC Biotherapeutics kan hver enkelt ged producere, hvad der svarer til 90.000 blodkollektioner. Denne produktionsmetode vil derfor på dramatisk måde kunne gøre kostbare lægemidler billigere i fremtiden. GTC Biotherapeutics har talrige patenter på området. Nexia har f.eks. måttet købe patentlicens hos firmaet til sine geder. I 1989 fik Harry Meade sammen med vennen Nils Lonberg USA-patent på at udskille transgene proteiner i pattedyrmælk. Harry Meade har siden sammen med andre opfindere bl.a. fået et patent, som kun angår produktion af transgent antitrombin-3 i gedemælk. Dette patent blev udstedt til Genzyme i 1998. GTC Biotherapeutics blev etableret i 1993 som et spinoff-firma fra det store biotekfirma Genzyme.
”Genetic pharming”
Test dig selv
Du kan tegne abonnement på BioNyt: Videnskabens verden her!
/DNA/Gentek/Farma/Planter/Det korte svar er: Mange afgrøder fra bananer til kartofler
Den første farmagodkendelse gik til en ged, og ingen farmaafgrøder er blevet godkendt, selv om det er mange år siden, at man fremstillede en bønneplante med indhold af vaccine mod mund- og klovsygevirus. Et enkelt blad indeholdt vaccine nok til 200 doser(204).
Bananer: Boyce Thompson Institute for Plant Research ved Cornell universitetet nær New York fremstillede en bananplante med hepatitisvaccine. Bananplanten kunne efter gensplejsning producere et antigen fra den ydre kappe af hepatitis-B virusset (ref.204).
Bananplanter vil muliggøre vaccineproduktion i u-landene, og det ville koste måske 1000 gange mindre end anden vaccineproduktion. 10 hektar bananplantage ville give bananer nok til at vaccinere alle børn under 5 år i Mexico mod hepatitis B. Bananmos på dåse kunne sikre barnet tilstrækkelig dosis af vaccinen(204). Bananer vil kunne indeholde vacciner mod mæslinger, gul feber, difteri og polio, samt diarré, som dræber mange børn i u-landene.
Kartofler: Hepatitis B antigener er også blevet overført til kartofler. Boyce Thompson Institute har vist, at rotter vaccineret med vaccine fra kartoffelplanten blev immune mod hepatitis B.
Charles Arntzen fra Arizona State University i Tempe har påvist, at rå kartofler, der danner hepatitis-B virus-proteiner, kan få mennesker til at danne antistoffer mod dette virus. Brugen af kartofler vil kræve, at de ikke koges før de spises, da vaccinen ellers ødelægges.
Diarré i u-landene skyldes ofte kolibakterier. Mus, der spiste kartofler, der danner antigener fra kolibakterier, blev immune mod bakterierne (ref.204).
Tomater: Man har fremstillet transgene tomatplanter, som danner et glycoprotein fra rabiesvirussets overflade. Planten dannede proteinet i både blade og frugter. Man har tidligere påvist, at dette protein aktiverer immunforsvaret i dyr og virker beskyttende mod rabies. Man håber, at tomatplanten med rabiesvaccinen vil lede frem til en vaccine, som virker blot indtaget via munden.
Rurik Salyaev fra et plantefysiologisk institut i den sibiriske by Irkutsk har fremstillet en gensplejset tomatplante, som indeholder dele af generne fra HIV-virus og HBV-virus. Tomatplanten producerer proteiner fra disse virusgener. Når tomaterne spises opnås en vaccination, ligesom man kender det fra poliovaccine: Når mus spiste en opløsning af tomaterne udviklede de høje niveauer af antistoffer i deres blod mod begge virusformer. Antistofferne kunne genfindes på musenes slimhindeoverflader, dvs. netop dér, hvor HIV-virus og hepatitis-B-virus angriber kroppen. Rose Hammond fra USA's landbrugsministerium samarbejdede med russerne og er imponeret over resultaterne (4867).
Lægen vil ikke behøve at have tomater på lager. Vaccinen vil kunne bringes på pilleform. Det vil gøre dosering af vaccinen lettere. I udviklingslandene vil man kunne undgå at skulle opbevare vaccinen i køleskab, og man vil kunne undgå at skulle bruge nåle. En sådan vaccine vil formentlig være billig og måske kunne produceres på stedet. Når det gælder HIV, mangler man dog, at vaccinen virker på T-cellerne. Antistoffer mod HIV er ikke beskyttelse nok.
Medicin fra valmuer :På Tasmanien dyrker man valmuer til medicinsk brug. I et samarbejde mellem CSIRO, Johnson & Johnson Research og Tasmanian Alkaloids forsker man i, hvilke nye farmaceutiske stoffer, man kan få valmuer til at producere i stedet for morfin. Morfin bruges, fordi det kan laves om til kodein. Måske vil man kunne ændre valmuen, så planten selv laver kodein i stedet for morfin. Valmuer kan også lave andre vigtige lægemidler (4975).
Man har kunnet blokere for dannelsen af et enzym, codeinon-reduktase, som har betydning for et trin sent i plantens syntese af morfin (4975). Når man stopper dannelsen af dette enzym opbygger planten et lager af et alkaloid, som kaldes reticulin, og som dannes undervejs i syntesen mod morfin. Det kan bruges som udgangspunkt for forskellige vigtige farmaceutiske stoffer.
Insulinproduktion i planter?: Vil human-insulin kunne produceres af planter? Et firma i Canada, Symbiosis Genetics, er godt på vej. I kimbladene af planter, som danner olieholdige frø, ophobes olier i kugleformede legemer (oleosomer), som er omgivet af proteiner. Firmaet fandt ud af, at man kan ændre på disse proteiner uden at det påvirker strukturen og funktionen af olielegemerne. Det vil være muligt at indsætte et lille peptid i dette protein, f.eks. et peptid som insulin. Peptidet vil koncentreres omkring olielegemerne, og på grund af olielegemernes særlige fysiske egenskaber vil man let kunne udtrække peptidet af planten. Man vil derefter kunne udklippe peptidet af proteinet og få et insulin-produkt (4975).
Udviklingen af plantegensplejsning: De første genmodificerede planter i landbruget havde f.eks. evne til at tåle ukrudtsmidler eller modstandsdygtighed mod insektangreb samt forskellige hybrid-egenskaber.
Man arbejder nu på at udvikle planter med modstandsdygtighed mod sygdomme, stress-tolerance samt afgrøder med sundere olier (flere ikke-mættede fedtsyrer og færre transfedtsyrer), allergi-frie eller vitaminer-rige planter. (4975)
I fremtiden vil komme en tredie bølge med planter med indhold af lægemidler eller som laver plast eller olielignende kemikalier til industrielle anvendelser.
Ved hjælp af såkaldt RNA-interferens kan man lukke udvalgte gener, f.eks. har man derved ændret oliesammensætningen i bomuld og canola-raps, hvorved man har nedsat mængden af de skadelige mættede fedtstoffer.
Når polyumættede fedtstoffer omdannes industrielt til mono-umættede fedtstoffer ved hydrogenering dannes sundhedsskadelige transfedtsyrer i produktet, og mennesker, som spiser sådanne fødevarer, får skadeligt højt cholesterol-tal. Man har fået planter til at lave færre polyumættede fedtstoffer og flere mono-umættede.
Den "gyldne ris": Den såkaldte "gyldne ris" er resultatet af indsættelsen af en ny biokemisk reaktionsvej i risplanter, som derved kan producere beta-karoten, som menneskets krop omdanner til vitamin A.
Risplanten blev lavet ved at indsætte tre gener fra mælkebøtte og en jordbakterie, som koder for produktionen af beta-karoten (ß-karoten).
Man har senere forbedret denne "gyldne ris", så mælkebøttegenet for phytoen-syntase (det første trin i biosyntesen) er erstattet af et gen fra majs (4975). Dette medførte 20 gange højere koncentration af ß-karoten end ved de første forsøg, og dermed tilstrækkelig høj koncentration til, at det vil være rimeligt at indsætte genet i fødevarer, navnlig i udviklingslande med stor A-vitaminmangel. 100 g ris dagligt ville give et tilstrækkeligt A-vitamintilskud.
Omega-3 fedtsyrer: Omega-3-fedtsyrerne DHA og EPA, som vi kan få fra fisk (eller i piller), er blevet anset for det vigtigste "funktionelle" levnedsmiddeltilskud. Planter danner kun den første fedtsyre, alfa-linoleinsyre (ALA), og fortsætter ikke med at producere de mere langkædede og mere polyumættede EPA og DHA. Man har i mikroalger fundet gener, der bidrager med de manglende biosyntese-trin, og ved at indsætte algegenerne i planter har man kunne forlænge plantefedtsyrernes kædelængde og umættethed. Tre forskergrupper har på den måde fået gåsemad (Arabidopsis thaliana) til at producere DHA (4975).
Jordnødder: Nogle planter er allergifremkaldende på grund af et indhold af visse stoffer (allergener). Disse stoffer kan være yderst farlige, undertiden dødelige, og nogle børn dør af jordnøddeprodukter. Man kender proteinerne, som medfører denne farlige allergiske reaktion, samt de gener, som styrer deres dannelse. Det vil derfor være muligt at lave planter, som mangler disse gener.
Olieplanten Ricinus: Olieplanten (Ricinus communis) indeholder et meget giftigt stof, ricin.
Udnyttelse af denne afgrødeplante ville blive meget lettere, hvis denne egenskab blev fjernet (4975).
Olieholdige planter: En anden mulighed i plante-genteknologien er at indsætte et nyt gen. Man har hentet et gen fra den vilde plante Crepis palaestina, som producerer en usædvanlig fedtsyre, der er industrielt anvendelig, og indsat genet i en hørplante, som derefter producerede denne fedtsyre i hørfrøolien. I dette tilfælde kunne en ny fedtsyre produceres ved at indsætte blot ét gen (4975).
Plaststoffer: Polyhydroxy-alkanoater er en gruppe af bionedbrydelige plaststoffer, som er baseret på en monomer med tre carbonatomer, og med sidekæder af forskelligt antal carbonatomer. Polyhydroxy-butyrat er en monomer, opbygget af fire carbonatomer, og produceres af en jordbakterie ved hjælp af kun tre enzymer ud fra molekyl-byggesten, som allerede findes i planter. Det vil derfor være muligt at indsætte gener for disse tre enzymer i f.eks. en gåsemad (Arabidopsis thaliana) og senere opsamle polyhydroxybutyrat fra grønkornene i planten. Et firma ved navn Metabolics forsøger at forfølge dette mål for at lave planteplast (4975).
(Artiklen her bragtes i BioNyt nr. 136 s.16-17 (marts 2007))
Test dig selv
Du kan tegne abonnement på BioNyt: Videnskabens verden her!
/DNA/Gentek/Farma/Planter/Det korte svar er: Måske vil det være vanskeligt for landbruget at håndtere tomater og æg, som har et indhold af farmaceutiske stoffer, - for produkterne ligner jo almindelige fødevarer
Der er ingen vejskilte til GTC Biotherapeutics' gedefarm. Man ønsker ingen dyreretsforkæmpere, som måtte ønske at "befri" gederne. For geder, som laver edderkoppespind-proteiner, har naturligvis rørt folks fantasi. Edderkoppespind i gedemælk lyder klamt - selv om der blot er tale om harmløse proteinfibre. Nogle mener, at det er synd for geden, "som ikke mere ved, om den er en ged" - selv om geden naturligvis er ligeglad. Nogle får fantasier og skriver: "vi vil ikke have giraf-jordsvin, som kan vises i zoologiske haver, så folk kan le af dem" (dette argument vedkommer ikke forskningen her, geden ligner enhver anden ged, og hvis "morsomme" dyr blev lavet, ville det enten blive kaldt kunst eller være en del af underholdningsindustrien; alt, selv Internettet, kan jo misbruges; det berettiger ikke til at hævde, at brugbar forskning og livreddende medicinudvikling skal ophøre). Nogle mennesker kommer til at tænke på små ottebenede geder, som spinder edderkoppespind, eller edderkoppemennesker (nu er vi i begge tilfælde virkelig for langt ude i fantasien) (4938).
Ud over at forske må forskerne altså også forholde sig til den debat, som kan opstå i samfundet. F.eks. hvis nogen bekymrer sig om, "at skellet mellem dyr og menneske nedbrydes" (dels kan mennesket ikke defineres ved dets individuelle gener, dels findes mange gener i identisk form hos alle biologiske dyreorganismer, og altså inklusive mennesket).
Det er også blevet nævnt, at menneskegener, som er transplanteret ind i husdyr, kan "lække" ud i levnedsmidlerne, nemlig hvis et gensplejset husdyr fik lejlighed til at få afkom med et ikke-gensplejset husdyr (hertil er at sige, at menneskeproteiner i sig selv ikke er farlige, ligesom det jo heller ikke er farligt at spise koprotein, svineprotein, gedeprotein osv. I øvrigt vil proteinerne blive nedbrudt. Det er f.eks. baggrunden for, at man ikke kan tage insulin i pilleform. Så end ikke den medicinske virkning vil blive opnået ved at spise et transgent dyr).
Alligevel vil man næppe tillade, at transgene dyr kommer på køkkenbordet. Det viser debatten i USA efter en sag i 2002, hvor University of Illinois ved Urbana-Champaign sendte 386 svin, som var blevet brugt til transgene forsøg, til slagteren. Kun ét af svinene indeholdt transgener, men svinene var ikke blevet godkendt af den amerikanske sundhedsstyrelse FDA til kommerciel brug. Ingen af svinene endte på køkkenbordet, men det skabte alligevel en del kritik (4931).
Lægemiddelproteiner, der er produceret ved farmalandbrug, kan dog tænkes at medføre nogle risici ved den tiltænkte anvendelse som et lægemiddel: Der kan tænkes at være smitsomme prioner i produktet (prioner er årsag til kogalskab og CJD-sygdom hos mennesker), eller der kan tænkes at fremkomme en immunreaktion (som ved katteallergi), eller der kan tænkes at opstå autoimmunsygdom (hvis kroppen begynder at angribe egne tilsvarende proteiner). Men disse risici findes også ved andre produktionsmetoder: F.eks. fremstilles nogle terapeutiske proteiner ud fra museceller eller gærceller, og dyrkningsmedierne indeholder ofte blodvæske fra kalvefostre. Uden blodvæske danner cellerne ikke altid de korrekte lægemiddelmolekyler (4981).
Måske vil det være vanskeligt for landbruget at håndtere tomater og æg, som har et indhold af farmaceutiske stoffer, - for produkterne ligner jo almindelige fødevarer. Ikke mindst når der tænkes på at lade udviklingslande producere lægemidler i sådanne "bioreaktorer" i landbrugsomgivelser, kan man have sine betænkeligheder. Selv i USA med dets gigantbødestraffe har man ikke altid kunnet overholde de restriktioner på gensplejsede afgrøder, som man har opstillet. I 2002 opdagede man, at der voksede farma-majs på marker med almindelige sojabønner i Iowa og Nebraska. Majsen lavede et farmaceutisk protein, idet nogle plantefrø fra en tidligere majsproduktion, som firmaet ProdiGene i Texas havde gjort forsøg med, senere spirede frem på markerne. Det kostede firmaet 3 millioner dollar til oprensningsudgifter og 1/4 million dollar i bøde (4868).
Man vil kunne nedsætte risikoen for spredning ved at bruge et system med hansterile planter, som ikke producerer pollen, eller ved at man efter dyrkning af farma-afgrøden efterfølgende dyrker herbicidresistente afgrøder på markerne, og der så sprøjtes med herbicid, som dræber alle de tidligere dyrkede farma-planter.
Da Charles Arntzen fra Arizona State University i Tempe indsatte gener fra det diarréfremkaldende Norwalk-virus i tomatplanter for at lave en vaccine mod virusset, byggede universitetet i den anledning et forseglet drivhus med finmaskede insektnet, dobbeltdøre, styrede luftstrømme og andre forholdsregler, som skal forhindre insekter og plantefrø i at komme ind eller ud. Desuden er det planen at gøre planterne sterile, og at bruge en hvid tomat med savsmuldssmag, så risikoen for at komme til at spise tomaterne ved en fejltagelse undgås (4868).
Charles Arntzen er dog noget bekymret over, at hvis myndighedernes restriktioner bliver for skrappe, vil farma-vacciner ikke kunne få den anvendelse, som de måske fortjener (4868).
Artiklen her bragtes i BioNyt nr. 136 s.19 (marts 2007)
Test dig selv
Du kan tegne abonnement på BioNyt: Videnskabens verden her!
/DNA/Gentek/Patent/Det korte svar er: Mindst hver sjette gen er i USA omfattet af patentrettigheder
Man kan spørge: Hvordan kan man tage patent på noget, som vi alle bærer rundt på? Det kan man ved at begrænse patentet til industriel anvendelse af genet eller til isolering af genet i f.eks. en dyrkningsbakterie eller egnet gærcelle, som kan dyrkes. Patenteringen af gener tog fart, da man begyndte at kortlægge det humane genom. Dengang kunne man tage patent på et gen, dvs. patentere det og alle dets funktioner. Sådan er det ikke i dag. I dag er det typisk brugen af en genetisk opdagelse, der bliver patenteret.
Forskere fra Massachusetts Institute of Technology (MIT) er nået frem til, at 4.382 (okt. 2005) af de knap 24.000 gener, der er registreret i USA’s nationale genetiske database, er omfattet af såkaldte intellektuelle rettigheder eller er patenterede.
Test dig selv
Du kan tegne abonnement på BioNyt: Videnskabens verden her!
/DNA/Gentek/Patent/Det korte svar er: I Canada blev 250 patentansøgninger på dyr sat i venteposition for at afvente oncomus-afgørelsen
Den amerikanske højesteret besluttede i 1980 for første gang at tillade patent på en levende organisme med argumentet, at den var menneskeskabt. Det var en mikroorganisme, som var gensplejset til at nedbryde olieforurening. Det fik de amerikanske patentmyndigheder til at godkende patenter på mange levende organismer, tilmed hele dyr, herunder oncomusen, som er en mus, der ved gensplejsning er blevet bærer af et kræftfremkaldende gen, og som derfor er af betydning for kræftforskningen.
Også en canadisk appelret bestemte i 2000, at Harvard Medical School kunne få patent på oncomusen. Det blev dermed slået fast, at genetisk ændrede pattedyr kan patenteres i Canada (4961).
Oncomus-patentet gjaldt alle pattedyr undtagen mennesket, hvis de blev tilsvarende gensplejset. Patentet er givet til Harvard Medical School, men de er overdraget til DuPont, som derfor har eneret til patentet og som hævder at have patentrettigheder til alle kræftprodukter, som udvikles på grundlag af denne oncomus. I Canada blev 250 patentansøgninger på dyr sat i venteposition for at afvente oncomus-afgørelsen.
I begyndelsen stillede DuPont oncomusen til rådighed for grundforskning for relativ billig betaling og uden restriktioner. Men i 1988 lavede DuPont en aftale med Charles River Laboratories om at avle oncomus, og det lå i aftalen, at der skulle betales afgifter, hvis anvendelsen af musen førte til produkter. Restriktionerne medførte, at mange forskere holdt op med at bruge oncomusen.
Man har diskuteret, om der nu er åbnet mulighed på patentering af alle dyr. F.eks. fik University of Missouri i 2001 et patent, som dækker kloning af alle pattedyr, og som ikke udelukker mennesker. Patentet dækker en måde at få ubefrugtede æg til at blive til embryoner, samt produktion af klonede pattedyr med denne teknik.
University of Wisconsin har et patent på foster-stamceller fra mennesket. Det er celler, som er udtaget af fostervæv og som kan udvikle sig til mange eller måske alle forskellige vævstyper. Universitetet har givet firmaet BioTransplant i Massachusetts licens til at udnytte patentet i svin. Dette firma arbejder på at udvikle svin, som kan anvendes som organdonorer til mennesker (4962).
Både de amerikanske, de canadiske og andre patentmyndigheder tillader patenter på menneskegener og menneskeceller. I 1997 udstedtes patent på det klonede får Dolly. Roslin Instituttet i Skotland fik et patent, som dækker anvendelse af kloningsteknologien i alle dyr, i øvrigt også mennesket. Kloning af mennesker udelukkes i mange lande af anden lovgivning, og desuden sagde Roslin Instituttet, at de havde medtaget mennesket for at sikre sig, at inden andre fik patent på kloning af mennesker. Omvendt er det altså nu blevet accepteret, at man i princippet kan patentere mennesker. Patentering af menneskeorganer skulle derfor også være en mulighed (4961).
Metoder kan også patenteres. Man vil f.eks. kunne patentere følgende: En forskergruppe ved Mount Sinai Hospital i Toronto, Canada, har haft held med at høste menneskeæg fra rygmusklerne af gnavere. Tanken er, at menneskeæg kan opbevares i mus, hvis kvinden skal gennemgå en medicinsk behandling, som kan skade hendes æg (4961).
Som de transgene dyr viser, kan det være meget vanskeligt at få overskud af forskning, selv efter mange års forskningsindsats. Det er baggrunden for, at patenter kan forsvares. Men der findes også samlebåndspatenter, som ret let kan fås, men som blot virker bremsende på udnyttelsen af den nye viden. Det gælder patenter på gener eller dele af gener. Det amerikanske patentkontor begyndte på et tidspunkt - til alles store overraskelse - at udstede patenter på gener. Måske en femtedel af menneskets gener er derfor nu patentbelagt i USA eller andre lande.
Patenter på gener fremmer ikke forskning, de hæmmer snarere forskningen. Den arvelige stofskiftesygdom, som kaldes Canavans sygdom, er et eksempel: Den rammer børn fra 3-måneders alderen, så de hverken kan kravle eller gå. De bliver lamme og dør i de tidlige ungdomsår af hjerteanfald. En gruppe forældre, som var bærere af sygdommen, hyrede en forsker til at påvise genet og lave en gentest. Genet blev fundet i 1993, og familierne lavede en aftale med et hospital i New York om at tilbyde gratis test til alle, der måtte ønske det. Men forskerens arbejdsgiver, Miami Childrens Hospital Research Institute tog imidlertid patent på genet og krævede royalties fra alle, som ville udføre testen. Da familierne var imod patentering, skrev de ikke under på patentaftalen, og de fik derfor ingen indflydelse på aftalen.
Mange firmaer oplever, at de ikke kan udføre relevant forskning og udvikling, fordi patenthaverne forlanger alt for mange penge. Patenthaveren kan nemlig forlange, hvad han vil. En brystkræft-test er f.eks. blevet højt prissat til 3000 dollar. Da genet er privatejet, forhindres andre i at udvikle en billigere test. En patient kan heller ikke donere sit syge gen til en forsker, hvis en anden ejer patentet.
Ejeren af virusgenet for hepatitis C indkasserer millioner af dollars fra forskere, som vil studere sygdommen, og mange forskere vælger i stedet at studere mindre omkostningstunge sygdomme.
Da SARS bredte sig, var nogle forskere betænkelige ved at studere det, fordi eventuelle patenter kunne komme på tværs af deres forskning (4998).
Også hele sygdomsorganismer, såsom virus, er blevet patentbelagt. Mindst 20 sygdomsfremkaldende organismer er i dag privatejet, bl.a. hæmophilus-influenza.
Patenter kan medføre, at man spilder penge til unødvendig medicin: En bestemt type astmamedicin virker kun på visse patienter. Men producenten forhindrer alle i at udvikle genetiske test, som kunne påvise, hvilke patienter medicinen ikke vil virke på. Genpatenterne ødelægger derved muligheden for den personlige medicin - for der er flere penge i, at ingen ved, at din medicin ikke virker på dig! Genpatenterne blev i sin tid givet med den begrundelse, at genet var kopieret, og at kopien derfor var menneskeskabt. Men i praksis betød det, at også selve genet blev patentbelagt. Hvis man i stedet indførte den regel, at det naturlige gen ikke kunne patenteres, ville der kunne skabes en sund konkurrence om at lave den bedste og billigste test på genet.
I USA er der begyndt at komme ansøgninger om patent på sekvenser af DNA, som er mindre end hele genet. Sådanne DNA-fragmenter har ingen kendt funktion; alligevel er der blevet udstedt patenter. Tidligere ville det ikke kunne lade sig gøre. Det er overladt til fremtidige, kostbare retssager at afgøre, om den senere patenthaver af hele genet skal betale licens til den, som tidligere fik patent på en del af genet (4967).
Den amerikanske lægeorganisation, American Medical Association, har foreslået, at man ikke skal kunne tage patent på lægeprocedurer, f.eks. kirurgiske metoder. (I Europa er dette område faktisk udelukket fra patentering).
Et patent gælder kun i de lande, hvor patentet er godkendt og holdes aktivt med et årsgebyr, - i 20 år fra ansøgningstidspunktet. Alle lande i verden har nu indført denne 20-års regel. (Artiklen her bragtes i BioNyt nr. 136 s.20-21) (marts 2007)
Test dig selv
Du kan tegne abonnement på BioNyt: Videnskabens verden her!
/DNA/Genterapi/Det korte svar er: Indførsel af nyt genetisk materiale i organismen med det formål at opnå et terapeutisk resultat
Man kan definere genterapi som indførsel af nyt genetisk materiale i organismen med det formål at opnå et terapeutisk resultat. Det vil sige, at genterapi er en måde at behandle eller forebygge sygdomme på, der er baseret på indførsel af genetisk materiale i patientens celler.
Det genetiske materiale, der overføres, kan enten være i form af DNA eller RNA, men da DNA er mere stabilt end RNA vil metoden med brug af DNA være nemmere. Hvis man bruger DNA, vil man ikke være i stand til at kontrollere den mængde protein, der evt. vil blive dannet. Dette vil til dels være nemmere med RNA.
Genterapi kan enten udføres på kønsceller eller på somatiske celler (kropsceller).
Genterapi på kønsceller vil medføre, at det efterfølgende individ og dets afkom vil have genet i fremtiden. Denne form for genterapi er forbudt hos mennesker.
Genterapi på somatiske celler vil medføre, at den genetiske ændring ikke overføres til næste generation, men kun kommer det ”inficerede” individ til gode. Det er denne form for genterapi, som vil få størst betydning i Danmark og i resten af verden.
Test dig selv
Du kan tegne abonnement på BioNyt: Videnskabens verden her!
/DNA/Genterapi/Det korte svar er: Til behandling af mange typer af sygdomme, f.eks. ved at indsætte et nyt gen til at erstatte et evt. ødelagt gen
Man forventer, at genterapi vil kunne bruges til behandling af mange typer af sygdomme. Formålet med at indsætte et nyt gen til at erstatte et evt. ødelagt gen vil være at fjerne årsagen til en sygdom, - modsat i dag, hvor medicin bruges til at nedsætte symptomerne og ikke helbreder sygdommen.
Kort kan man sige, at genterapi kan bruges til:
- At erstatte et muteret/defekt gen med et rask gen. Dette kunne være nyttigt inden for sygdomme, som f.eks. cystisk fibrose.
- At indsætte et gen, som medfører, at cellen undergår apoptose (dvs. programmeret celledød). (Læs her og mere
her).
Kan bruges ved f.eks. kræftbehandling.
- At indsætte et gen, som hæmmer virusreplikation, eller som udtrykker et bestemt protein. Kan bruges ved HIV/AIDS eller Huntingtons chorea
Der er mange flere muligheder, som vi ikke vil komme ind på her, men igen er der inden for en lang række behandlingsmuligheder med genterapi en del etiske overvejelser, som kan komme på tværs.
Test dig selv
Du kan tegne abonnement på BioNyt: Videnskabens verden her!
/DNA/Historie/Det korte svar er: Et grundforskningscenter om geologisk og biologisk fortid
Center for Geogenetik er et grundforskningscenter, der blev officielt åbnet 7. sep. 2010, og hvor stort set alt arbejdet handler om noget med fortiden. Der er dels ansat geologer og dels genetikere.
Professor Eske Willerslev fortæller: “Vi udvinder DNA fra gamle sedimenter, og kan på den måde rekonstruere, hvordan tidligere tiders økosystemer af planter og dyr hang sammen, vi leder f.eks. efter de sidste spor af mammutter. I 2007 var Eske Willerslev den danske forsker, som tiltrak sig mest international opmærksomhed: To artikler i Science og en i PNAS skabte mediestorm tre gange dette år om tre forskellige emner. Journalister fra bl.a. CNN, NBC og New York Times ringede utallige gange dagligt i fire dage, og skulle hver gang have den samme forklaring. Eske Willerslev fortæller, at den store medieomtale medførte ansøgninger fra udenlandske forskere, som gerne ville arbejde i hans afdeling. Medens der dengang var 23 ansatte, og er der nu 60 ansatte, når alt personale medregnes.
Test dig selv
Du kan tegne abonnement på BioNyt: Videnskabens verden her!
/DNA/Historie/Det korte svar er: Professor på Center for Geogenetik. København
I 2005 blev han som 33-årig ansat som den yngste professor på Københavns Universitet som resultat af sit pionerarbejde på gammelt DNA, og siden har han og hans gruppe fundet DNA i knogler, hår, lorte, jord, søvand og ved at besøge museer rundt i verden eller tage på 1-2 ekspeditioner om året, typisk en måned af gangen. Eske Willerslev fik i 2011 den såkaldte "videnskabsministerens forskningskommunikationspris" for at formidle sine banebrydende DNA-opdagelser på en spændende og engagerende måde. Et af de største forskningsmæssige gennembrud var, da han sammen med Anders J. Hansen viste, at fossilt DNA kan være bevaret i gamle, ikke-forstyrrede sedimenter uden så meget som skyggen af et fossil. De fandt ud af, at en jordklump på 2 gram (på størrelse med en sukkerknald) er nok til at finde DNA fra de dyr og planter, som havde været på stedet. Hvis jordklumpen f.eks. er fra tiden for 20.000 år siden, så kan man finde ud af, hvilke dyr og planter der var på stedet for 20.000 år siden!
Test dig selv
Du kan tegne abonnement på BioNyt: Videnskabens verden her!
/DNA/Historie/Det korte svar er: De enæggede tvillinger Eske Willerslev og Rane Willerslev, begge professorer
De enæggede tvillinger Eske Willerslev og Rane Willerslev blev født i 1971 og boede i den pæne københavnerforstad Gentofte. De har i bl.a. radio og i bøger fortalt om deres opvækst. Trods deres som udgangspunkt identiske DNA og levested, var deres miljø ikke identisk. Rane var mest mors dreng, og Eske var så mest fars dreng, især da han et år som 10-årig boede sammen med faderen, der havde arbejde i Sverige. Moderen var en beskyttende lærer, medens faderen, Richard Willerslev, var 56 år ved tvillingernes fødsel og historieprofessor ved Københavns Universitet. Han tog dem med på sine rejser for at studere svenske folkevandringer i 1800-tallet, og tvillingerne tilbragte somrene i familiehytten i en svensk skov. “Vores far var temmelig hård - vi blev sendt ud i isfyldt vand og skulle f.eks. hugge mængder af brænde,” har de fortalt.
De lærte at snitte med kniv, sejle kano, skyde med gevær og allerede som 13-årige var de alene i Lapland, hvilket var en kæmpeudfordring. Deres første ekspedition til Sibirien var i 1991, det år hvor Sovjetunionen brød sammen, hvilket de først fik at vide to måneder efter, fordi de havde befundet sig ude i tundraen. I 1992 var de sammen i nord-Kamchatka. I sommeren 1993 tog Rane og Eske sammen på en fossil-indsamlingstur i det østlige Sibirien. Turen blev udvidet til et fem-måneders vinterophold med blandt jægerfolket jukagirerne i NØ-Sibirien. Senere rejste de alene med hver deres fagfæller. De er begge medlem af Eventyrernes Klub.
Trods deres som udgangspunkt identiske DNA og levested, var deres miljø ikke identisk. Rane var mest mors dreng, og Eske var så mest fars dreng, især da Eske Willerslev et år som 10-årig boede sammen med faderen, der havde arbejde i Sverige.
Rane Willerslev har levet som pelsjæger blandt de indfødte ved Altai-bjergene nær ved grænsen til Mongoliet. Han skrev ph.d.-afhandling om de sibiriske jukagireres åndetro, og blev professor i social antropologi ved Aarhus Universitet, leder af De Etnografiske Samlinger på Moesgård Museum og tiltrådte 1.oktober 2011 stillingen som direktør ved Kulturhistorisk museum ved Universitetet i Oslo. Han har skrevet en bog i 2009 om oplevelserne i Sibirien (blandt andet et forsøg på at indføre andelsbevægelsen blandt pelsjægere. Dette forsøg blev brutalt forhindret af russisk mafia, som myrdede den lokale leder af andelsselskabet. Selv måtte Rane Willerslev, forfulgt af det korrupte politi, tilbringe 1 år som ufrivillig pelsjæger (ref.9571).
Rejserne tændte deres interesse for at vide mere om, hvordan mennesker klarer sig i den oprindelige natur og på naturens betingelser.
Eske Willerslev har rejst på langrendsski, brugt hundeslæde og jaget elge. Han var i Sibirien som pelsjæger sammen med en erfaren, lokal jæger fra Jakutien og en ung russer, som oprindelig var elektriker, men som på grund af den økonomiske krise 1999-2000, hvor folk ikke fik løn, havde slået sig på jagt. Eske Willerslev sultede faretruende i en periode, hvor han jagede zobel sammen med russeren. De havde startet deres jagttur 300-400 km fra nærmeste landsby, og med et forråd bestående af to skudte elge, som mere erfarne jægere havde skudt til dem. Men fordi de havde stor succes med zobeljagten, som følge af en medbragt hund med talent for at jage zobel op i et træ, fik de aldrig bygget et stativ til elgkødet. Den ene elg blev ædt af en jærv, og den anden af ulve. Eske Willerslev har også oplevet at ligge vågen 5 timer en nat, hvor teltet blev opsøgt af to bjørne.
“Jeg troede ikke mine sibiriske fældefangst-erfaringer ville kunne bruges i min videnskabelige karriere", siger Eske Willerslev, "men det lærte mig, hvor langt jeg kan skubbe krop og sind. Jeg synes, at jeg kan arbejde dobbelt så længe som nogle kolleger”.
Han anvendte sin udholdenhed i sit akademiske arbejde ved Københavns universitet som kandidatstuderende, hvor Eske Willerslev i to år knoklede med at påvise, at man kan udvinde fossilt DNA fra iskerner, og senere som ph.d-studerende (for private penge, fordi Eske Willerslev fik nej fra udvalget, som skulle godkende hans ph.d.-projekt), hvor han netop påviste, at det er muligt at udvinde gammelt DNA fra snavset is i polariskerner og fra permafrostjord - og afleverede opgaven som en doktordisputats. I 2003 fik Eske Willerslev et stipendium til at arbejde hos Alan Cooper i hans laboratorium på Oxford Universitet i England. Alan Cooper er en fremtrædende forsker af gammelt DNA.
Eske Willerslev vendte i 2005 tilbage for at etablere Danmarks første laboratorium for gammel DNA, som senere voksede ind i Center for GeoGenetik, som Eske Willerslev opbyggede med 50 millioner kroner fra det danske forskningsråd og 25 millioner kroner i statstilskud. Eske Willerslev blev professor som 33-årig som palæogenetiker ved Københavns Universitet.
I en bog der skrevet i 2008 beskriver Eske Willerslev sin tilgang til genetikstudierne. Bogen hedder "Fra pelsjæger til professor: En personlig rejse gennem fortidens dna-mysterier", af Eske Willerslev (Jyllands-Postens forlag. 183 sider, 249 kroner). Han har også skrevet andre bøger, bl.a. sammen med sin enæggede tvillingebror Rane Willerslev.
Test dig selv
Du kan tegne abonnement på BioNyt: Videnskabens verden her!
/DNA/Hoppende_gener/Det korte svar er: DNA-segmenter der bevæger sig fra et sted på kromosomet til et andet
I 1940´erne studerede Barbara McClintock nedarving i majsplanter. Hun fandt ud af, at visse DNA-segmenter kunne bevæge sig fra et sted på kromosomet til et andet, og at de endda kunne hoppe fra et kromosom til et andet. McClintock fandt ud af, at disse ”hoppende gener”, som i dag kaldes transposoner, kan lande midt i et gen, og ødelægge genet.
Test dig selv
Du kan tegne abonnement på BioNyt: Videnskabens verden her!
/DNA/Hoppende_gener/Det korte svar er: I alle slags organismer
Mobilt DNA findes i alle former for levende organismer, bl.a. planterm dyr, bakterier og archae-organismer. (9785s.1)
Test dig selv
Du kan tegne abonnement på BioNyt: Videnskabens verden her!
/DNA/Hoppende_gener/Det korte svar er: Det er meget varierende
Jo større genomet er hos en art, jo mere mobilt DNA indeholder dens genom. Gærceller og fisken pufferfish (Takifugu rubripes) (der har små genomer) indeholder kun få procent mobilt DNA, medens byg og majs (der har store genomer) indeholder 60-70% mobilt DNA. Mennesket og andre pattedyr har også store genomer, og indeholder ca. 50% DNA som er eller har været mobilt. (9785s.5)
Test dig selv
Du kan tegne abonnement på BioNyt: Videnskabens verden her!
/DNA/Hoppende_gener/Det korte svar er: Forskellige typer af “retro-transposoner” og “DNA-transposoner”
Blandt det mobile DNA i mennesket er 75% af den type, som kaldes retrotransposoner. Gærceller (såsom øl-gær) indeholder også retrotransposoner, faktisk er al det mobile DNA af denne type, men det er af en anden type end hos mennesket. Hos rundormen C. elegans er 90% af det mobile DNA af en type, som kaldes DNA-transposoner. Man ved ikke hvorfor nogle organismer har en type medens andre organismer har en anden type af mobilt DNA. (9785s.5)
Test dig selv
Du kan tegne abonnement på BioNyt: Videnskabens verden her!
/DNA/Hoppende_gener/Det korte svar er: “DNA-transposoner” (cut/paste) og tre typer af “Retro-transposoner” (copy/paste)
Man inddeler mobilt DNA i forskellige typer, kaldet DNA-transposoner (der udklipper og indsætter DNA uden at der sker duplikation af DNA-stykket, dvs. at DNA-stykket flyttes), og retrotransposoner ((der udklipper og indsætter DNA under kopiering, således at der sker duplikation af DNA-stykket, dvs. at DNA-stykket bevares på det oprindelige sted). Retrotransposonerne opdeles i tre grupper, kaldet LTR (dominerer i planter) og non-LTR (dominerer i pattedyr). (LTR = long terminal repeat, fordi de har gentagelser i enderne). Begge koder for proteiner, men non-LTR ender i en lang A-A-A-A-hale. Den tredie gruppe af retrotransposoner koder ikke for proteiner, og kaldes non-autonome retrotransposoner (og de kræver revers-transskriptase og endonuklease fra LINE1- og L1-elementer). (9785s.6)
Test dig selv
Du kan tegne abonnement på BioNyt: Videnskabens verden her!
/DNA/Hoppende_gener/Det korte svar er: “DNA-transposoner” har cut & paste funktion
DNA-transposoner er mobilt DNA, som især findes i bakterier og visse andre organismer. Der er ca. 6% DNA-transposoner i menneskets genom, men der kendes ikke aktive DNA-transposoner i pattedyr (som mus og primater). DNA-transposoner (der udklipper og indsætter DNA uden at der sker duplikation af DNA-stykket, dvs. at DNA-stykket flyttes), og retrotransposoner (der udklipper og indsætter DNA under kopiering, således at der sker duplikation af DNA-stykket, dvs. at DNA-stykket bevares på det oprindelige sted). (9785s.7)
Test dig selv
Du kan tegne abonnement på BioNyt: Videnskabens verden her!
/DNA/Hoppende_gener/Det korte svar er: Hoppende elementer kan bl.a. bruges til identifikation, evolutionsstudier og i retsgenetik.
Hoppende gener (mobilt DNA) såsom transposoner kan bruges til at studere evolutionen i både nulevende arter og uddøde arter. F.eks. kan de bruges til at studere artsdannelsen blandt mammutter. Studier af menneskets retrotransposoner har vist, at i de fleste humane loci har de ældste alleler de højeste hyppigheder blandt afrikanere, hvilket tyder på at de har afrikansk oprindelse. Dette tyder på en nylig afrikansk oprindelse af det moderne menneske. Allel-frekvenser af Alu-retrotransposoners polymorfisme passer ikke til dette synspunkt ved at vise, at mønsteret af øgede allel-frekvenser i Afrika ikke er så fremtrædende som hidtil antaget. Der er en stærk sammenhæng for de proteinkodende loci, men ikke for Alu-loci og ikke-kodende loci. Dette kunne tyde på at sammenhængen, når det gælder de proteinkodende loci, skyldes noget andet, dvs. en anden faktor, som varierer med datasættene. En sådan faktor kunne være mutationshyppigheden i de forskellige loci. Den afrikanske sammenhæng er nemlig størst for de loci, hvor der er den højeste mutationsrate. Nyere forskning har dog påvist, at L1-elementernes data passer godt sammen med de tidligere data for cellekerne-DNA og mitokondrie-DNA og støtter derved teorien om, at det moderne menneske har en afrikansk oprindelse. Hoppende elementer kan også bruges til identifikation og i retsgenetik. (9785s.45-47)
Test dig selv
Du kan tegne abonnement på BioNyt: Videnskabens verden her!
/DNA/Hoppende_gener/DNA-transposoner/Det korte svar er: Et mobilt DNA-stykke, som var meget anvendt til genetisk forskning i bananfluer
P-elementet hos bananfluen Drosophila melanogaster er et berømt DNA-transposon (mobilt DNA-element). Visse han-fluer (P står for paternal, dvs. faderlig) der krydsedes med visse hun-fluer (M for maternal, moderlig) fik afkom, som døde eller var sterile og med høje mutationshastigheder og øget omflytning af kromosomdele og rekombination. De fluer, der er P-bærere, har kopier af et DNA-transposon, kaldet “P-elementet”, som M-fluerne manglede. Derimod manglede M-fluerne en somatisk proteinhæmmer for transposition. P-elementets opdagelse var nyttig, fordi det kunne bruges til at påvise gener hos bananfluerne. P-elementet koder for et transposase-enzym. Man kunne vise, at der var tale om det fænomen, som kaldes “lokale hop” (local hopping). Det forklares ved en direkte DNA-til-DNA transposition. Næsten hver gang der skete et sådant lokalt hop mistedes nogle baser på de berørte steder. Indtil bananfluens genom blev bestemt i 2001 var P-elementet derfor afgørende for meget af bananflue-genetikken. (9785s.22)
Test dig selv
Du kan tegne abonnement på BioNyt: Videnskabens verden her!
/DNA/Hoppende_gener/Retrotransposoner/Det korte svar er: Retrotransposoner afkodes til RNA og videre til protein (i cellens cytoplasma), og dette protein danner sammen med RNA'et et kompleks, som kan vandre tilbage og ind i cellekernen
Retrotransposoner afkodes til RNA og videre til protein (i cellens cytoplasma), og dette protein danner sammen med RNA'et et kompleks, som kan vandre tilbage og ind i cellekernen. Disse komplekser kaldes “ribonucleoprotein-partikler”, RNP. (Non-LTR retrotransposoner virker direkte på DNA-strengen, der klippes over på et sted ved hjælp af en endonuklease, som non-LTR-elementet selv indeholder og reverstranskriptase-enzymet virker så direkte på DNA'et i cellekernen, således at RNA'et tjener som skabelon, Endonuklease-skæringsstedet virker som en primer for reverstransskriptasen). Specielt non-LTR-retrotransposonerne har været kraftige motorer for evolutionsprocessen. De har givet genomerne plasticitet og diversitet gennem over en halv milliard år.
Mennesket har aktive non-LTR-retrotransposoner, som kaldes L1-elementer. F.eks. kan et L1-element i et menneske transskriberes til RNA, som transporteres ud i cytoplasmaet og her ved translation danner to proteiner, som i en såkaldt cis-præference sammenkobles med RNA-stykket og derved danner en RNP-partikel, som importeres til cellekernen, hvor revers-transskription og integration af det nydannede L1-DNA sker i et-enkelt-trin på et nyt kromosomsted.
L1-elementerne kan også drive indsættelsen af andre elementer, som kaldes Alu og SVA. De kan også medføre fænomenet “pseudogener”, som er mRNA, der via revers-transskriptase er blevet omkodet til DNA og indsat i kromosomerne som et slags gen (men typisk uden promotor, dvs. den aktivitetskontrol, som normale gener har, og genet vil derfor ofte være uvirksomt - med mindre en nærliggende promotor kan bruges). Der er over 500.000 L1-elementer i menneskets genom, heraf mindst 100 aktive. Der er desuden over 1 mill. Alu-elementer i menneskets genom. Der kendes 3000 SVA og 8000 aktive pseudogener. Menneskets genom indeholder DNA-transposoner og LTR-retrotransposoner, men de synes alle at være inaktive hos mennesket. (9785s.13, 17 (fig), 18)
Test dig selv
Du kan tegne abonnement på BioNyt: Videnskabens verden her!
/DNA/Hoppende_gener/Retrotransposoner/Det korte svar er: Der kendes mange sygdomme som skyldes retrotransposoner
I mennesket kender man nu 70 sygdomme, som skyldes mobilt DNA. I mennesker, mus, hunde og andre dyr har man konstateret sygdomme, som skyldes naturlige indsættelse af retrotransposon-elementer i cellens DNA. Man taler om cis-typer og trans-typer. I en cis-begivenhed koder det specifikke retrotransposon for dets egne proteiner, som har betydning for dets mobilitet. I en trans-begivenhed bliver elementet mobiliseret ved hjælp af proteiner, som stammer fra en anden retrotransposon. Den første opdagelse af et sygdomsvoldende mobilt DNA-element hos et menneske var opdagelsen af et L1-element, som medførte A-hæmofili i 1987 hos en patient JH-27. (9785s.16, 53, 67)
Test dig selv
Du kan tegne abonnement på BioNyt: Videnskabens verden her!
/DNA/Hoppende_gener/Retrotransposoner/Det korte svar er: Det varierer meget mellem forskellige typer organismer, fra 80% til 0,1% af mutationerne
Hos mennesket er retrotransposoner skyld i 0,1% af mutationerne , men hos mus er de skyld i ca. 10% af mutationerne, og hos bananfluen er de skyld i 80% af mutationerne. Genom-analyser tyder på, at der var en stor opblomstring af retrotransposon-aktivitet i både L1-elementer og Alu-elementer i primaternes udviklingsrække for ca. 40 milliioner år siden, og at der senere har været faldende retrotransposon-aktivitet, og meget lav aktivitet i nutiden hos primaterne og mennesket. (9785s.18)
Test dig selv
Du kan tegne abonnement på BioNyt: Videnskabens verden her!
/DNA/Hoppende_gener/Retrotransposoner/Det korte svar er: Ja, men de er inaktive
I menneskets genom er omkring 8% endogene retrovirus, som dog alle er inaktive. (Idet en eller flere af deres gag-, pol- og env-gener er defekte). I bananfluen er Gypsy-elementet et aktivt endogent retrovirus. (9785s.32)
Test dig selv
Du kan tegne abonnement på BioNyt: Videnskabens verden her!
/DNA/Hoppende_gener/Retrotransposoner/Det korte svar er: Chimpansen har dobbelt så mange polymorfe L1-elementer i forhold til mennesket.
Mennesket og chimpansen har stort set lige mange L1-elementer, men chimpansen har dobbelt så mange polymorfe L1-elementer i forhold til mennesket. Dette tyder på, at den effektive størrelse af populationen af chimpansens forfædre var større end størrelsen af populationen af menneskets forfædre. I chimpanse-genomet har to L1-familier fortsat med at udvikle sig i de sidste 5 millioner år. Menneskets genom indeholder ca. 17 forskellige underfamilier af L1-elementer. På ethvert tidspunkt over de sidste 40 mill. år har dog kun een underfamilie af L1-elementer været aktiv. I de sidste 25 mill. år er der opstået 5 underfamilier af L1-elementer i primatlinien. Det vil sige at for 25 mill. år siden opstod en ny underfamilie af L1-elementer, som efter nogle få millioner år uddøde og blev efterfulgt af en ny underfamilie af L1-elementer og så fremdeles. Man kender til mindst 10 forskellige mekanismer, hvorved L1-elementer og andre retrotransposoner kan ændre genomet hos et pattedyr, f.eks. ved at blande kortene, når det gælder exon-dele af generne eller ved at påvirke i hvilken grad forskellige gener er aktive eller lader sig aktivere. (9785s.44,54)
Test dig selv
Du kan tegne abonnement på BioNyt: Videnskabens verden her!
/DNA/Kilder/Det korte svar er: Bl.a. i hår, søvand osv.
Ud over at alle organismer indeholder DNA har man fundet DNA i søvand og jord, og gammelt DNA i knogler, hår, ekskrementer, permafrostlag, is og ved at besøge museers samlinger (af hår, væv og knogler).
Test dig selv
Du kan tegne abonnement på BioNyt: Videnskabens verden her!
/DNA/Kilder/Alder/Det korte svar er: Ud fra mutationshyppigheder
Selve aldersbestemmelsen af DNA er et speciale i sig selv. Det sker ved, at man ser på forskelle i genomet. Man ved noget om, hvor hyppigt forskellene sker, og på den baggrund kan man foreslå en alder.
Test dig selv
Du kan tegne abonnement på BioNyt: Videnskabens verden her!
/DNA/Kilder/Jord/Det korte svar er: Ja, dette er bekræftet ved studier i Grønland på gammel nordboplads.
Man har undersøgt, hvor anvendeligt det vil være at bruge DNA-analyser til arkæologiske undersøgelser. Det har man fået svar på ved at lede efter DNA på en gammel nordbo-bosættelse i Grønland (ref.9643). Stedet kaldes “Gården under sandet” (GUS, "The Farm Beneath the Sand"). (Se "Hvad er Gården Under Sandet?"). Et stykke fra den arkæologiske udgravning tog man DNA-prøver i et område, som dengang havde været åbne marker til gården. DNA-prøverne viste, at en søjle af jord fra en tidligere mark kan fortælle, hvilke dyr, der græssede her. Der er gjort traditionelle arkæologiske fund i nærheden, og den rækkefølge af begivenheder, der er blevet påvist ved de gamle metoder med at grave i jorden og lave analyser af spor efter husdyr, korrelerede godt med den historie, der kunne udledes af analysen af det gamle DNA på stedet! Dette betragtes som en revolution for den arkæologiske videnskab, og potentialet for kortlægning af menneskers, dyrs og planters arkæologiske udvikling er dermed formidabel (ref.9534).
Gården Under Sandet blev etableret i det første tiår af 1000-tallet. Det blev forladt ca. år 1400. Man har på stedet påvist 8 forskellige perioder, hvor der ikke var insekter af den type, som findes ved menneskers bygninger (synantropiske insekter). Dette tyder på, at gården ikke blev brugt uafbrudt i hele perioden. Men der var selv i disse pe- rioder en insektfauna, som viser, at der har været husdyr på stedet, formentlig dyr, der tilhørte nærliggende gårde. I prøverne fandt man DNA fra mennesker, kvæg, får, geder og mus. Desuden fandt man i en prøve, som kunne dateres til ca. år 1243 (+/- 38 år), noget DNA, der var 100% identisk med rensdyr-DNA (undergruppen Odocoileinae med 97% sandsynlighed ifølge genbank-standarder). Denne undergruppe af rensdyr findes kun i Grønland. Det fundne menneske-DNA må skyldes senere forurening, for det blev kun fundet i prøver fra efter nordbotiden (nemlig i lag fra år 1450-1520). DNA fra husdyrene blev fundet, selv om sedimenterne har været våde og ufrosne i længere tid, og selv om DNA’et er fra et sted, som dengang lå hen som ubeskyttet, åben mark. DNA-undersøgelserne viste udviklingen over tid. Kvæg, får og geder var til stede i hele nordbo-tiden på stedet (år 1040-1365). Mængden af fåre-DNA er relativ stabil over tid. Mængden af kvæg-DNA varierer derimod gennem tiden med den overordnede tendens, at der var en tilbagegang over tid. Der var ikke DNA fra husdyr i de øverste lag, efter at området blev opgivet af nordboerne. Koncentrationen af kvæg-DNA er utypisk, fordi man i sådanne undersøger typisk vil finde, at koncentrationen aftager med dybden. Faldet i kvæg- DNA i perioden fra år 1180 til 1260 afspejler derfor sandsynligvis den faktiske historie på stedet i denne periode, hvor gården blev opgivet, men hvor markederne dog stadig lejlighedsvis anvendtes af nærliggende gårde til afgræsning af husdyr. Resultatet er i overensstemmelse med knogle-fundene på stedet, der tyder på, at antallet af kvæg på gården faldt i den undersøgte periode i forhold til vilde dyr. Sælknogler udgør 28% af alle de dyreknogler, der er fundet i gårdens bygninger fra ca. år 1000-1150, og 41% fra perioden år 1300-1400. I samme periode faldt fund af kvægknogler fra 15% til 6%, mens knogler af får/geder steg fra 27% til 33-35%. Samlet set er overensstemmelsen mellem kvæg-DNA og de fossile data (kvæg-knogler) betydelig. Kvægknoglerne blev fundet i udgravninger inde i gården, medens kvæg-DNA’et er fra den menneskeskabte jord, der akkumuleredes rundt om gården. De arkæologiske udgravninger og DNA-prøverne udgør to uafhængige beviser, som fortæller forskellige dele af den samme historie og understøtter hinanden. Tilstedeværelsen af rensdyr-DNA taler for, at der har været åbne græsarealer tæt på gården - med vilde rensdyr, der græssede i sensommeren. Gammelt DNA kan altså bevares i menneskeskabte sedimenter - i det mindste på en historisk tidsskala. DNA-forskerne opfordrer nu arkæologer til at overveje steril prøvetagning af sedimenter i og omkring arkæologiske udgravninger for at muliggøre senere studier af DNA-profilering (ref.9534).
Test dig selv
Du kan tegne abonnement på BioNyt: Videnskabens verden her!
/DNA/Kilder/Jord/Det korte svar er: Ja, bl.a. i stabile, gamle sedimenter
Prof. Eske Willerslev har sammen med Anders J. Hansen påvist, at fossilt DNA kan være bevaret i gamle sedimenter uden så meget som skyggen af et fossil. De fandt ud af, at en jordklump på 2 gram (på størrelse med en sukkerknald) er nok til at finde DNA fra dyr og planter, som havde været på stedet. Hvis jordklumpen f.eks. er fra tiden for 20.000 år siden, så kan man finde ud af, hvilke dyr og planter der var på stedet for 20.000 år siden!
Test dig selv
Du kan tegne abonnement på BioNyt: Videnskabens verden her!
/DNA/Kilder/Jord/Det korte svar er: Ikke på de tidlige studier
Studiet af gammelt DNA begyndte i 1984, da man undersøgte DNA fra en quagga, en uddød zebra-variant fra Sydafrika. Man brugte et eksemplar, som havde stået på et museum i 160 år. På det tidspunkt, hvor man udførte dette studie, vidste man ikke, om DNA'et ville være ødelagt.
Senere publicerede man fund af DNA i mumier, der var 2000-3000 år, DNA i planter, der var 5-10 millioner år gamle, i insekter fra rav, der var 20-30 millioner år gammelt, og dinosaurknogler, der var 80 millioner år gamle. Men da man i 1990'erne gik disse forskellige studier igennem igen, viste det sig, at ofte var det DNA, som man havde fundet, faktisk fra de forskere, som havde udført arbejdet og nogle gange var det DNA fra den kyllingesandwich, som forskerne havde spist. Det medførte, at man blev mere skeptisk over disse DNA-studier. - Men i dag har billedet ændret sig. Man har lært at forhindre eller begrænse DNA-forureningen, og der er kommet langt bedre maskiner til DNA-sekvensbestemmelse (ref.9583).
Test dig selv
Du kan tegne abonnement på BioNyt: Videnskabens verden her!
/DNA/Kilder/Jord/Det korte svar er: Ja, hvis forholdene har været stabile
Undersøgelser af permanent frosne sedimentprøver, der er op til flere hundredetusind år gamle, har overraskende vist, at det indeholder sedimentært- DNA, "sedaDNA", altså genetiske spor af uddøde organismer fra tidspunktet for sedimenteringen. sedaDNA kan bruges som erstatning for fossiler, hvis disse mangler, forudsat at der ikke er sket fysisk tilførsel af ældre lag eller opblanding med nyere sedimentaflejringer. En af de teknikker, man kan bruge til kontrol, er OSL (optisk stimuleret luminescens), som viser, om en prøve har været udsat for lys. For at vise, at sedaDNA kan være upåvirket af både ældre og yngre sedimenter, har man undersøgt permafrosne vådområder på Taimyr-halvøen i Sibirien samt ved en flod i Alaska. Man fandt, at ikke alle permafroststeder egner sig til sedimentære DNA-studier. Småsøer er mere sikre, hvorimod floder kan have omlejret sedimenterne. Vindaflejret DNA kan være brugbart på steder, hvor det tidligere DNA er eroderet væk (ref.9532).
Man har fundet gammelt DNA, der stammer fra forskellige mikroorganismer, planter og dyr, herunder mammut, hest, bison og moskusokser. DNA fra 2-gram prøver har muliggjort detaljerede beskrivelser af palæoøkosystemer i Sibirien, Nordamerika og New Zealand. For første gang har man kunnet forbinde fortidens dyr og planter i tid og rum, selv i mangel af makro-fossiler. Ved iskerneboring tværs gennem Grønlands indlandsis kunne man hente direkte DNA fra 450.000-800.000 år gammel, mudderholdig is, der er blevet opbevaret i al denne tid på bunden af Grønlands indlandsis. Det viste spor fra planter og dyr, som slet ikke er på Grønland i dag, bl.a. beviser på nåleskov i Grønland (Willerslev et al. 2007).
Man har undersøgt, hvor anvendeligt det vil være at bruge DNA-analyser til arkæologiske undersøgelser. Det har man fået svar på ved at lede efter DNA på en gammel nordbo-bosættelse i Grønland (ref.9643). Stedet kaldes “Gården under sandet” (GUS, "The Farm Beneath the Sand"). (Se "Hvad er Gården Under Sandet?"). Et stykke fra den arkæologiske udgravning tog man DNA-prøver i et område, som dengang havde været åbne marker til gården. DNA-prøverne viste, at en søjle af jord fra en tidligere mark kan fortælle, hvilke dyr, der græssede her. Der er gjort traditionelle arkæologiske fund i nærheden, og den rækkefølge af begivenheder, der er blevet påvist ved de gamle metoder med at grave i jorden og lave analyser af spor efter husdyr, korrelerede godt med den historie, der kunne udledes af analysen af det gamle DNA på stedet! Dette betragtes som en revolution for den arkæologiske videnskab, og potentialet for kortlægning af menneskers, dyrs og planters arkæologiske udvikling er dermed formidabel (ref.9534).
Gården Under Sandet blev etableret i det første tiår af 1000-tallet. Det blev forladt ca. år 1400. Man har på stedet påvist 8 forskellige perioder, hvor der ikke var insekter af den type, som findes ved menneskers bygninger (synantropiske insekter). Dette tyder på, at gården ikke blev brugt uafbrudt i hele perioden. Men der var selv i disse pe- rioder en insektfauna, som viser, at der har været husdyr på stedet, formentlig dyr, der tilhørte nærliggende gårde. I prøverne fandt man DNA fra mennesker, kvæg, får, geder og mus. Desuden fandt man i en prøve, som kunne dateres til ca. år 1243 (+/- 38 år), noget DNA, der var 100% identisk med rensdyr-DNA (undergruppen Odocoileinae med 97% sandsynlighed ifølge genbank-standarder). Denne undergruppe af rensdyr findes kun i Grønland. Det fundne menneske-DNA må skyldes senere forurening, for det blev kun fundet i prøver fra efter nordbotiden (nemlig i lag fra år 1450-1520). DNA fra husdyrene blev fundet, selv om sedimenterne har været våde og ufrosne i længere tid, og selv om DNA’et er fra et sted, som dengang lå hen som ubeskyttet, åben mark. DNA-undersøgelserne viste udviklingen over tid. Kvæg, får og geder var til stede i hele nordbo-tiden på stedet (år 1040-1365). Mængden af fåre-DNA er relativ stabil over tid. Mængden af kvæg-DNA varierer derimod gennem tiden med den overordnede tendens, at der var en tilbagegang over tid. Der var ikke DNA fra husdyr i de øverste lag, efter at området blev opgivet af nordboerne. Koncentrationen af kvæg-DNA er utypisk, fordi man i sådanne undersøger typisk vil finde, at koncentrationen aftager med dybden. Faldet i kvæg- DNA i perioden fra år 1180 til 1260 afspejler derfor sandsynligvis den faktiske historie på stedet i denne periode, hvor gården blev opgivet, men hvor markederne dog stadig lejlighedsvis anvendtes af nærliggende gårde til afgræsning af husdyr. Resultatet er i overensstemmelse med knogle-fundene på stedet, der tyder på, at antallet af kvæg på gården faldt i den undersøgte periode i forhold til vilde dyr. Sælknogler udgør 28% af alle de dyreknogler, der er fundet i gårdens bygninger fra ca. år 1000-1150, og 41% fra perioden år 1300-1400. I samme periode faldt fund af kvægknogler fra 15% til 6%, mens knogler af får/geder steg fra 27% til 33-35%. Samlet set er overensstemmelsen mellem kvæg-DNA og de fossile data (kvæg-knogler) betydelig. Kvægknoglerne blev fundet i udgravninger inde i gården, medens kvæg-DNA’et er fra den menneskeskabte jord, der akkumuleredes rundt om gården. De arkæologiske udgravninger og DNA-prøverne udgør to uafhængige beviser, som fortæller forskellige dele af den samme historie og understøtter hinanden. Tilstedeværelsen af rensdyr-DNA taler for, at der har været åbne græsarealer tæt på gården - med vilde rensdyr, der græssede i sensommeren. Gammelt DNA kan altså bevares i menneskeskabte sedimenter - i det mindste på en historisk tidsskala. DNA-forskerne opfordrer nu arkæologer til at overveje steril prøvetagning af sedimenter i og omkring arkæologiske udgravninger for at muliggøre senere studier af DNA-profilering (ref.9534).
Test dig selv
Du kan tegne abonnement på BioNyt: Videnskabens verden her!
/DNA/Kilder/Jord/Det korte svar er: Bl.a. fra plantedele, hår og knogledele
Det er til dels uklart, hvordan DNA fra planter og dyr kan ende i sedimenter, men teoretiske overvejelser, kombineret med undersøgelser af DNA i moderne jord, peger på, at afkastede rodhætte-celler er den største kilde til plante-DNA i sedimenterne. (Bakterie-enzymer mv. kan lette frigivelsen af DNA i planters rodzone). Andre DNA-kilder er pollen og blade, og ekskrementer, hudflager og keratinmateriale, såsom hår, fjer og negle, er væsentlige kilder til DNA fra hvirveldyr. Disse væv er gået i opløsning, men har frigivet DNA til omgivelserne. Det er interessant, at for både planter og dyr er det gamle DNA af regional oprindelse. Der er kun i ubetydeligt omfang sket spredning af DNA over lange afstande. I virkeligt gamle prøver er der næsten intet DNA til stede. Man har ganske vist ekstremt effektive opformeringsmetoder for DNA (PCR-teknikken), men da der overalt findes DNA fra nutiden, er der en meget stor risiko for, at de små fund af meget gammelt DNA er forurenet med DNA fra nyere tid. Dertil kommer risikoen for, at DNA siver ind eller ud af sedimenterne. Frit DNA bindes dog hurtigt til lerpartikler, sand, humusstoffer og komplekser af mineraler og organiske stoffer, som findes i sedimentet. Jord kan være positivt ladet, og da DNA er negativt ladet kan det måske af denne grund holde sig bedre. Under frosne eller tørre forhold er DNA-sivning et mindre problem. Men i varme og fugtige miljøer er det et stort problem.
Test dig selv
Du kan tegne abonnement på BioNyt: Videnskabens verden her!
/DNA/Kilder/Jord/Det korte svar er: Gammelt DNA
Det er meget gammelt, nedbrudt DNA (ancient DNA, aDNA).
Test dig selv
Du kan tegne abonnement på BioNyt: Videnskabens verden her!
/DNA/Kilder/Jord/Gården_under_sandet/Det korte svar er: Nordbo-gård nær Nuuk
Stedet, der kaldes “Gården under sandet” (GUS, "The Farm Beneath the Sand"), er en gammel nordbo-bosættelse i Grønland. Stedet ligger i SV-Grønland ca. 80 km øst for Nuuk. I 1990 fandt man rester af gården under et 1½ m tykt lag af sand og grus på et plateau omgivet af lave bjerge. I dag er området en sandet ørken med slyngende smeltevandsfloder fra indlandsisen, men på nordbotiden voksede her græs, som husdyrene kunne æde. Gården Under Sandet blev etableret i det første tiår af 1000-tallet. Det blev forladt ca. år 1400.
Test dig selv
Du kan tegne abonnement på BioNyt: Videnskabens verden her!
/DNA/Kilder/Kirkegårde/Det korte svar er: De sociale forhold, tilrejsende osv.
En af Danmarks tidligste kristne kirkegårde er Kongemarken ved Roskilde, der er fra ca. år 1000-1250. De tidlige skandinaviske kristne kirkegårde er kønsopdelte: Kvinder ligger begravet i den nordlige side, mænd i den sydlige. Der var dog undtagelser: I de få tidlige kristne kirkegårde, der er udgravet i Skandinavien, er der altid et par mænd, der er blevet placeret i den nordlige side, og nogle kvinder i sydsiden. På Kongemarken blev flere mænd med sidestillede kvinder fundet på nordsiden af kirkegården. Forskere fra Eske Willerslev's gruppe ekstraherede mitokondrie-DNA fra ni personer, der var blevet udgravet i to forskellige områder inden for kirkegården. Der blev fundet en overraskende mangfoldighed af mitokondrie-haplogrupper: H, I, J, T, T2 og U7. Selv de tre med haplogruppe H var af forskellige undertyper. Dette indikerer, at ingen af personerne inden for hvert område var beslægtet på mødrene side. Den observerede haplogruppe U7 er almindelig i Indien og hos stammer fra det vestlige Sibirien, men ikke fundet i etniske skandinaver. Haplogruppe I er sjælden (2%) i Skandinavien. Disse observationer tyder på, at de personer, der boede i Roskilde-området omkring år 1000, ikke alle var medlemmer af en tæt sammentømret lokalbefolkning, og at der var personer med genetiske forbindelser til befolkningsgrupper, som geografisk var fra steder meget langt væk (ref.9535).
Test dig selv
Du kan tegne abonnement på BioNyt: Videnskabens verden her!
/DNA/Kilder/Kirkegårde/Det korte svar er: Arkæologiske DNA-studier
Arkæologiske DNA-studier kan på engelsk kaldes "archaeogenomics". I bred forstand betyder det, at man anvender DNA til at få viden om menneskets fortid. Tilsvarende kan DNA-baserede studier om fortiden ud over menneskets fortid kaldes "palaeogenomics".
Test dig selv
Du kan tegne abonnement på BioNyt: Videnskabens verden her!
/DNA/Kilder/Kirkegårde/Det korte svar er: Test af spor efter forbrydelser, f.eks. DNA-test
Undertiden kan fortolkningen af arkæologisk DNA forbedres ved at inddrage DNA fra nulevende. Det skete ved identifikationen af skeletterne af den russiske zarfamilie, Nikolaj II og zarinaen Alexandra samt deres fem børn, Olga, Tatjana, Maria, Anastasia og Aleksej, der alle blev myrdet af bolsjevikkerne d. 16. juli 1918. De blev dræbt efter i tre måneder - sammen med familiens læge og tre fra staben - at være holdt fanget i Ipatiev- huset i Jekaterinburg (Sverdlovsk i Sovjettiden). Dermed sluttede Romanov- slægtens næsten 300-årige herredømme over det russiske rige. Der hvor Ipatiev-bygningen stod, er der nu en katedral, Blodskirken. I 1979 fandt man graven i en skov ca. 30 km uden for Jekaterinburg. Den indeholdt skeletter af fem kvinder og fire mænd. I 1994 analyseredes skeletterne for de dele af mitokondrie-DNA, der har særligt hyppige mutationer. Man sammenlignede med DNA fra levende slægtninge af samme kvindelinie til de afdøde (mitokondrier nedarves udelukkende fra moder til børn). Zarinaen var barnebarn af den engelske dronning Victoria. For zaren undersøgtes to slægtninge, der i kvindelinier var efterkommere af zarens mormor (den danske dronning Louise af Hesse-Cassel, der var gift med Christian IX). Man fandt det forventede mitokondrie- DNA hos zarinaen og de tre børn i graven. Zaren havde to forskellige DNA-baser på position 16169 (30 % af hans mitokondrie-DNA havde en T-base her og 70 % havde basen C). Zaren havde altså to forskellige populationer af mitokondrier, idet der var sket en mutation i et mitokondrie. Mutationen kan være opstået i dronning Dagmar (zarens mor), og endt op i hendes ægceller - eller denne heteroplasmi kan have været i zarens mors kvindelinie i nogle få generationer. Heteroplasmien blev ikke genfundet hos den nulevende slægtning til Dagmar. Den var blevet til en homoplasmi (for T) i position 16169 hos denne person. Dagmar var datter af den danske konge Christian IX og dronning Louise. I Rusland, hvor hun nu er begravet, hed hun zarina Maria Feodorovna. Mitokondrier deler sig selv, og viderefører altså mutationen. Efter få menneskegenerationer vil alle celler i kroppen dog hurtigt igen have kun én type mitokondrie-DNA i alle celler - en tilstand, der benævnes homoplasmi. Men indtil da vil der altså optræde to populationer af mitokondrie-DNA hos samme person (heteroplasmi). Skelettet af zarens bror, Georgij Romanov, som var død af tuberkulose i 1899, viste også heteroplasmi i position 16169. I 1920 påstod en Anna Anderson, der var bosat i USA, at hun var Anastasia. Anastasias skelet var nemlig ikke blevet fundet i graven. Mange personer har i tidens løb påstået det samme, men denne Anna Anderson hævdede dette særlig ihærdigt livet igennem. Hun døde i 1984 og blev kremeret, og da hendes lig altså blev brændt kunne man ikke skaffe DNA fra hende, men i 1970 var hun blevet opereret på et hospital i Charlottesville, Virginia. 24 år efter hendes død, i 1994, lavede man en DNA-profilanalyse på væv, som var fjernet ved operationen. Hun var hverken beslægtet med zaren eller zarinaen.
I juli 2007 blev der fundet en ny grav ved Jekaterinburg med brændte knogler fra to individer. På grund af teknologiske fremskridt kunne man undersøge det komplette genom af mitokondrie-DNA'et fra hvert individ samt dele af kerne-DNA’et. Man undersøgte også 1991-graven og zarens kvindelinie blev repræsenteret ved en nulevende slægtning til hans mor. En mandlig linie blev repræsenteret ved mandlige efterkommere af zar Nikolaj I, der var Nikolaj II’s oldefar. Det blev påvist, at knoglerne fra 2007-graven repræsenterede førstegradsslægtninge til medlemmer af 1991-graven. Ingen af Romanov-familien overlevede altså massakren i 1918.
Zaren havde overlevet et attentat i 1891 i forbindelse med et besøg i Japan. Hans blodplettede skjorte opbevares i dag på Hermitage museet i St. Petersborg. DNA fra blodpletterne havde samme sjældne Dagmar-type mtDNA, som i zar-knoglen, og heteroplasmien i position 16169 blev også fundet i blodpletterne. Analyse af Ykromosomet og DNA-markører fra andre kromosomer gav perfekt match mellem blodpletterne og zar-knoglen.
Test dig selv
Du kan tegne abonnement på BioNyt: Videnskabens verden her!
/DNA/Kilder/Vand/Det korte svar er: Ja, i flere dage gamle vandprøver
Ud fra vandprøver fra 100 søer og vandløb fra Europa har forskere på Center for Geogenetik påvist, at DNA'et i vandet viser hvilke arter, der findes der. Man påviste f.eks. tilstedeværende fisk og padder, men fandt samtidig DNA fra arter, som blot lever omkring vandet (f.eks. den lille fugl kærsanger og kronhjort. Man fandt DNA fra dyr så forskellige som odder og guldsmed. Man kan tilmed ved hjælp af DNAstudier søge efter udvalgte arter. Man afprøvede metoden på et fladt tag af en museumsbygning. I såkaldte "mesokosmos" (store baljer med naturligt søvand, smådyr og planter) havde man haletudser af forskellige arter, og kunne vise en sammenhæng mellem mængden af DNA i vandet og tætheden af individer. To uger efter, at dyrene var fjernet, kunne DNA'et ikke påvises mere (ref.9579). Metoden kan bruges som en billig test for den aktuelle biodiversitet i vandløbet. Et snapseglas søvand indeholder DNA fra hele søens dyreliv, og metoden spås stor betydning for overvågningen af naturens mangfoldighed. Hidtil har dette arbejde krævet eksperters møjsommelige artsbestemmelser, der, afhængig af hvad man søgte efter, kræver brug af f.eks. elfiskeri, planktonnet osv. (og stadig færre eksperter tilegner sig et stort artskendskab). Behovet ses af, at hver 4. ferskvandsdyr er truet eller allerede uddød i nyere tid (dvs. at 4600 ferskvandsdyrearter er truede eller uddøde for nylig), og nedgangen i verdens biodiversitet er nu 100 - 1000 gange hurtigere end i tiden før mennesket påvirkede naturen. Artiklen om DNA'et i søen var forsidehistorie i det ansete videnskabelige tidsskrift Molecular Ecology. De tre hovedpersoner bag forskningen er ph.d.- studerende Philip Francis Thomsen og specialestuderende Jos Kielgast og Lars L. Iversen. Da DNA-mængden for en art fortæller om artens bestandsstørrelse, mener forskerne, at det i fremtiden f.eks. kan være muligt at fastsætte fiskekvoter ud fra DNA-spor i stedet for at benytte fangster.
Forskergruppen fokuserede på 6 truede arter fra forskellige dyregrupper:
Padder: Løgfrø (Pelobates fuscus) og Stor Vandsalamander (Triturus cristatus)
Fisk: Dyndsmerling (Misgurnus fossilis)
Pattedyr: Odder (Lutra lutra)
Krebsdyr: Forårsdamrokke (Lepidurus apus)
Insekt: Stor Kærguldsmed (Leucorrhinia pectoralis)
De sjældne dyr blev eftersøgt på lokaliteter, hvor man havde fundet dem med konventionelle, ekspertbaserede metoder (natlytning efter kvæk samt nethøst af haletudser af de to padder, og af guldsmedens larve og krebsdyret, elektrofiskeri af dyndsmerling, spor efter odder) - samt på kontrollokaliteter, hvor de med sikkerhed ikke forekom. Der blev indsamlet 3 x 15 milliliter vandprøver fra 98 naturlige søer, damme og vandløb i Danmark, Sverige, Tyskland, Polen og Estland mellem 2009 og 2011. Alle prøverne blev opbevaret ved minus 20 grader, indtil de kunne undersøges. For de to paddearter blev bestandstætheden på en række lokaliteter desuden bestemt ved hjælp af ketching af larver (den konventionelle undersøgelsesmetode) for at sammenligne tætheden af dyr med mængden af DNA i vandet under naturlige forhold.
På museets tag udførtes forsøg med de samme to arters larver i tætheder på 0, 1, 2 eller 4 dyr pr. 80 liter. Vandprøver blev taget før forsøget og 2, 9, 23, 44 and 64 dage efter dyrenes introduktion til vandbeholderen. Derefter fjernedes dyrene, og man målte DNA i vandprøver 2, 9, 15 og 48 dage efterfølgende. Arterne kunne kendes på deres DNA i mitokondrie-generne for cytochromoxidase I og cytochrom-b. Der var for hver art mindst 10 af de undersøgte vandområder, hvor arten fandtes. DNA-testen virkede 100% for fisken og krebsdyret, 91–100% for padderne og 82% for insektet. I 8 søer, hvor der tidligere var fundet løgfrø, men hvor konventionelle metoder ikke registrerede denne art i undersøgelsessæsonen, kunne man med DNA-metoden bekræfte, at arten stadig fandtes i 5 af søerne, men i mindre bestande. For at teste det videst mulige perspektiv af den nye DNA-baserede naturovervågning i ferskvand undersøgte man 4 søer særlig grundigt. De undersøgte lokaliteter blev udvalgt i samarbejde med Statens Naturhistoriske Museums fiskeatlas-medarbejdere og konsulentfirmaet Amphi Consult, således at man med sikkerhed på forhånd kendte alle tilstedeværende arter. Der var således grundlag for at undersøge, hvor stor en andel af hele faunaer, som kunne genfindes med den nye DNAmetode. Overraskende viste det sig, at arts-specifik DNA fra alle tilstedeværende arter af padder og fisk på de respektive lokaliteter kunne genfindes med vandprøver på størrelse med et snapseglas. Desuden fandt man DNA fra bl.a. kronhjort (Cervus elaphus), kærsanger (Acrocephalus palustris), blishøne (Fulica atra), ringdue (Columba palumbus).
Test dig selv
Du kan tegne abonnement på BioNyt: Videnskabens verden her!
/DNA/Klima/Det korte svar er: Selv om havis forsvandt i nogle områder kunne den være bevaret i andre områder
Havis kommer og går uden at efterlade sig spor, så før satellitovervågning og observationer fra fly og skibe blev indført, vidste man ikke noget om den arktiske havis-udbredelse til forskellig tid. Men Eske Willerslevs afdeling har ændret på dette. De udviklede en metode, hvormed det er muligt at måle variationer i isen flere årtusinder tilbage - baseret på indsamling af data om landbaseret materiale (bl.a. drivtømmer) langs kysten i det nordlige Grønland. Resultaterne af undersøgelserne på land viser altså, hvordan forholdene var i havet. Svend Funder var leder af undersøgelsen, hvor bl.a. også Eske Willerslev og Kurt Kjær deltog. Undersøgelsen viste, at der har været store udsving i mængden af sommerens havis i løbet af de sidste 10.000 år. Under det såkaldte Holocæne klimaoptimum, fra for cirka 8000 til 5000 år siden Holocæn = den helt nye tid, hvor temperaturen var noget varmere end i dag, var der signifikant mindre havis i Det Arktiske Ocean, formentlig mindre end 50% af den havis, som der var i sommeren 2007, som ellers var rekord-lav for havis. Undersøgelserne viste også, at selv om isen forsvinder i et område, kan havis ophobes i et andet arktisk område. Man opdagede dette ved at sammenligne resultaterne med observationer fra det nordlige Canada: Samtidig med, at mængden af havis faldt i det nordlige Grønland, steg det i Canada. Dette skyldtes sandsynligvis ændringer i de fremherskende vindsystemer. Denne faktor indgår ikke i tilstrækkelig grad i vurderingen af den forventede forsvinden af havis i Arktis. Disse overraskende konklusioner har man fået efter adskillige ekspeditioner til Peary Land i Nordgrønland - et sjældent besøgt område, hvor sommersnestorme ikke er ualmindelige. Nøglen til mysteriet om havisens udbredelse under tidligere tids-epoker ligger i det drivtømmer, som man finder langs kysten. Man kunne tro, at drivtømmeret - som navnet siger - er kommet flydende over havet, således, at drivtømmer = frit hav og ikke-drivtømmer = havis. Men det forholder sig modsat! En sådan rejse i frit hav ville kræve mindst 6 års rejse, og tømmeret kan ikke holde sig flydende så længe. "Træet kan kun krydse oceanet, hvis det er indlejret i havis, og mængden af tømmer viser derfor mængden af havis på det pågældende tidspunkt", fortæller Svend Funder, der var leder af ekspeditionen. Det drivtømmer, man fandt rester af, var fra starten af rejsen indlejret i havis, og det nåede derfor den nordlige Grønlands kyst sammen med havisen. Mængden af drivtømmer udgør derfor et mål for, hvor meget havis, der var i havet dengang. Det er netop den type is, der i dag er i fare for at forsvinde. Efter at ekspeditionerne var afsluttet, daterede man drivtømmeret ved kulstof-14 metoden. Kulstof-14 dateringen viser, at i den varme periode fra ca. 8000 år siden og indtil for 4000 år siden, var der mere åbent vand end i dag. Træet til drivtømmeret opstod nær store floder i Nordamerika og Sibirien. Trætypen var næsten udelukkende gran, som er udbredt i Nordamerika, samt lærk, som dominerer i den sibiriske taiga. De forskellige træsorter viser forandringer i rejseruterne som følge af ændrede strømforhold og vindforhold i havet. Man undersøgte også strandvolde langs kysten. I dag forhindrer flerårig havis enhver form for stranddannelse langs kysterne i det nordlige Grønland. Men dette har ikke altid været tilfældet. Man kortlagde strandvoldene på et 500 kilometer langt stykke langs kysten. Bag den nuværende kyst ses lange rækker af strandvolde. Det viser, at der på et tidspunkt var bølger, som kunne bryde op på stranden - altså uhindret af havis. Undersøgelserne viser altså, at der er store naturlige variationer i mængden af arktisk havis i perioden. Den dårlige nyhed er, at der er en klar sammenhæng mellem temperaturen og havis-mængden, så fortsat global opvarmning vil føre til en reduktion i mængden af sommerens arktiske havis. Den gode nyhed er, at selv med en reduktion til under 50% af den nuværende havis-mængde, vil havisen ikke nødvendigvis nå et "point of no return", hvor den ikke længere kan gendannes, hvis klimaet bliver køligere. Endelig understreger undersøgelserne, at ændringerne i høj grad er forårsaget af den virkning, som temperaturen har på de fremherskende vindsystemer. Dette har man tidligere ikke taget tilstrækkeligt hensyn til, når man har vurderet havisens tilbagegang. Udover at give en bedre forståelse af, hvordan klimaet i det nordlige Grønland var for tusinder af år siden, kan disse data også give et tip om, hvordan isbjørnene klarede sig i de varmere klimaperioder. Forskerholdet har planer om at bruge DNA i fossile isbjørneknogler til at studere isbjørnebestanden i løbet af Holocæns varmeste tid.
Test dig selv
Du kan tegne abonnement på BioNyt: Videnskabens verden her!
/DNA/Klima/Det korte svar er: Det signalerer tilstedeværelse af havis og transport fra det vestlige Arktis
Drivtømmer i Nordgrønland kan saves op til træ-identifikation og 14C-datering. Træet er blevet slidt under sin mindst 6 år lange rejse over havet, hvor den lå indlejret i havis. Et stykke drivtømmer-træ blev identificeret til sandsynligvis at have vokset for ca. 2000 år siden i Mackenzie eller Yukon ca. 5000 km væk. Dette signalerer tilstedeværelse af havis og transport fra det vestlige Arktis, som det også ses i dag, og denne rejserute blev udbredt for ca. 5000 år siden.
Test dig selv
Du kan tegne abonnement på BioNyt: Videnskabens verden her!
/DNA/Klima/Det korte svar er: 16.568 baseenheder i mange kopier i hver celle
Hver celle indeholder mange kopier (hundredevis) af små DNA-ringe (cirkulært DNA), der hos mennesket består af ca. 16.568 baseenheder. Denne del af arvemassen findes inde i de små celleorganeller, der kaldes mitokondrier, og som er ansvarlige for cellens energiforsyning.
Test dig selv
Du kan tegne abonnement på BioNyt: Videnskabens verden her!
/DNA/Mitokondrie/Det korte svar er: Fritlevende bakterier for 2,5 mia. år siden
Oprindeligt i evolutionen var mitokondrier fritlevende bakterier, men for ca. 2,5 milliarder år siden blev de optaget i “stamfaderen” til alle højerestående celler (eukaryote organismer). Mitokondrierne har altså delvis bevaret deres eget genom (mitokondrie- DNA, mtDNA), og hver mitokondrie indeholder 5-10 identiske kopier af mtDNA.
Test dig selv
Du kan tegne abonnement på BioNyt: Videnskabens verden her!
/DNA/Mitokondrie/Det korte svar er: F.eks. op til 5000
En celle med f.eks. 500 mitokondrier kan have 2500 - 5000 ens stykker af mtDNA. Dette store antal af disse mtDNA-kopier per celle er en del af forklaringen på, at det kan lade sig gøre at analysere på “fossilt” mtDNA.
Test dig selv
Du kan tegne abonnement på BioNyt: Videnskabens verden her!
/DNA/Mitokondrie/Det korte svar er: Via moderen
Moderens ægcelle indeholder tusinder af mitokondrier, der går i arv til børnenes celler. Sædcellens kerne befrugter ægcellen, men sædcelle- mitokondrie overføres ikke. mtDNA nedarves fra mor til datter og fra mor til søn, men kun datteren kan videreføre mtDNA-arven.
Test dig selv
Du kan tegne abonnement på BioNyt: Videnskabens verden her!
/DNA/Mitokondrie/Det korte svar er: 10 gange oftere end celle-DNA
Mitokondrie-DNA muterer oftere end kerne-DNA (generelt ca. 10 gange oftere). De hyppige mutationer gør mtDNA velegnet til at afklare slægtskabsforhold mellem meget nærtstående arter og populationer og til studiet af oprindelsen og udbredelsen af det moderne menneske, Homo sapiens.
Test dig selv
Du kan tegne abonnement på BioNyt: Videnskabens verden her!
/DNA/Mitokondrie/Det korte svar er: Via faderen
Medens mtDNA (mitokondrie-DNA) kun nedarves via moderen, bliver Y-kromosom-DNA kun nedarvet fra far-til-søn. Bortset fra evt. mutationer i Y-kromosomet nedarves det uforandret. Alle Y-kromosomer i nulevende mænd kan føres tilbage til en enkelt mand. Og alt mtDNA i nulevende mennesker kan føres tilbage til én kvinde. Y-kromosomet indeholder en meget stor sekvensvariation, der kan anvendes til slægtskabstest. Det bedst egnede genetiske system til at fastlægge oprindelsen og udbredelsen af Homo sapiens på Jorden, er Y-DNA, fordi de forskellige varianter er tæt knyttet til bestemte geografiske regioner.
Test dig selv
Du kan tegne abonnement på BioNyt: Videnskabens verden her!
/DNA/Mitokondrie/Det korte svar er: Der findes DNA i mitokondrier. (Samt i planters grønkornpartikler)
Ja, der findes DNA i mitokondrier, som er de organeller, som står for størstedelen af cellens ATP-syntese (Planter har DNA i deres grønkornpartikler i cellerne). Det DNA, som findes i mitokondrier, er cirkulært (ligesom hos bakterier), og mitokondrier udfører da også deres egen syntese af proteiner, men mitokondrierne har ikke alle de gener, der skal bruges at danne alle de typer af proteiner, som mitokondrierne skal bruge. Derfor får de også nogle proteiner, som dannes ud fra cellekernens DNA. Man kan sige, at cellen og dens mitokondrier lever i en form for symbiose. Mitokondriernes livscyklus er dog uafhængig af cellens cyklus.
Test dig selv
Du kan tegne abonnement på BioNyt: Videnskabens verden her!
/DNA/PCR/Sporing/Det korte svar er: Ved DNA-analyse ved hjælp af PCR-teknikken
PCR-reaktionen vil gøre det muligt at opspore uønskede organiske komponenter i vores fødevarer, ulovligt indførte arter, samt produkter fremstillet af disse arter. En DNA-stregkodeanalyse kan nu gennemføres på få timer. Den rå maskinpris (hvis der ikke kræves en vurdering af resultatet) er under 100 kr. DTU Aqua fik f.eks. en henvendelse fra et firma, som havde købt et parti fisk i udlandet. De følte sig usikker på, om det var den dyre fisk, de havde betalt for. Den DNA-analyse, som Einar Eg og Thomas Damm Als fra DTU Aqua foretog, viste ved sammenligning med et DNA-bibliotek over 8000 fiskearter, at det slet ikke var den fine fisk, som firmaet havde betalt for. Biokemiker Nicolai Z. Ballin fra Fødevareregion Øst har påvist hestekød i oksekødspegepølser og svinekød i hakket okse- og lammekød. Svar på sådanne spørgsmål tager ikke engang 1 døgn. Det var også DNA-stregkoden, som fældede en mand fra Malaysia, der forsøgte at forfalske naturmedicin. Han "fremstillede" en traditionel malaysisk urtemedicin, kaldet Tongkat Ali. Dette middel sælges som impotensmiddel og i mindre grad også som malariamiddel. I stedet for at bruge den rigtige plante, Eurycoma longifolia, behandlede manden et råmateriale, der var udvundet af gummitræer, med kinin for at efterligne den bitre smag hos Tongkat Ali. En kemiker fra Kuala Lumpur universitet, der har opbygget et DNA-stregkodebibliotek over 1200 malaysiske medicinplanter, påviste snyderiet. I Danmark arbejder Ole Seberg fra Statens Naturhistoriske Museum på at opbygge en DNA-stregkodebase over alle Danmarks planter sammen med Århus Universitet og Danmarks Miljøundersøgelser, i alt ca. 1400-1600 plantearter.
Test dig selv
Du kan tegne abonnement på BioNyt: Videnskabens verden her!
/DNA/PCR/Sporing/Det korte svar er: Ved sammenligning med kendte DNA-sekvenser
PCR-reaktionen vil gøre det muligt at opspore uønskede organiske komponenter i vores fødevarer, ulovligt indførte arter, samt produkter fremstillet af disse arter. En DNA-stregkodeanalyse kan nu gennemføres på få timer. Den rå maskinpris (hvis der ikke kræves en vurdering af resultatet) er under 100 kr. DTU Aqua fik f.eks. en henvendelse fra et firma, som havde købt et parti fisk i udlandet. De følte sig usikker på, om det var den dyre fisk, de havde betalt for. Den DNA-analyse, som Einar Eg og Thomas Damm Als fra DTU Aqua foretog, viste ved sammenligning med et DNA-bibliotek over 8000 fiskearter, at det slet ikke var den fine fisk, som firmaet havde betalt for. Biokemiker Nicolai Z. Ballin fra Fødevareregion Øst har påvist hestekød i oksekødspegepølser og svinekød i hakket okse- og lammekød. Svar på sådanne spørgsmål tager ikke engang 1 døgn. Det var også DNA-stregkoden, som fældede en mand fra Malaysia, der forsøgte at forfalske naturmedicin. Han "fremstillede" en traditionel malaysisk urtemedicin, kaldet Tongkat Ali. Dette middel sælges som impotensmiddel og i mindre grad også som malariamiddel. I stedet for at bruge den rigtige plante, Eurycoma longifolia, behandlede manden et råmateriale, der var udvundet af gummitræer, med kinin for at efterligne den bitre smag hos Tongkat Ali. En kemiker fra Kuala Lumpur universitet, der har opbygget et DNA-stregkodebibliotek over 1200 malaysiske medicinplanter, påviste snyderiet. I Danmark arbejder Ole Seberg fra Statens Naturhistoriske Museum på at opbygge en DNA-stregkodebase over alle Danmarks planter sammen med Århus Universitet og Danmarks Miljøundersøgelser, i alt ca. 1400-1600 plantearter.
Test dig selv
Du kan tegne abonnement på BioNyt: Videnskabens verden her!
/DNA/PCR/Sporing/Klinisk/Det korte svar er: Kræver ikke i samme grand dyrkning af bakterierne som traditionelle metoder
I Danmark er der hvert år ca. 250.000 indlæggelser, som skyldes en mikroorganisme (patogen), dvs. bakterie, svamp, virus eller parasit. Bakterier bestemmes ved mikroskopi og dyrkning på forskellige typer af næringsmedier (på agarplader eller i blodkolber) og ved forskellige biokemiske test til at skelne bakterierne fra hinanden.
Dyrkning viser samtidig, om bakterien er resistent overfor forskellige antibiotika. Der går dog mindst 1-2 døgn, før man har resultatet, og nogle mikroorganismer kan slet ikke dyrkes.
DNA-baserede metoder er væsentligt hurtigere. F.eks. kan bakterier identificeres på baggrund af deres 16S rRNA gen (ref.9542).
For at kunne identificere bakterie-DNA’et er det nødvendigt at opformere en lille del af dens genom. I en PCR (polymerase-kæde-reaktion) efterlignes den naturlige DNA-kopiering, og på få timer laves millioner af kopier af en udvalgt DNA-sekvens, idet man tilsætter små DNA-stykket, som kaldes “primere”. Primeren har en sekvens af DNA-byggesten (nukleotider), der modsvarer en del af den DNA-se-kvens, man gerne vil opformere. Med udgangspunkt i disse primere kan enzymet DNA-polymerase lave en kopi af den ønskede DNA-sekvens. (Se: "Hvordan virker PCR-reaktionen til bakteriebestemmelse?").
Test dig selv
Du kan tegne abonnement på BioNyt: Videnskabens verden her!
/DNA/PCR/Sporing/Klinisk/Det korte svar er: PCR-reaktionen forløber i tre trin, der gentages f.eks. 30-40 gange.
Bakterier kan identificeres ved PCR-reaktionen, der forløber i tre trin, der gentages f.eks. 30-40 gange. Selvom alle organismer har unikke genomer, er der ofte store ligheder mellem generne hos de forskellige arter, f.eks. hos genet 16S rDNA, der koder for en del af bakteriers ribosomer (ribosomer indgår i proteinsyntesen hos alle organismer). Genet består af ni variable områder, der er unikke for bakteriearten, og otte konserverede områder, som stort set er ens i næsten alle bakterier.
De variable og konserverede områder er fordelt imellem hinanden over hele 16S-genet. Ved at anvende primere, der er specifikke for to konserverede områder, kan man få et PCR-produkt, der både omfatter de konserverede og de mellemliggende variable dele af genet, og man kan derved opformere noget DNA fra dette gen fra mange forskellige bakterier på en gang. 16S genet anvendes derfor ofte til at identificere bakterier.
Da metoden er baseret på analyse af DNA fra mikroorganismens genom, er man ikke afhængig af, om bakterien kan dyrkes. Den direkte 16S-sekvensbestemmelse bruges til bredspektret PCR-analyse.
Metoden kan ikke umiddelbart bruges, hvis der er flere bakteriearter i den samme prøve. Men ved hjælp af såkaldte klonbiblioteker, hvor hvert produkt, der dannes i den bredspektrede PCR-proces, adskilles og analyseres for sig, kan man påvise flere forskellige bakteriearter i samme prøve. Fremstilling af klonbiblioteker er dog tidskrævende og anvendes især i forsknings-sammenhæng.
Den specifikke metode kan give et resultat i løbet af få timer. Man bruger et apparat, hvori prøven opvarmes til 95 °C for at adskille de to DNA-strenge i dobbelt-helix-strukturen. Derefter sænkes temperaturen til 55-60 °C, som får små DNA-stykker (primere) til at hæfte sig til DNA-strengen, idet de genkender deres komplementære se-kvens. Når temperaturen efterfølgende hæves til 72 °C fungerer primerne som startpunkt for enzymet DNA-polymerase, som genopbygger den komplementære DNA-streng (DNA-polymerase-enyzymet er specielt ved at kunne tåle denne høje temperatur). Processen gentages så f.eks. 30 gange.
De større sygehuse anvender i stigende omfang PCR til at identificere virus, svampe og svært dyrkbare bakterier, som f.eks. Mycobacterium tuberculosis, Mycoplasma pneumoniae og pneumocyster. På de klinisk mikrobiologiske afdelinger benyttes oftest specifik-PCR i stedet for den bredspektrede metode. Det vil sige, at der foretages en specifik PCR-analyse for hver af de mi-kroorganismer, som lægen mistænker som årsag til infektionen. En negativ PCR-analyse udelukker ikke nødvendigvis, at der er tale om en infektion, idet infektionen kan skyldes en anden organisme. Dette er den bredspektrede metode bedre til at afsløre. Bredspektret PCR med efterfølgende fremstilling af klonbiblioteker er dog meget tidskrævende og er derfor ikke egnet som et hurtigt diagnostisk værktøj.
En specifik PCR-analyse er især vel-egnet, hvis der må forventes en del normalflora, så patogenets DNA ”drukner” i mængden af DNA fra normalfloraen.
På Ålborg universitet har man undersøgt PCR-metoden på infektiøs endokardit, der er en alvorlig sygdom, som rammer ca. 300 danskere om året, og som skyldes, at mikroorganismer i blodet slår sig ned på indersiden af hjertet (endokardiet), og inficerer en eller flere af de fire hjerteklapper.
I svære tilfælde kan en ultralydsscan-ning af hjertet afsløre store biofilm-klumper af bakterier, der vokser på hjerteklapperne og medfører, at hjer-teklapperne ikke længere slutter tæt, så hjertet skal arbejde hårdere for at pumpe blodet rundt i kroppen. Hvis klappen bliver helt utæt, opstår hjertesvigt med en meget høj dødelighed.
Det er derfor vigtigt med hurtig identifikation. I mange tilfælde kan der ikke dyrkes bakterier frem, evt. fordi der forud er gået en bredspektret antibiotikabehandling, som hæmmer bakteriens fremvækst på mediet.
I et studie, hvor man inficerede 19 hjerteklapper, fandt man kun bakterier i 7-9 prøver ved traditionel identifikation, men PCR-metoderne kunne påvise DNA fra bakterier i alle 19 prøver, og med PCR fandt man desuden både de velkendte syndere (stafylokokker, streptokokker og enterokokker) og sjældne bakterier (Legionella, Pseudomonas og Propionibacterium).
Visse mikroorganismer kræver usædvanlige vækstforhold. Bakterier af Chlamydia-familien lever inden i celler, f.eks. epitelceller, og kræver derfor helt særlige cellekulturer at dyrkes på. Kighoste-bakterien Bordetella pertussis kan ikke vokse i standard-vækstmedier, og den vokser så langsomt, at kolonierne først kan ses med det blotte øje efter 4-5 dages vækst.
Desuden er bakterier, der vokser i biofilm, vanskelige at dyrke. Biofilm er en slimet substans af en blanding af flere bakterie- eller svampearter, og som indeholder både DNA, proteiner og polysakkarider, der er udskilt af mikroorganismerne. Biofilm ses på overflader, f.eks. ved kateter-infektioner og plak på tænderne.
Patienter, der får indlagt et central-vene-kateter i en central blodåre, hvor kateteret skal sidde i flere måneder og anvendes til at give medicin og væske direkte til blodet, har øget risiko for infektion.
Dyrkning af bakterier fra katetre foregår normalt ved en såkaldt Makis-metode, hvor spidsen af kateteret rulles på en agarplade. Indstik-stedet siges at være inficeret, hvis der fremkommer mindst 14 bakteriekolonier på pladen. Makis-metoden er imidlertid utilstrækkelig, idet bakterier længere oppe ad kateteret og på indersiden af kateteret kan blive overset.
Da PCR-metoden er baseret på påvisning af DNA, er metoden uafhængig af, om mikroorganismerne er i stand til at vokse. Desuden har PCR-meto-den vist, at der er stor forskel på hvilke bakterier, der findes på inder- og ydersiden af katetrene.
PCR-metodens høje følsomhed kan være en ulempe derved, at metoden ikke skelner mellem DNA fra levende og døde bakterier. Der findes dog i dag kit på markedet, der kan fjerne frit DNA fra en prøve, så der kun arbejdes videre med DNA fra intakte, aktive bakterier.
På sigt bliver det måske muligt at bruge PCR til at undersøge bakteriers resistensmønstre ved at målrette PCR mod resistensgener, men indtil videre er dette ikke muligt. PCR er altså kun et supplement: Det er stadig nødvendigt at dyrke bakterierne for at påvise, hvilke antibiotika de er resistente overfor.
Test dig selv
Du kan tegne abonnement på BioNyt: Videnskabens verden her!
DNA/RNA/Det korte svar er: Først bindes 16S rRNA til en målsekvens, hvorefter initiator-tRNA bindes til en AUG-sekvens.
Proteinsyntesen i bakterier starter altid med en AUG, som koder for en fMet (N-formylmethionin). Opstrøms for AUG-koden findes en såkaldt Shine-Dalgarno sekvens, som er en region, der består af mange puriner (G eller A). Det første, der sker i translationen, er, at 16S rRNA binder til denne Shine-Dalgarno sekvens, hvorefter der kommer en initiator-tRNA, som binder til AUG-sekvensen. Da 16S rRNA er en bestanddel af den lille ribosomale enhed, har vi den på plads. Nu mangler vi bare den store ribosomale 50S til at sætte sig på. Til at hjælpe med at bringe den store ribosomale enhed på, har vi en fMet-tRNA, som kan danne bro mellem 30S og 50S, og derved bliver der dannet tre steder, hvor nye tRNA-molekyler kan binde. Disse kaldes A-site (aminoacyl), P-site (peptidyl) og E-site (exit).
Test dig selv
Du kan tegne abonnement på BioNyt: Videnskabens verden her!
DNA/RNA/Det korte svar er: Alternativ splejsning sker under udarbejdelsen af det færdige mRNA, idet det færdige mRNA bliver sammensat af forskellige exons der kan medføre forskellige proteinprodukter
Alternativ splejsning sker under udarbejdelsen af det færdige mRNA. Det færdige mRNA bliver sammensat af forskellige exons, og de forskellige exons kan sættes sammen på mange forskellige måder. På figuren ses nogle af de alternative måder, man kan sammensætte det færdige mRNA på. De farvede områder forestiller forskellige exons og til højre i figuren ses de forskellige kombinationsmuligheder.
Dette kan medføre anderledes proteiner, og denne variation er vigtig i immunsystemet. Se i øvrigt spørgsmålet om gener i immunsystemet.
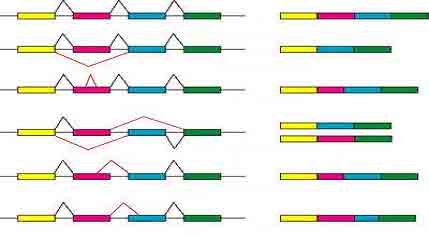
Test dig selv
Du kan tegne abonnement på BioNyt: Videnskabens verden her!
DNA/RNA/Det korte svar er: Genaktivitet kan f.eks. vises ved "in situ hybridisering" der angiver mængden af mRNA i cellen
Påvisning af genaktivitet kan gøres på flere forskellige måder. Den mest effektive i dag er en ikke-radioaktiv "in situ hybridisering" (en metode til bestemmelse af mængden af mRNA i cellen). Et forskerteam på Max-Planck Instituttet i Hannover i Tyskland har kortlagt gen-udtryksmønstret hos musefostre. Deres resultater kan ses på hjemmesiden www.genepaint.org, hvor det er muligt at se billeder af de fleste fosterstadier hos mus, samt hvor og hvornår de forskellige gener er udtrykt.
Test dig selv
Du kan tegne abonnement på BioNyt: Videnskabens verden her!
DNA/RNA/Det korte svar er: Et fusionsgen er et hybridt gen, som er sat sammen af to eller flere stumper af forskellige gener
Et fusionsgen er et hybridt gen, som er sat sammen af to eller flere stumper af forskellige gener for at producere et nyt protein. Et hybridt gen kan også være at sætte et gen på en bestemt promotor for at gener, som styres af promotoren, kan blive udtrykt i øget omfang eller i formindsket omfang.
Test dig selv
Du kan tegne abonnement på BioNyt: Videnskabens verden her!
DNA/RNA/ Det korte svar er: DNA-bindende proteiner kan føre RNA-polymerase-2 hen til det sted, som skal transkriberes
Under indvirkning af en kaskade af DNA-bindende proteiner kan RNA-polymerase II (RNA-polymerase-2) guides hen til det sted, som skal transkriberes. En række af DNA-bindende proteiner binder sig til DNA-strengen. Disse proteiner kaldes TFII (TF står for transkriptionsfaktor og II refererer til RNA-polymerase II). De forskellige TFII-proteiner hedder henholdsvis TFIIA, -B, -D, -E og -F. På figuren ses i hvilken rækkefølge, de binder til DNA.
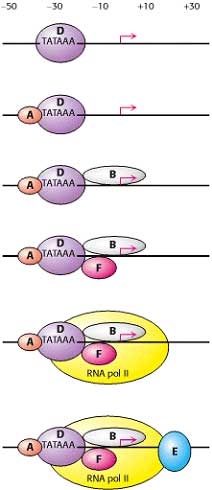
Læg mærke til, at det første protein, som binder til DNA´et, er protein TFIID, som binder til TATA-boxen. Denne box findes ca. 25-30 baser opstrøms for initieringsstedet (se pilen på figuren). Funktionen af de forskellige proteiner vil vi ikke komme ind på her, men de er alle nødvendige for en korrekt transkription.
Endvidere kan enhancere også promovere transkriptionen, altså fremskynde den. Man ved langt fra alt om dette, men man ved dog, at disse enhancer-sekvenser kan befinde sig tusindvis af baser væk fra det gen, som de promoverer. Endvidere har man fundet ud af, at de kan befinde sig før (opstrøms), efter (nedenstrøms) eller endda midt i genet! (kilde: L. Stryer: Biochemistry 5. udg. s. 797)
Test dig selv
Du kan tegne abonnement på BioNyt: Videnskabens verden her!
DNA/RNA/ Det korte svar er: Small nuclear RNA, der indgår i transskriptionen
Small nuclear RNA (snRNA) er korte RNA-stykker på 100-300 baser, som associerer med proteiner for at danne små nukleare ribonukleoprotein-partikler (snRNPs). Disse bruges i transskriptionen.
Test dig selv
Du kan tegne abonnement på BioNyt: Videnskabens verden her!
DNA/RNA/ Det korte svar er: Der er bl.a. forskelle i opbygningen, forekomsten og funktionen
RNA og DNA er opbygget på samme måde, men hvor RNA indeholder ribose er der deoxyribose i DNA (deraf navnene). RNA indeholder ikke thymin-basen, men derimod uracil i stedet. Desuden er RNA ofte enkeltstenget, medens DNA typisk er dobbeltstrenget (to anti-parallelle strenge). Meget RNA kan danne dobbeltbindinger til dele af sig selv, men det ligner ikke derved DNA-dobbeltstrengen. DNA findes i cellekernen (og i mitokondrierne), medens meget RNA findes i cytoplasmaet. RNA kan let trænge ud og ind gennem cellekernemembranen. Næsten alt DNA er pakket sammen med proteiner i de såkaldte kromosomer. RNA kan oversættes til protein, medens DNA ikke direkte kan oversættes til protein. I livets udvikling kom RNA først, medens DNA først udvikledes senere. Det betyder, at enzymet revers transskriptase (der ud fra RNA kan danne DNA) må have været et af de første enzymer i livets udvikling. En sådan revers transskriptase findes i Mauriceville-plasmidet i mitokondrierne hos Neurospora crassa, som er et svampedyr (tidligere kaldt “slimsvampe”). I modsætning til andre revers-transskriptase-enzymer behøver netop dette revers-transsskriptase-enzym ikke en nukleinsyreprimer for at blive aktiv. Det kunne tyde på, at netop dette enzym er særlig primitivt og beslægtet med det første revers-transskriptase-enzym, som livet udviklede. (9785s.9-10-11)
Test dig selv
Du kan tegne abonnement på BioNyt: Videnskabens verden her!
DNA/RNA/iRNADet korte svar er: Man mener, at man med interferens-RNA (iRNA og siRNA) vil kunne bremse ethvert gen til under 1/4 af dets normale aktivitet ved derved at blokere for det mRNA, der normalt dannes fra genet.
RNA-interferens (iRNA) virker ved at igangsætte et angreb på det meddeler-RNA (mRNA), som er blevet dannet ud fra koden i et gen, og mRNA er skabelon for et proteins dannelse. RNA-interferens virker derved som små, smarte bomber, der lukker for gener. Man kan lave små RNA-molekyler, "small interference RNA" (siRNA), som matcher en del af sekvensen af mål-genet. Disse små RNA-molekyler forekommer også naturligt. De udgør en del af det forsvarssystem, som celler bruger til at ødelægge virus og til at styre geners aktivitet.
iRNA er en gave til genforskere, for ved at stoppe et gens aktivitet kan man lære dets funktion at kende. Man arbejder også på at bruge metoden som behandling mod visse sygdomme. Umiddelbart burde alle siRNA-molekyler virke, men i praksis kan nogle kun reducere genets aktivitet til det halve, og for nogle gener er det vanskeligt at finde effektive siRNA'er. Nogle sekvenser synes at være helt ineffektive. Ved at undgå disse sekvenser, og ved samtidig at benytte flere siRNA-molekyler mod samme gen, har man dog kunne forøge effektiviteten dramatisk. Man mener at man derved vil kunne bremse ethvert gen til under 1/4 af dets normale aktivitet.
Test dig selv
Du kan tegne abonnement på BioNyt: Videnskabens verden her!
DNA/RNA/mRNA/Det korte svar er: Deres struktur og funktion
mRNA (meddeler-RNA [engelsk: messenger RNA]): er den kopi af DNA´et, som bliver aflæst af tRNA i ribosomet.
tRNA (transport-RNA [engelsk: transfer RNA]): er den RNA-type, som bringer de frie aminosyrer til ribosomet. Man kan generelt sige, at tRNA er den eneste RNA-type, som kender den genetiske kode (se s. 25 i BioNyt nr. 132), da den aflæser mRNA-strengen med sit ”anti-codon”. Når aminosyren er påsat en tRNA, kaldes den en ”aminoacyl-tRNA”.
rRNA (ribosomalt RNA): er det RNA, som findes i ribosomer. Omkring to trediedele af ribosomet består af rRNA, og dette er nødvendigt for at opretholde den specielle rumlige struktur, som ribosomerne har.
(kilde: L. Stryer et.al. Biochemistry, 2003, 5. udg.)
Test dig selv
Du kan tegne abonnement på BioNyt: Videnskabens verden her!
/DNA/RNA/Protein/Det korte svar er: Det forhold, at genetisk information generelt foregår i retningen DNA-> RNA ->Protein
Det "centrale dogme" er Francis og Crick´s koncept med, at genetisk information generelt er foregået i retningen DNA-> RNA ->Protein.
I dag er der vedtaget en lidt revideret udgave, efter at man har fundet enzymet "revers transkriptase", som kan lave RNA om til DNA:
DNA <-> RNA -> Protein
Test dig selv
Du kan tegne abonnement på BioNyt: Videnskabens verden her!
/DNA/RNA/Protein/Det korte svar er: Proteinsyntesen i bakterier starter altid med en AUG, som koder for en methioninforbindelse
Proteinsyntesen i bakterier starter altid med en AUG, som koder for en fMet (N-formylmethionin). Opstrøms for AUG-koden findes en såkaldt Shine-Dalgarno sekvens, som er en region, der består af mange puriner (G eller A). Det første, der sker i translationen, er, at 16S rRNA binder til denne Shine-Dalgarno sekvens, hvorefter der kommer en initiator-tRNA, som binder til AUG-sekvensen. Da 16S rRNA er en bestanddel af den lille ribosomale enhed, har vi den på plads. Nu mangler vi bare den store ribosomale 50S til at sætte sig på. Til at hjælpe med at bringe den store ribosomale enhed på, har vi en fMet-tRNA, som kan danne bro mellem 30S og 50S, og derved bliver der dannet tre steder, hvor nye tRNA-molekyler kan binde. Disse kaldes A-site (aminoacyl), P-site (peptidyl) og E-site (exit).
Test dig selv
Du kan tegne abonnement på BioNyt: Videnskabens verden her!
/DNA/RNA/Protein/Det korte svar er: Indtil der er for få A-molekyler i halen
Det er en kendsgerning, at DNA er mere stabilt end RNA, men levetiden for RNA er ikke afgørende for, hvor mange protein-molekyler der kan dannes af et stykke RNA. Cellen har de fleste processer under kontrol. Derfor er det også afgørende, at den kan styre mængden af det protein, som skal syntetiseres. Styringen af syntesen sker primært med antallet af adenin-molekyler, som tilføjes af poly(A) polymerasen. For hver ribosom, som har aflæst mRNA´et, bliver der fraspaltet nogle A-molekyler. Dvs. at poly(A) halen bliver kortere og kortere, og til sidst, når der er meget få A-molekyler i halen, vil mRNA´et blive opløst, og translationen vil stoppe.
Test dig selv
Du kan tegne abonnement på BioNyt: Videnskabens verden her!
/DNA/RNA/Protein/Det korte svar er: Kroppen har over 200 forskellige celler
Det er naturligvis ikke alle gener, som er aktive i alle celler. Kroppen har over 200 forskellige celler, som alle har forskellige funktioner, og dermed også forskellige proteiner. Der findes dog de såkaldte ”husholdningsgener”, som er gener, som er mere eller mindre aktive i alle celler. Som eksempler på disse kan vi nævne polymerase-gener, gener som koder for proteiner i ribosomer, gener som koder for proteiner i plasma-membranen osv. Herunder ses en tabel over forskellige proteiners tilstedeværelse i fem forskellige celletyper:
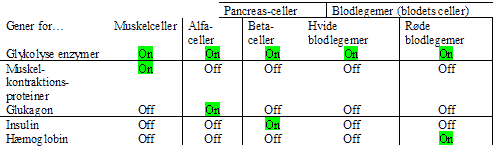 (kilde: N.A. Campbell:”Biology – concepts & connections” 4. udg. 2003)
(kilde: N.A. Campbell:”Biology – concepts & connections” 4. udg. 2003)
Test dig selv
Du kan tegne abonnement på BioNyt: Videnskabens verden her!
/DNA/RNA/Protein/Det korte svar er: At DNA-sekvenser oversættes til RNA-sekvenser og dette videre oversættes til proteiners aminosyre-sekvenser
Dette er betegnelsen for det længe opretholdte dogme, at DNA altid oversættes til RNA, som altid oversættes til protein. Fundet af enzymtypen revers-transskriptase (der kan lave DNA ud fra RNA) kuldkastede dette dogmes universelle sandhed. Retrotransposoner (en type af mobilt DNA) danner revers-transskriptase. (9785s.9)
Test dig selv
Du kan tegne abonnement på BioNyt: Videnskabens verden her!
/DNA/RNA/Protein/Åben_læseramme/Det korte svar er: Åben læseramme i et gen
ORF kan være en forkortelse for “Open Reading Frame”. Det henviser til at cellens proteinsyntetiserende maskineri kan syntetisere et protein ved at afkode den information, som findes i en sådan “åben læseramme”. (9785s.55)
Test dig selv
Du kan tegne abonnement på BioNyt: Videnskabens verden her!
/DNA/RNA/Protein/Åben_læseramme/Det korte svar er: Det henviser til at cellens proteinsyntetiserende maskineri kan syntetisere et protein ved at afkode informationen her
ORF kan være en forkortelse for “Open Reading Frame”. Det henviser til at cellens proteinsyntetiserende maskineri kan syntetisere et protein ved at afkode informationen som findes i en sådan “åben læseramme”. (9785s.55)
Test dig selv
Du kan tegne abonnement på BioNyt: Videnskabens verden her!
/DNA/RNA/Revers_transkriptase/Det korte svar er: Enzymer, som kan oversættes RNA-sekvenser (tilbage) til DNA-sekvenser
Det er et enzym, som er en RNA-afhængig DNA-polymerase (altså i stand til at lave DNA ud fra en RNA-kode). (9785s.9)
Test dig selv
Du kan tegne abonnement på BioNyt: Videnskabens verden her!
/DNA/RNA/Revers_transkriptase/Det korte svar er: David Baltimore og Howard Temin
Revers transskriptase blev opdaget af David Baltimore og Howard Temin, som fik Nobelprisen herfor i 1975. (9785s.9)
Test dig selv
Du kan tegne abonnement på BioNyt: Videnskabens verden her!
/DNA/RNA/Revers_transkriptase/Det korte svar er: Det er en type retro-transposoner, som stadig har bevaret en kappe, så de kan inficere celler
Retrovirus er virus, som formentlig udvikledes fra LTR-retrotransposoner (en type mobilt DNA med copy/paste-funktion). Retrovirus adskiller sig blot ved at have sekvenser, som koder for en beskyttende kappe (envelope), som giver virusset mulighed for at inficere nye celler. Da LTR-retrotransposoner mangler en beskyttende kappe er de ikke i stand til at trænge ud igennem cellemembranen, og er altså fanget inde i cellen. (9785s.11)
Test dig selv
Du kan tegne abonnement på BioNyt: Videnskabens verden her!
DNA/RNA/rRNADet korte svar er: Sted hvor proteinsyntesen sker i cellen
Som beskrevet ovenfor består ca. to-tredjedele af alt ribosom af rRNA (ribosomalt RNA). Resten er forskellige proteiner. Ribosomet består af to forskellige enheder, hos bakterier dels en lille 30S enhed og dels en stor 50S enhed (S står for Svedberg-units, som mål for kompleksets vandring i et centrifugalt felt). Disse to enheder vil tilsammen udgøre ribosomet (som er 70S, da Svedberg-enheder ikke bare sådan lige kan adderes). 30S-enheden består af 21 forskellige proteiner (disse kaldes S1, S2, …, S21) og et 16S rRNA-molekyle. 50S-enheden består af 34 forskellige proteiner (disse kaldes L1, L2,…,L34) og to rRNA-molekyler er henholdsvis et 23S-RNA og et 5S-RNA. En af de afgørende faktorer, som gør rRNA i stand til at være med i translationen, er, at rRNA består af små korte duplex-regioner, dvs. RNA´et antager en dobbelt-strenget form, som normalt ikke ses på f.eks. mRNA.
Test dig selv
Du kan tegne abonnement på BioNyt: Videnskabens verden her!
/DNA/Sekvens/Det korte svar er: Ved at mærke det pågældende DNA med en stregkode-sekvens til kontrol af, at der ikke senere kommes ny DNA (uden stregkoden) til under laboratoriearbejdet
For at beskytte mod forurening kan man mærke hvert fragment af det udvundne DNA med en "stregkode" (dvs. en sekvens af baser, der påsættes DNA-fragmenterne, medens disse stadig er i renrums-laboratoriet, så de senere under sekvensbestemmelsen kan skelnes fra eventuelt omstrejfende DNA fra moderne mennesker, der jo ikke vil have denne stregkode-sekvensstart på fragmenterne).
Test dig selv
Du kan tegne abonnement på BioNyt: Videnskabens verden her!
/DNA/Sekvens/Det korte svar er: Fordi DNA'et er beskyttet af proteiner i håret
Hår er lavet af keratin, og ikke porøst. Det er som plast på oversiden af et rør, der indeholder rester af døde celler. Der er masser af bakterier, men de sidder alle på overfladen af håret, og er aldrig trængt dybt ind. "Det var så der, hvor vi i virkeligheden gik ned og købte noget lokumsrens, klorin, i Netto", fortæller Eske Willerslev, "Så dyppede vi noget mammuthår ned i det, skyllede klorinen bort med vand, og ekstraherede og bingo! 90% af alt det DNA, der kom ud, var mammut-DNA". Klorin nedbryder rigtig meget organisk materiale. Det vil sige, at alt, som ligger uden på håret, forsvinder. Men klorin nedbryder ikke selve det keratin, som hårvævet er lavet af. Hår er rørformet, og er kun levende i roden. Døde celler bliver skudt ud, efterhånden som håret vokser, men de bliver lukket inde i keratin-skaftet. For at få fat i DNA'et bruger man et enzym, som nedbryder alle proteiner - herunder keratin, som også er et protein. Håret på et levende menneske har en vis fragmentering af DNA’et. Fragmenteringen opstår, når håret dannes. Men derefter sker der kun langsomt fragmentering af DNA’et, især langsomt hvis håret ligger i permafrost.
Test dig selv
Du kan tegne abonnement på BioNyt: Videnskabens verden her!
/DNA/Sekvens/Det korte svar er: Parallelsekventeringsmetoder udviklet siden omkr. 2005
I dag drømmer man gerne om at finde DNA fra f.eks. et 500.000 år gammelt fortidsmenneske. Sådanne "drømme" skyldes den tekniske revolution siden ca. 2005 inden for DNA-sekvensbestemmelse. Fra 1977 til 2005 brugte man stort set samme metode til at bestemme DNA-sekvenser på. I 1977 kunne man kikke på ét fragment af DNA af gangen, og i 2005 kunne man kikke på 400 af gangen, så det gik hurtigere efterhånden, men der var tale om en slags langsom, liniær vækst. Men så kom en meget innovativ amerikaner, Jonathan Rothberg, på en idé, som førte til, at GS20-teknikken kom på markedet ca. 2005. Det var en revolution inden for sekvensbestemmelse, fordi man nu pludselig kunne se - parallelt - på 100.000 småstumper af DNA af gangen, hvor hvert DNA-fragment var fra ca. 20 til ca. 100 baser langt. I dag har man en teknik, som hedder GS-FLX-titanium- plus, som kan se fragmenter, som er 1000 baser lange, og man kan se på 1 million fragmenter af gangen - specielt mange hos det konkurrerende firma Illumina (som opkøbte det engelske firma Solexa) . Med teknikken i 2005 var man også nået op på fragmentlængder på 1000 baser, men stadig kunne man kun se på 400 fragmenter af gangen.
Der er nu kommet konkurrence på sekvensmaskine-området. På Center for Geogenetik har de en maskine, som i én kørsel kan se på 2 milliarder DNA-fragmentstykker på hver 200 baser. Så maskinen kan altså kikke på op til 400 milliarder baser i én kørsel. (En kørsel afsluttes inden for en dag).
Udviklingen i de senere år har altså været i eksponentiel vækst. Denne high throughput teknologi er som sagt en revolution: Man tager et tilfældigt DNA-stykke, og sætter et kendt håndtag (såkaldte tags) i hver ende, så man kan lave PCR-opformering på DNA-stykket. (Man kan endnu ikke lave en sekvensbestemmelse af ét DNA-molekyle - der kræves mange tusinde DNA-molekyler) . Udfordringen for at kunne skabe parallel-sekvensbestemmelser var for det første at finde en smart metode til at klistre håndtagene på, og dels at finde på en måde, hvor man kunne opformere DNA'et inden for et bestemt lille begrænset område, såsom f.eks. en kugleoverflade. Se spørgsmålet: "Hvordan laver man hurtig DNA-sekvensbestemmelse?"
Test dig selv
Du kan tegne abonnement på BioNyt: Videnskabens verden her!
/DNA/Sekvens/Det korte svar er: Ved forskellige metoder, der bruger parallelle sekventeringer
Der er i dag især tre måder at lave sekvensbestemmelsen på. GS-serien bruger kugler påsat med den ene primer (der er modpart til det ene af de to påsatte håndtag - tags - på DNAstykkerne) , sådan at kuglen bliver helt dækket med kopier af det samme DNA-stykke. Derefter kan man læse DNA-stykkets basesekvens ved at sørge for, at kuglen lyser op, når en base sætter sig. Lad os f.eks. antage, at den første base, der kan sætte sig på de mange ens DNA-stykker, er C. Hvis man først smider A-baser hen over pladen, vil pladen ikke lyse op. Men hvis man i vores eksempel derefter forsøger med C-baser, vil kuglen med de mange ens DNA-fragmenter lyse op, fordi man har designet det sådan, at lys udsendes, når DNA-strengen forlænges (lysstyrken angiver, om der evt. påsættes flere ens baser i række) . Denne første base lader man blive siddende og går derefter videre med at teste næste base-plads i DNA-fragmentet. På den måde kan man sammenstykke hele sekvensen af DNA-fragmentet. I virkeligheden har man en plade, hvori der er små brønde. På hver plade (der er under 10 cm lang) er der 3,4 millioner brønde. Hver brønd er under 30 mikrometer stor. Der ligger én kugle i hver brønd. Brøndene er nemlig så små, at der kun er plads til én kugle. Der må ikke komme to kugler i en brønd, for så ville man få to blandede lyssignaler. Hver kugle bliver i brønden dækket af ens DNA-stykker (dannet ved PCR-opformeringsteknik) . De forskellige kugler er belagt med forskellige DNA-fragmenter, som altså kan sekvensbestemmes parallelt. I en anden metode (den britiske) sidder primerne på en glasplade. DNA'et sætter sig fast på primerne og forlænger dem. Den anden primer sidder også fast på glaspladen. DNA'et, der altså sidder fast i den ene ende, bukker sig som en tangplante, indtil det får fat i den anden primer. DNA-strengene opformeres derefter med PCR-teknik, hvorved man får en lille skov af ens DNA-træer. De gøres oprette og enkeltstrengede igen, og bruges som skabelon for basepåsætning. I denne metode tilsættes alle fire baser samtidig, og når en af de fire basetyper finder sin plads, afgives lys, som ud fra farven af lyset fortæller, hvilken base, der satte sig på stedet. Man kan have rigtig mange af disse små skove af DNA-molekyler på pladen. Det er med denne teknik, at man kan have 2 milliarder på en glasplade, der er endnu mindre end pladen i den første (amerikanske) teknik. Disse 2 milliarder sekvensstumper kan sekvensbestemmes parallelt og altså samtidigt. Det er det, der ligger i betegnelsen "high throughput". Så i stedet for at snakke om 400, som man kan se med øjnene - at dér er en brønd og dér er en anden brønd - så er der nu tale om noget, hvor man slet ikke kan se det, men hvor man bruger en laser til at definere, hvor på pladen noget lyser op. Så der er tale om en masse optik og præcision i disse nye metoder.
Senest har det amerikanske bioteknologifirma Life Technologies udviklet en tredie metode. Det er en maskine, der også er baseret på en opfindelse af Jonathan Rothberg. Det nye apparat kan fastlægge hele det humane genom på mindre end én dag (9652note2) . Prisen på apparatet er forholdsvis lav, nemlig 870.000 kr. Det skal især bruges til humangenom-test, hvor prisen pr. test bliver omkring 1000 US-dollar, ca. 5-6000 kr. Apparatet er udviklet af firmaet Ion Torrent, der er en del af den amerikanske bioteknologivirksomhed Life Technologies (ref.9652note3) . Apparatet er på størrelse med en laserprinter. Det er baseret på halvlederteknologi - og er altså ikke som andre gensekventeringsmaskiner baseret på optisk læsning af data med anvendelse af komplicerede kameraer og lasere. Anvendelsen af halvledere i stedet for optisk læsning viste sin styrke, da apparatet blev anvendt ved opklaring af E.coli-udbruddet i Tyskland i foråret 2011. Den toksiske stamme blev sekventeret på et par timer (ref.9652note4) . Opfinderen, Jonathan M. Rothberg, og grundlægger og administrerende direktør for firmaet Ion Torrent, er stolt over denne udvikling, hvor det i to trin er lykkedes at opskalere processen 10.000 gange (100 gange med det første apparat baseret på halvlederprincippet (i apparatet "Ion PGM") og 100 gange med det nye apparat, kaldet "Ion Proton" (ref.9652note4) . Rækkefølgen af DNA-baserne i det DNA, der skal analyseres, fastlægges ved at syntetisere (opbygge) den komplementære DNA-streng. Det DNA, der skal analyseres, tjener som skabelon (template) . Under dannelse af en kopi af skabelon-DNA'et frigøres en hydrogen-ion hver gang, der kobles en base på. Hvis der kobles to identiske DNA-baser på, frigøres 2 hydrogen-ioner og signalet vil blive dobbelt så stort (ref.9652note5) . En mikrobrønd, der indeholder en skabelonstreng, hvis sekvens skal bestemmes, tilføres én af de fire mulige DNA-baser. Hvis denne base er komplementær til den base, der ifølge skabelonen skal sættes på, bliver basen koblet på, og der frigøres en hydrogen-ion. Hydrogen- ionen ændrer pH-værdien af opløsningen, hvilket kan opfanges af en meget følsom ionsensor (pH-meter) som tegn på, at der er sket en reaktion. DNA-sekventeringsapparatet er nok verdens mindste pH-meter. Det omformer kemisk information til digital information. Hvis DNA-basen ikke kobles på, skylles DNA-basen ud og de tre andre DNA-baser afprøves en af gangen. Firmaet mener, at det dermed bliver nemmere at tilpasse en medicinsk behandling til den enkelte patient - specielt kræftpatienter, der derved kan få en behandling, som passer til deres genprofil. Desuden bliver det nemmere at forske i sammenhængen mellem gener og sygdomme. Jonathan Rothberg vurderer, at 5000-10.000 mennesker i dag har fået sekventeret deres genom, men at tallet kan stige til millioner i løbet af de næste 10 år (ref.9585).
Test dig selv
Du kan tegne abonnement på BioNyt: Videnskabens verden her!
/DNA/Sekvens/Det korte svar er: Projektet at kortlægge hele menneskets genom
Da man fik ideen om at kortlægge hele menneskets genom ("Humangenomprojektet"), så opgaven næsten håbløs omfattende ud. Men i 2000 havde man i form af en råskitse kortlagt stort set alle 3,2 milliarder basepar, som udgør bogstaverne i den genetiske opskrift på et menneske. Den genetiske kode består kun af 4 bogstaver: A, T, C og G, der er navne på kemiske "baser". Deres rækkefølge (... TCGCTAATCGTAGCCAATGA... osv.) afgør arten - og i samspil med miljøet individets egenskaber og sygdomme. Det blev til sidst en dyst mellem HUGO (HumanGenom-Projektet), der var betalt af det offentlige, og det private firma Celera Genomics med molekylærbiologen Craig Venter i spidsen. Inden for HUGO klonede man korte stykker af DNA på ca. 150.000 basepar ind i kunstige kromosomer. Hver klon blev så kortlagt stykke for stykke. Celera's metode var hurtigere. De begyndte deres humangenom-sekventering, da HUGO-projektet var halvvejs i det planlagte tidsforløb, men stadig kun havde kortlagt 3%. Firmaet valgte at hakke hele arvematerialet op i småstykker, som fik forskellig længde - men ud fra de overlappende sekvenser kunne alle småbidderne lægges i korrekt rækkefølge. Firmaet nåede deres mål fem gange hurtigere end det offentlige projekt. Men da HUGO, som var startet meget tidligere, satte alt ind på at blive først, nåede de dette nogle få måneder før firmaet. Især drevet af frygten for, at firmaet ville udtage pa patent på sekvenserne og dermed hæmme anvendelsen af den nye viden. Menneskets DNA-kode blev i løbet af 15 år kortlagt af 1100 forskere fra 16 laboratorier i 6 lande - 5 år før forventet (ref.9570).
Allerede 10 år efter, at det for milliarder kroner lykkedes at sekvensbestemme det første genom fra et nulevende menneske, var teknikkerne blevet så forbedret, at man kunne sekvensbestemme ganske små mængder af fragmenteret og ødelagt DNA - såsom fra en 4000 år gammelt hårtot. Det er forklaringen på, at Eske Willerslevs gruppe fra Københavns Universitet kunne sekvensbestemme et 4000 år gammelt genom - altså de ca. 3 milliarder af DNA-baser.
Denne nye DNA-sekventeringsteknologi kaldes “next generation high throughput sequencing”, og har fuldstændig revolutioneret molekylærbiologien og i særdeleshed studierne af fossilt DNA. Dels kan man læse rigtig mange "bogstaver" på én gang, og dels kan man finde meget lidt DNA, endog fragmenteret ned til 50-60 baser. Så små stykker kunne man tidligere ikke fange - og man ville konkludere, at der ikke var noget DNA tilbage i prøven.
Test dig selv
Du kan tegne abonnement på BioNyt: Videnskabens verden her!
/DNA/Sekvens/Det korte svar er: DNA fra forskellige kilder er svære eller umulige at skelne fra hinanden
DNA-sekventeringsteknikken kan ikke skelne mellem menneske-DNA og bakterie-DNA, men læser det, der er mest af. Da man for første gang forsøgte sig på gammelt materiale, bl.a. en mammutknogle med intakt marv, var 50% af de påviste sekvenser bakterie- DNA. Knogler er porøse, så de bliver bakteriefyldt. I Neandertal-knogler var 99% af DNA'et fra bakterier. Kun 1% var enten Neandertal-DNA eller arkæologernes eget DNA.
Test dig selv
Du kan tegne abonnement på BioNyt: Videnskabens verden her!
/DNA/Sekvens/Det korte svar er: Til fjernelse af bakterie-DNA og andet, forurenende DNA på hår før sekventering
Der er masser af bakterier, men de sidder alle på overfladen af hår, og er aldrig trængt dybt ind. En ph.d.-studerende på Eske Willerslevs afdeling kom på den idé, at man skulle bruge hår i stedet for knogler, hvis man kunne rense håret for bakterier. Det var Tom Gilbert, der nu er blevet professor, der ii virkeligheden gik ned i Netto og købte noget lokumsrens, klorin ", fortæller Eske Willerslev, "Så dyppede vi noget mammuthår ned i det, skyllede klorinen bort med vand, og ekstraherede og bingo! 90% af alt det DNA, der kom ud, var mammut-DNA". Klorin nedbryder rigtig meget organisk materiale. Det vil sige, at alt, som ligger uden på håret, forsvinder. Men klorin nedbryder ikke selve det keratin, som hårvævet er lavet af.
Test dig selv
Du kan tegne abonnement på BioNyt: Videnskabens verden her!
/DNA/Sekvens/Det korte svar er: Cellerne inde i hårskaftet er ikke forurenede med fremmed DNA
Hår er lavet af keratin, og ikke porøst. Det er som plast på oversiden af et rør, der indeholder rester af døde celler. Der er masser af bakterier, men de sidder alle på overfladen af håret, og er aldrig trængt dybt ind. "Det var så der, hvor vi i virkeligheden gik ned og købte noget lokumsrens, klorin, i Netto", fortæller Eske Willerslev, "Så dyppede vi noget mammuthår ned i det, skyllede klorinen bort med vand, og ekstraherede og bingo! 90% af alt det DNA, der kom ud, var mammut-DNA". Klorin nedbryder rigtig meget organisk materiale. Det vil sige, at alt, som ligger uden på håret, forsvinder. Men klorin nedbryder ikke selve det keratin, som hårvævet er lavet af. Hår er rørformet, og er kun levende i roden. Døde celler bliver skudt ud, efterhånden som håret vokser, men de bliver lukket inde i keratin-skaftet. For at få fat i DNA'et bruger man et enzym, som nedbryder alle proteiner - herunder keratin, som også er et protein. Håret på et levende menneske har en vis fragmentering af DNA’et. Fragmenteringen opstår, når håret dannes. Men derefter sker der kun langsomt fragmentering af DNA’et, især langsomt hvis håret ligger i permafrost.
Test dig selv
Du kan tegne abonnement på BioNyt: Videnskabens verden her!
/DNA/Sekvens/Det korte svar er: Mellem en milliard og en billiion
I 1990, da man begyndte at kortlægge menneskets genom, kunne man finde 400 basepar pr. dag. Siden er gensekvens-bestemmelsesmaskinerne forbedret enormt, så man nu kan sekvens-bestemme 500.000.000.000 basepar i én kørsel, og endnu stærkere maskiner er på vej.
Test dig selv
Du kan tegne abonnement på BioNyt: Videnskabens verden her!
/DNA/Sekvensbestemmelse/Det korte svar er: Opklipninger og sorteringer på forskellige måder
DNA er store molekyler, som normalt kan DNA alligevel ikke ses i et almindeligt lysmikroskop – idet det dels vil afhænge af bølgelængden af det synlige lys i det elektromagnetiske spektrum (ca. 400 – 720 nm) og dels af en faktor, som kaldes den numeriske aperture.Læs mere her og her.
Men der findes mange andre teknikker til at undersøge DNA, RNA og andre nukleinsyremolekyler.
1) Restriktions-enzym analyser: Restriktionsenzymer er præcise, molekylære ”sakse”, som gør det muligt for forskere at manipulere med DNA-segmenter.
2) Blotting-teknikker: De teknikker, som kaldes Southern blot og Northern blot, bruges til at separere og karakterisere henholdsvis DNA og RNA. En anden af blottingteknikkerne er Western blot, som bruges til, ved hjælp af antistoffer, at karakterisere proteiner.
3) DNA-sekventering: Den præcise nukletidsekvens af DNA kan bestemmes. Sekventering har været brugt til at skaffe store mængder af information om geners arkitektur, cellens kontrol med genudtrykkelse (expressionen), og proteiners struktur.
4) Solid-phase syntese af nukleinsyrer: Præcise sekvenser af nukleinsyrer kan fremstilles helt fra bunden, eller de kan bruges til at genkende specifikke gen-sekvenser.
5) Polymerase Kæde Reaktion (PCR): Polymerase-kædereaktionen kan fremstille tusindvis af ens DNA-molekyler ud fra et stykke DNA. Et molekyle kan mangfoldiggøres til så store mængder, at de kan bruges til manipulation og karakterisering. Denne PCR-teknik er god til at fremstille meget DNA ud fra materiale, hvor der kun er begrænsede mængder af DNA, f.eks. i fossiler eller vævprøver i kriminalsager (PCR-metoden er beskrevet i BioNyt nr.72).
Test dig selv
Du kan tegne abonnement på BioNyt: Videnskabens verden her!
/DNA/Sekvensbestemmelse/Genbestemmelse/Det korte svar er: Med DNA-chips (biochips), hvormed man kan fiske mRNA
I praksis arbejder man med DNA-chips (biochips), hvormed man kan fiske mRNA, der er karakteristisk for en celles aktivitet, eller for en syg celles sygdom.
Test dig selv
Du kan tegne abonnement på BioNyt: Videnskabens verden her!
/DNA/Sekvensbestemmelse/Southern_blotting/Det korte svar er: Metode til at påvise mutationer i form af bl.a. deletioner (ikke effektiv til påvisning af enkeltbase-mutationer)
Denne teknik, der er opkaldt efter Ed Southern, som opfandt den, går ud på at skære genomisk DNA i mindre stykker, der har en størrelse, som gør det muligt at adskille småstykkerne ved elektroforese (f.eks. 1000 - 20.000 nukleotider i længde). Efter gel-elektroforese kunne han hybridisere disse fragmenter (som enkeltstrengede DNA-stykker) til radioaktive prober af faktor VIII genet. Derved kunne han påvise alle de fragmenter, som var tæt beslægtede med dette faktor VIII gen (i suppen af de millioner af ikke-beslægtede DNA-fragmenter).
Når et fragment fra faktor VIII genet havde en unormal størrelse, betød det, at det sygdomsfremkaldende mutationssted sandsynligvis var i det gen-område, som svarede til DNA-fragmentet. Southern blotting er fremragende til at finde DNA-ændringer, såsom deletioner (hvor baser mangler), men metoden er ikke effektiv til at finde enkelt-nukleotid-ombytninger (som hvis et A er blevet erstattet af et G). De fleste mutationer er sådanne enkelt-base ændringer. (9785s.65).
Test dig selv
Du kan tegne abonnement på BioNyt: Videnskabens verden her!
/DNA/Stregkode/Det korte svar er: Studiet er i gang
Teoretisk skulle det være muligt at få styr på alverdens levende organismer ved hjælp af små “DNA-stregkoder”, som ville kunne bruges til at artsbestemme organismer på grundlag af korte, velkarakteriserede dele af organismens DNA. Stregkoder kender vi bl.a. fra supermarkedet. Når stregkoden aflæses, oplyses varens pris. Hvis man tilsvarende kunne aflæse en unik DNA-sekvens for en organisme, ville man dels kunne få arten bestemt, og dels bruge disse data på anden måde. De industrielle stregkoder er unikke for det enkelte produkt. Arternes stregkoder ville også kunne gøres unikke for hver enkelt art. De industrielle stregkoder anvender 10 cifre på 10 positioner (plus 2 cifre til at markere landet og 1 kontrolciffer, i alt 13 cifre) for at danne 10 milliarder koder. Selv om DNA-stregkoder kun betjener sig af 4 forskellige farver, behøver man i teorien kun 15 positioner for at kunne danne 415 (ca. 1 milliard) biologiske stregkoder. Det totale antal nulevende arter på Jorden vurderes til i størrelsesordenen 10 - 15 millioner. Med en stregkodelæser med satellitforbindelse ville man kunne artsbestemme organismer, selv om man stod midt i en regnskov, ved blot at putte lidt DNA fra organismen ind i stregkode-apparatet og så (via satellit) sende data til f.eks. det verdensomspændende netværk af biologidatabaser Global Biodiversity Information Facility (GBIF). Det er faktisk visionen for et seriøst internationalt forskningsprojekt, International Barcode of Life (iBOLhttp://ibol.org/), der har hovedkvarter i Canada. Man håber i 2015 at have en DNA-stregkode-database med en halv million arter. Det vil i så fald svare til ca. 1/4 af de kendte arter med undtagelse af bakterierne. De levende organismer grupperes i dyreriget, planteriget, svamperiget, samt forskellige riger af protister, bakterier osv. Fælles for alle organismer er, at de har en fælles oprindelse, hvilket afspejles i arvematerialet. Nogle gensekvenser er konserverede, altså ens hos mange forskellige organismer. Disse gensekvenser er ubrugelige til artsbestemmelse. Andre DNA-områder varierer fra art til art. Sekvenser på ca. 500-700 baser vil være optimale, idet man helst vil have korte sekvenser, men samtidig skal de indeholde tilstrækkelig information (ref.9775). Consortium for the Barcode of Life (CBOLwww.barcodinglife.com) står bag iBOLprojektet. Projektets start støttedes af Alfred P. Sloan Foundation. Det blev i starten foreslået, at man skulle fremstille stregkoderne ud fra de typeeksemplarer, som arterne er beskrevet ud fra. Men det er unødvendigt og uønskeligt, da det ville kunne indebære ødelæggelse af typeeksemplaret. I princippet er det ret let at fremstille stregkoderne. Man oprenser noget DNA, og opformerer det ønskede gen (eller DNA-sekvens) ved hjælp af Polymerase Chain Reaction (PCR). Sekvensen ses på en computerskærm. De fremstillede stregkoder kan sammenlignes med stregkoder for kendte arter. Hvorvidt en stregkode svarer til vores opfattelse af en given art må undersøges på traditionel vis. Men stregkoden gør det meget nemmere at sammenkoble forskellige livsstadier hos dyreog plantearter. Det kan f.eks. være æg, larve, puppe og imago (“voksen”) hos insekter med fuldstændig forvandling (f.eks. biller og sommerfugle). Stregkoder ville kunne bidrage til at adskille arter, der er svære at skelne på ydre træk alene, f.eks. "tvillingearter", mikroorganismer, parasitter, samt fragmenter af arter. Stregkoden ville kunne give et fingerpeg om, hvorvidt en given prøve kan repræsentere en ny art (nemlig hvis stregkoden er ukendt), eller stregkoden ville kunne vise, at variationen for en kendt art skal revideres. Hvis man sekvensbestemmer et helt gen, vil man kunne få oplysning om arternes indbyrdes slægtskab. Endelig kan teknikken hurtigt og effektivt identificere parasitter samt epidemiske sygdomme som SARS, fugleinfluenza, AIDS, eller sygdomme, der benyttes som biologiske våben. Retsmedicinen har været pioner i brugen af stregkoder. Sekvenser, der optræder i mange kopier, er nemmest at arbejde med. Derfor bruges ofte DNA fra mitokondrier.-I-dyreriget har interessen samlet sig om et DNA-område på ca. 650 basepar, der findes i mitokondrierne, og som koder for proteiner i cellens stofskifte. Hos svampe og protister er variationen i mitokondrie-generne for stor, og hos planter er variationen i mitokondrierne for lille. Men for dyr er variationen passende til, at man kan anvende mitokondrie-DNA'et til universelle stregkoder. Det er derfor nødvendigt at bruge forskellige typer af sekvenser hos de forskellige grupper af organismer. Man kikker efter DNA, der har med organismens energiomsætning at gøre. Hos dyr kikker man ofte efter en gensekvens, som hedder COX1. Hos planter kikker man efter en kombination af to gensekvenser, kaldet rbc1 og matK. Gener i cellekernen vil bedre kunne afspejle artsdannelsen. Her samler interessen sig om variable områder i de gener, der koder for ribosomerne, som deltager i cellens proteinsyntese, og som i modsætning til mange andre gener i kernen findes i et meget stort tal (ref.9544).
Test dig selv
Du kan tegne abonnement på BioNyt: Videnskabens verden her!
/DNA/Transskription/Translation/Det korte svar er: Polyadenylering på poly(A)-siden, som signalerer til polymerasen, som stadig laver RNA, at den skal adskille sig fra DNA-strengen
Transkriptionsprocessen sker i to trin. Først vil transkriptionen blive udsat for en co-transkriptionel kløvning inden for terminerings-regionen nedenstrøms for polyadenylerings-sitet (se næste spørgsmål). Dernæst sker der en kløvning og polyadenylering på poly(A)-siden, som signalerer til polymerasen, som stadig laver RNA, at den skal adskille sig fra DNA-strengen. (Kilde: R.F. Weaver Molecular Biology 3.udg., 2005)
Test dig selv
Du kan tegne abonnement på BioNyt: Videnskabens verden her!
/DNA/Transskription/Translation/Det korte svar er: Efter transkriptionen sker der nogle postmodifikationer (efter-ændringer) af det ny-transkriberede RNA
Efter transkriptionen sker der nogle postmodifikationer (efter-ændringer) af det ny-transkriberede RNA (kaldet præ-mRNA). mRNA har nu to frie ender, som begge skal beskyttes. På den ene ende (5´ enden) bliver der påsat en cap, som er med til at stabilisere RNA-molekylet, samt beskytte det mod phosphataser og nucleaser, som begge er enzymer, der kan ødelægge RNA.
På den anden ende bliver der påsat en masse adenin-molekyler. Denne adenylering danner en poly(A) kæde. Det er vigtigt at bemærke, at denne poly(A) kæde ikke bliver kodet af en DNA-streng, men derimod bliver påsat af en poly(A)-polymerase, som påsætter ca. 250 adenylat-enheder på 3´ enden af mRNA.
præ-mRNA består af både såkaldte exons og introns. Det er kun exons, som koder for et protein i sidste ende, dvs. at alle introns bliver kløvet af. Der er flere kendetegn, som sikrer, at introns bliver genkendt: f.eks. begynder alle introns med baserne GU og ender med baserne AG. Lige inden de sidste AG-baser finder man også en konsensus-sekvens med ca. 10 pyrimidiner (dvs. enten U eller C). Desuden finder man mellem 20 og 50 baser opstrøms for AG et såkaldt branch site, som i f.eks. gærceller altid er UACUAAC. Alle disse steder skal være til stede, før enzymet spliceosome (som man kalder det enzym, som fjerner introns) kan genkende en intron. Spliceosomet består først og fremmest af to U-enheder, kaldet U1 og U2, og det genkender henholdsvis GU og branch site sekvensen. Når disse sekvenser er blevet genkendt, vil der bindes et yderligere U4-U5-U6 kompleks, som til sidst er i stand til at fjerne hele intron-sekvensen.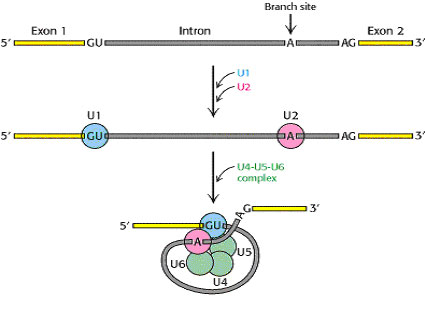
Test dig selv
Du kan tegne abonnement på BioNyt: Videnskabens verden her!
/DNA/Transskription/Translation/Det korte svar er: Poly(A) halen er nødvendig for transport af mRNA ud af kernen
Hvis det færdige mRNA blot skulle flyde rundt i kernen ved hjælp af tilfældige termiske bevægelser, ville sandsynligheden for, at mRNA´et ender ude i cytoplasmaet, være meget lille, selv om kernemembranen har mange porer. Derfor skal der være et transportsystem til stede.
I 2001 udførte Patricia Hilleren og kolleger studier på gærceller, som indeholdt en temperaturfølsom mutation i poly(A)-polymerasegenet. Konklusionen var, at de celler, som ikke kunne udtrykke poly(A)-polymerasen, ikke var i stand til at gennemføre en hel proteinsyntese, og man så en ophobning af mRNA-molekyler uden en poly(A)-hale i kernen. Dvs. at poly(A) halen er nødvendig for transport af mRNA ud af kernen. (kilde: P. Hilleren et.al. ”Quality control of mRNA 3´-end processing is linked to the nuclear exosome” Nature 413, s. 538-542 år 2001)
Test dig selv
Du kan tegne abonnement på BioNyt: Videnskabens verden her!
/DNA/Virus/Det korte svar er: De negative (-) strenge af dsDNA er dem, som bliver transskriberet til mRNA. Det mRNA, som bliver translateret til et protein, kaldes plus(+)
Virus kan have flere forskellige former for genetisk materiale. Virus kan enten have RNA eller DNA som arvemateriale, og både RNA og DNA kan være enten dobbeltstrengede (ds-formen) eller enkeltstrengede (ss-formen). De negative (-) strenge af dsDNA er dem, som bliver transskriberet til mRNA. Det mRNA, som bliver translateret til et protein, kaldes plus(+), og dette mRNA er en komplementær streng til den negative DNA streng.
Læs mere her
Virus er det eneste, som kan have enkeltstrenget DNA og dobbeltstrenget RNA. Der er enkelte steder i en organisme (bemærk, at virus ikke er en organisme), hvor der findes dobbeltstrenget RNA i cellen (f.eks. i tRNA), men der findes ikke eksempler hos organismer, hvor dobbeltstrenget RNA indgår som arvemateriale (altså bortset fra virus, der som sagt ikke er organismer).
Eksempler på forskellige virus og den form for arvemateriale, som de har:
DNA ds lineær: Myoviridae T4, Siphoviridae
DNA ss cirkulær: Microviridae, Inoviridae M13
RNA ds lineær: Cystoviridae
RNA ss lineær: Leviviridae MS2
Test dig selv
Du kan tegne abonnement på BioNyt: Videnskabens verden her!
/Enzym/Ribozym/Det korte svar er: Ribozymer er ikke proteiner
Ikke alle enzymer er proteiner. Ganske vist er størstedelen af alle enzymer proteiner, men der findes også aktive RNA-stykker, som ligeledes har en katalytisk virkning og derfor er enzymer. Disse aktive RNA-stykker findes bl.a. i enzymet telomerase. De aktive RNA-stykker kaldes ribozymer.
Test dig selv
Du kan tegne abonnement på BioNyt: Videnskabens verden her!
/Evolution/Det korte svar er: Generne, og dermed de karaktertræk de koder for, bliver med opståede mutationer undervejs arvet ned igennem generationerne
Gener er portioner af en organismes DNA, som er afgørende for en korrekt opbygning af en organisme. Generne, og dermed de karaktertræk de koder for, bliver arvet ned igennem generationerne. Forskellige molekylære mekanismer sørger for at kopiere, blande og ændre generne på en måde, så der opstår genetisk variation. Denne variation er ophav til evolutionen.
Læs mere her
Test dig selv
Du kan tegne abonnement på BioNyt: Videnskabens verden her!
/Evolution/Det korte svar er: Den genetiske variation, som den naturlige selektion udnytter, kan være tilfældig, men naturlig selektion i sig selv er slet ikke tilfældig, idet de karaktertræk, der bedst sikrer genets videreførelse i arten, har størst chance for netop at blive videreført.
Evolutionen er ikke en tilfældig proces. Den genetiske variation, som den naturlige selektion udnytter, kan være tilfældig, men naturlig selektion i sig selv er slet ikke tilfældig. Overlevelsen og den forplantningsdygtige succes af et individ er direkte relateret til den måde, hvormed individet nedarver sine karaktertræk samt hvilken påvirkning omgivelserne giver. Om et individ overlever eller ej afhænger af, om dette individ har gener, som producerer karaktertræk, som passer godt til det omgivende miljø.
Test dig selv
Du kan tegne abonnement på BioNyt: Videnskabens verden her!
/Evolution/Store_dyr/Det korte svar er: At de uddøde af forskellige grunde for hver art
DNA-studier samt klimastudier mv. har vist, at årsagen til de store dyrs uddøen skyldtes kombinationer af årsager - der findes ikke én fælles årsag; det afhang af arten, konkurrence fra andre arter, klimaet, mennesket osv.
Ved den sidste istids begyndelse for ca. 115.000 år siden(Weichsel-istiden) fandtes vidt udbredte, store bestande af bison, moskusokse, rensdyr, vildhest, uldhåret næsehorn og mammut. Af disse overlevede kun bison, moskusokse og rensdyr til nutiden. I november 2011 publicerede Eske Willerslevs gruppe fra Københavns Universitet en artikel om de store pattedyrs uddøen under den sidste istid (ref.9581). For en periode på 50.000 år sammenlignede de DNA-data med klimadata og 1600 arkæologiske udgravninger (ref.9581). De fandt, at mennesket absolut ingen rolle spillede i udryddelsen af det uldhårede næsehorn eller moskusokse-bestanden i Eurasien, fordi disse to arter levede nordligere end mennesket (omend mennesket kan have været medvirkende til næsehornets uddøen i Europa). Næsehornet nåede aldrig til Nordamerika. Disse arters uddøen skyldtes andre årsager, formentlig især klimaændringerne. Omvendt skyldtes uddøen af de vilde heste og bisonokser i Sibirien næsten udelukkende menneskets jagttryk. 2/3 af Sibiriens stenalderbopladser viser rester efter heste, og i 3/4 ses rester efter steppebison. I Nordamerika skete der en hurtig tilbagegang af steppebison for ca. 11000 år siden, og mennesket var årsag til denne tilbagegang. Rensdyret var ret upåvirket af både jagt og klima og kan have overlevet dels fordi rensdyr reproducerer sig hurtigt, med flere kalve, og dels fordi rensdyret tidligt blev tæmmet af mennesket (ref.9582). Årsagen til, at den uldhårede mammut uddøde, er stadig et mysterium. I Europa og Asien var der en femdobling af bestanden for ca. 26.000 år siden, selv om mennesket formentlig havde jaget det i de forrige 10.000 år. Mammuten var næsten helt uddød for ca. 10.500 år siden, bortset fra et en dværgform klarede sig indtil for ca. 4000 år siden på Wrangel-øen i ishavet ud for Sibirien. Mammutbestandene var ret indavlede, så sygdomme var måske en medvirkede årsag (ref.9583). Ved at studere DNA i ekskrementer har man kunnet vise, at der var stort overlap af planter, som arterne spiste, så dyrene konkurrerede altså om føden. Studiet kan tyde på, at steppebisonen overlevede i Nordamerika, fordi vildhesten og mammuten uddøde. I Asien og Europa kan uddøen af steppebisonen, mammut og det uldhårede næsehorn have været årsag til, at hesten overlevede her (dog kun som tamhest). Man kan konkludere, at de store arters uddøen skete over flere tusind år, og årsagerne var bl.a. klima, jagt og indbyrdes konkurrence, men hvilke arter, der uddøde, var tilfældigt. Hvis hele scenariet kunne genopføres, ville resultatet sikkert blive et andet. Vi kunne godt have haft mammut i dag eller være gået glip af hesten. Det er utrolig svært at forudsige, hvem der bliver taberne. Moskusokserne overlevede i isfri områder, hvor der ikke var jagt og konkurrence fra andre planteædere. Steppebison overlevede, fordi den ved at bevæge sig over store afstande kunne finde steder uden for konkurrence. “Vores resultater sætter en endelig stopper for enkelt-årsag teorier om istidens uddøen”, fortæller Eske Willerslev. Man bør være forsigtig med at generalisere om årsager til uddøden - det gælder nok både for fortiden, nutiden og fremtiden. Virkningerne af klimaændringer og menneskelige indgreb i forhold til arters udryddelse afhænger af, hvilke arter man kigger på.“ Dog har klimaforandringerne været tæt forbundet med de store dyrs bestandsændringer i de seneste 50.000 år, og det giver støtte til det synspunkt, at bestandene af mange arter vil falde i fremtiden på grund af klimaændringer og tab af levesteder (ref.9555).
Test dig selv
Du kan tegne abonnement på BioNyt: Videnskabens verden her!
/Evolution/Øer/Det korte svar er: At evolutionsprocesserne sker hurtigere ved isolering
(artikel af Finn Bjerre)
Da Charles Darwin midt i 1800-tallet - efter sin jordomrejse med det britiske orlogsskib ”The Beagle” - fik udgivet sit berømte værk ”The Origin of Species by Means of Natural Selection or The Preservation of favored Races in the Struggle for Life” eller i J. P. Jacobsens oversættelse ”Arternes Oprindelse ved naturligt Udvalg eller ved de heldigst stillede Formers Sejr i Kampen for Tilværelsen” havde han indsamlet en stor mængde prøver af dyr og planter og naturhistoriske observationer, ligesom han også samlede en næsten utrolig mængde oplysninger og data fra andre naturforskere og meddelere fra store dele af verden. Blandt hans egne observationer var de tydelige forskelle mellem finkerne på Galapagos-øerne i Stillehavet. Disse finker, som udgør en underfamilie (Geospizinae) af finkefamilien (Fringillidae), omfatter seks slægter og 14 arter, som med en enkelt undtagelse kun findes på denne øgruppe (en art findes på Cocos-øerne). De adskiller sig fra hinanden ved deres størrelse og ved næbformen og næbstørrelsen. Næbform og næbstørrelse svarede til deres fødevalg. Nogle af arterne er vegetarer og spiser udelukkende plantefrø, mens andre er insektædere. Det anses for at være sandsynligt, at Galapagos-finkerne alle nedstammer fra en lille gruppe af fugle, måske et enkelt par, som i forbindelse med en voldsom storm er fløjet eller er blevet blæst ud over Stillehavet fra Sydamerika. Det naturlige udvalg eller selektionen har påvirket deres efterkommere anderledes end på fastlandet, fordi de og deres gener kun udgjorde en meget lille og begrænset del af fastlandets finkebestand og af disses genmasse. Stillehavet og afstanden mellem Galapagos- øerne og fastlandet udgjorde med andre ord en flaskehals, som kun nogle få individers gener slap igennem. Da finkerne først havde etableret sig på en af øerne, var det kun et spørgsmål om tid, før nogle af deres efterkommerne nåede frem til andre af øerne i øgruppen. Bestande på de forskellige øer, hvis miljø og fødekilder varierer, blev underkastet forskellige selektionstryk, og dette kombineret med ”flaskehalsprincippet” bevirkede, at de hurtigt (i løbet af få generationer) udviklede sig forskelligt. Galapagos-finkernes størrelse og deres næbform hænger sammen med deres fødevalg, og fuglene bruger også disse egenskaber og forskelle til at finde og identificere deres mager. Mængden og tilgængeligheden af forskellige fødekilder varierer mellem øerne, og det er sandsynligt, at det naturlige udvalg/selektionen ved isoleringen af bestandene på øerne førte til en differentiering af fuglenes næb. En sådan differentiering synes også at give en ekstra fordel, idet den formindsker antallet af individtyper, som udnytter de samme fødekilder, og derved begrænser konkurrencen mellem individer af samme art, men af hver sin type. Darwin og andre naturforskere fandt lignende udviklingsmønstre i isolerede øgrupper, hvor plante- og dyrearterne ikke findes magen til andre steder i verden. Sådanne arter kaldes endemiske arter. Darwin nævner blandt andet de oprindelige plante- og dyrearter på St. Helena, landsneglearterne på Madeira og fuglearterne på Bermuda. Senere har evolutionsbiologer fundet mange andre eksempler på sådanne evolutionsmønstre, f.eks. i de store østafrikanske søer Victoria-, Tanganyika- og Nyasa-søerne, hvor cichlider (en fiskefamilie) fra floder og vandløb gentagne gange med tidsmellemrum koloniserede Victoriasøen, og hvor bestandene har udviklet sig så forskelligt, at der nu findes godt 70 forskellige endemiske arter af slægten Haplochromis, som lever i tre forskellige økologiske omgivelser og med forskellige fødevalg med deraf følgende forskellige kæbe- og kraniestrukturer. Mens de færreste danskere har haft lejlighed til selv at opleve galapagosfinkerne på Galapagos-øerne eller cichliderne i de østafrikanske søer, har mange danskere som turister besøgt
Test dig selv
Du kan tegne abonnement på BioNyt: Videnskabens verden her!
/Evolution/Øer/Galapagos/Det korte svar er: Darwins finker fra Galapagos-øerne viser evolutionen, idet de er udviklet evolutionært fra samme finke-stamform
Darwins finker fra Galapagos-øerne er udviklet evolutionært fra samme finke-stamform, men øernes forskellige miljøer og udvikling har presset finkernes evolution i forskellige retninger. Deres enormt forskellige næb er udtryk for, at de nye arter har forskellig føde. DNA-undersøgelser kan bruges til at fastlægge slægtskab, som kan være svært at få øje på umiddelbart. www.bionyt.dk/DNA/sekventering De forskellige moderne metoder til at lave DNA-sekventeringer er beskrevet på siden FOTO: På et slide (reagensglas) fanges DNA-fragmenter, der opformeres ved en parallel form for PCR-opformering, hvor fluorescerende basepåsætninger ved laserbelysning viser base-rækkefølgen. Foto: Mikal Schlosser
Test dig selv
Du kan tegne abonnement på BioNyt: Videnskabens verden her!
/Evolution/Øer/Kanariske_øer/Det korte svar er: Oprindelsen af og skæbnen for befolkningsgrupperne på øerne
(artikel af Finn Bjerre)
De kanariske Øer består af vulkanske bjergarter og er dannet ved undersøiske vulkanudbrud. Deres oprindelige flora og fauna formodes at være kommet til øerne med vinden og med havstrømmene ligesom tilfældet er for andre øgrupper, som befinder sig i oceanerne langt fra de nærmeste fastlande. De største og bedst kendte af De kanariske Øer er Tenerife, Gran Canaria, Lanzarote, Fuerteventura og La Gomera. Disse fem øer og de andre mindre øer (især El Hierro og La Palma - der ikke må forveksles med hovedstaden Las Palmas på Gran Canaria) menes at have modtaget de første menneskelige indvandrere omkring år 400 før vor tidsregning (fvt.). Disse mennesker var efter alt at dømme berbere fra Nordafrika, der som slaver blev deporteret af romerne på grund af opstande blandt berberne, som modsatte sig den romerske kolonisering. Nogle forskere (antropologer og arkæologer) mener, at andre indvandrergrupper måske som slaver blev indført med fønikiske skibe og blev efterladt på øerne. Forskellige grupper af indvandrere kan have befolket hver sin ø eller dele af en ø. De synes at have manglet kendskab til skibsbygning og søfart, eller også er denne viden med tiden gået tabt, i forbindelse med at befolkningstilvæksten på øerne var så stærk, at folkene på de forskellige øer blev fjendtligt indstillet overfor hinanden (på øen La Palma fandtes et dekret, som påbød beboerne at slå udefra kommende folk ihjel). Der gik omkring 1800 år, altså næsten totusinde år, før spanierne i 1400-tallet erobrede og koloniserede øerne. Indbyggerne på de forskellige øer var isolerede uden indbyrdes kontakt på grund af manglen på skibe og både, og i dette lange tidsrum fandt en kulturel udvikling sted, som kan minde om differentieringen af de endemiske arter på Galapagos-øerne og andre steder i verden. Indbyggernes sprog udviklede sig så forskelligt, at da spanierne erobrede øerne, havde folkene fra de forskellige øer svært ved at forstå hinanden. På nogle af øerne søgte indbyggerne tilflugt i naturlige huler og grotter i de vulkanske bjergarter og indrettede sig som hulemennesker. Nogle af disse huler er stadig i brug, f.eks. på Gran Canaria, hvor de er attraktive boliger, som er indrettede med elektricitet og moderne bekvemmeligheder. Opvarmning er unødvendig, da temperaturen i hulerne er konstant omkring 20 grader hele året. Dette skyldes den vulkanske varme fra undergrunden. Beboerne skal ikke betale ejendomsskatter af deres huleboliger, som i øvrigt via ejendomsmæglere sælges til overkommelige priser. Andre steder efterlignede man de naturlige huler ved at bygge lave huse/ hytter af natursten. Klædedragten og den keramiske udformning af skåle og potter udviklede sig også forskelligt fra ø til ø, så specialister, der får forelagt et stykke tøj eller en skål, kan se, hvorfra det/den stammer. Øernes beboere og deres samfund udviklede sig også forskelligt på andre områder såsom samfundenes sociale organisation og begravelsesskikke, så man fristes til at kalde dem endemiske. De oprindelige beboeres tilværelse og deres kulturelt forskellige udviklingsmønstre blev brat afbrudt af de spanske erobringer i 1400-tallet, som sluttede i 1496, da Tenerife blev erobret. Beboerne gjorde modstand og ville ikke underkaste sig. Spanierne var teknologisk overlegne med hensyn til våben, og mange af øernes mandlige beboere omkom i kampene med spanierne. Mange andre blev taget til fange og blev eksporteret som slaver til markeder i de spanske kongeriger Castilien, Aragonien og i Portugal. Epidemier fremkaldt af sygdomme, som spanierne førte med sig, reducerede yderligere størrelsen af den oprindelige befolkning, men den blev ikke udryddet. Tilflyttere fra Spanien, Portugal og andre dele af Europa fandt vej til De kanariske Øer, og nogle af dem medbragte negerslaver fra Nordafrika og fra Vestafrika syd for Sahara. De fleste af tilflytterne var mænd, og mange af dem giftede sig med kvinder fra den oprindelige befolkning, som jo havde mistet deres mænd i erobringskampene eller havde set dem blive bortført som slaver. Nutidens befolkning på øerne er altså sammensat af elementer af den oprindelige befolkning, europæere og afrikanere, og den endemiske differentiering mellem øernes beboere synes at være ophørt. Skæbnen for øernes oprindelige indbyggere dokumenteres af moderne genetiske undersøgelser af den mandlige befolknings Y-kromosomer, hvis gener kun nedarves via mandslinjerne, og af mitokondrie-DNA, der nedarves via kvindelinjerne. Mellem 6 % og 8 % af de nutidige mænds Y-kromosomgener viser en sammenhæng med de oprindelige mænds arveanlæg, mens tallet for kvindernes vedkommende er ca. 50 %. Dette viser, at kun få af de oprindelige mænd, (formentlig drenge, som under de spanske erobringer ikke var gamle nok til at deltage i kampene eller til at blive gjort til slaver, men som senere blev kønsmodne), førte deres gener videre til de følgende generationer, mens de oprindelige kvinder i langt højere grad har været i stand til at give deres arveanlæg videre til nutidens indbyggere (ref.9624).
Test dig selv
Du kan tegne abonnement på BioNyt: Videnskabens verden her!
/Ernæring/Det korte svar er: Talrige bl.a. hormon-virkende effekter i kroppen
D-vitamin er vigtig af flere årsager. Det er bl.a. vigtigt for immunsystemet, og hvis der er alvorlig mangel på D-vitamin (som det var tilfældet for børn, der levede i industri-slumbyer i 1700-tallet og 1800-tallets England) kan der udvikles "Engelsk syge". Det skyldtes også dårlig kost. En alvorlig følgevirkning af Engelsk syge (D-vitaminmangel) er forsnævring af fødselskanalen, der under primitive forhold kan medføre, at både barnet og moderen dør under fødslen (ref.9706).
Test dig selv
Du kan tegne abonnement på BioNyt: Videnskabens verden her!
/Ernæring/Det korte svar er: Hjernens udvikling under fosterudviklingen
Folinsyre er nødvendig for udvikling af hjernen under fosterudviklingen - og for sædcelledannelsen hos mænd. Hvis en gravid kvinde er i underskud med folinsyre risikerer hun, at barnet fødes med svære misdannelser af hjernen og rygmarven (neuralrørsdefekt, f.eks. rygmarvsbrok eller alvorlige misdannelser er langt mere almindelige hos de lyse europæere end hos de mørke, indfødte afrikanere (ref.9704).
Test dig selv
Du kan tegne abonnement på BioNyt: Videnskabens verden her!
Homo/Det korte svar er: Menneskelinien startede for ca. 6 mill. år siden
Menneskets udvikling starter for så vidt med livets opståen for mindst 3800 mill. år siden; de flercellede dyr for 600 mill. år siden; pattedyrene (i starten æglæggende) for 225 mill. år siden. Primaterne var unikke ved at kombinere gribehånd/fod, atletiske evner, farvesyn med kompliceret social adfærd og hjernebark-intelligens. De udvikledes for 70 mill. år siden. Det var på et tidspunkt efter Gondwanaland- superkontinentets opbrud til mindre kontinenter og efter dinosaurernes uddøen. Udvikling af menneskeaberne skete for 25 mill. år siden, da Jordens klima steg 15 grader og faldt igen. Det var omkring tidspunktet, hvor den østafrikanske riftzone indledtes (for 20 mill. år siden). Der opstod nu en stamfader til gorilla, chimpanser og mennesket. Mest sandsynligt var denne stamform Drypithecus (som indvandrede fra Europa (!) til Afrika for 9 mill. år siden) (ref.9655). Asiens orangutang er en fjernere gren af menneskeaber. På et tidspunkt, hvor lange tørkeperioder i Østafrika ændrede regnskoven til mere åbent land, opstod menneskelinien for 6 mill. år siden.
Test dig selv
Du kan tegne abonnement på BioNyt: Videnskabens verden her!
/Homo/Arter/Det korte svar er: Nej
Eske Willerslevs gruppe fra Københavns Universitet på Københavns Universitet arbejder bl.a. med sekvenser fra et halv million år gammelt fund i Spanien af Homo heidelbergensis - et af de første mennesketyper, man kom til Europa. Det er meget, meget vanskeligt at arbejde med, fortæller Eske Willerslev. Men måske har man fundet nogle DNA-sekvenser, som er Homo heidelbergensis.
Homo heidelbergensis er opkaldt efter et tysk fundsted: Heidelberg. Man diskuterer, om det var et forstadie til neanderthaleren, eller om det er en separat gren af Homo-slægten.
Test dig selv
Du kan tegne abonnement på BioNyt: Videnskabens verden her!
/Homo/Arter/Det korte svar er: Måske
Eske Willerslev skal til Kina for at se på et fund af fem fund af fortidsmennesker, der er 12.000 år gamle, og som man mener potentielt kan være en ukendt Homo-art, beslægtet med det moderne menneske, men med kæmpekæber og anderledes udseende.
Test dig selv
Du kan tegne abonnement på BioNyt: Videnskabens verden her!
/Homo/DNA/Genom/Aborigin/Det korte svar er: Et dansk ledet forskerteam
Et dansk ledet forskerteam (Eske Willerslevs gruppe fra Københavns Universitet på Københavns Universitet) opnåede at blive de første, som kortlagde en aboriginers fulde genom. (Tyskeren Mark Stoneking, molekylær antropolog ved Max Planck Instituttet for Evolutionær Antropologi i Leipzig, Tyskland, har publiceret en SNiP-analyse af aborigin-DNA skaffet via andre forskere, som har fået samtykke fra aborigin-australiere (ref.9556 note2. Dette var ikke en fuld genom-sekvensbestemmelse).
Test dig selv
Du kan tegne abonnement på BioNyt: Videnskabens verden her!
/Homo/DNA/Genom/Aborigin/Det korte svar er: En dansk-ledet forskergruppe
Et dansk ledet forskerteam (Eske Willerslevs gruppe fra Københavns Universitet på Københavns Universitet) opnåede at blive de første, som kortlagde en aboriginers fulde genom. (Tyskeren Mark Stoneking, molekylær antropolog ved Max Planck Instituttet for Evolutionær Antropologi i Leipzig, Tyskland, har publiceret en SNiP-analyse af aborigin-DNA skaffet via andre forskere, som har fået samtykke fra aborigin-australiere (ref.9556 note2. Dette var ikke en fuld genom-sekvensbestemmelse).
Aboriginerne er urbefolkningen i Australien. Det lykkedes den dansk-ledede gruppe at sekventere et aborigin-genom uden at der i genomet var genetiske spor efter de moderne immigranter til Australien. Europæerne kom til Australien i 1780'erne, og i nord-Australien kan der have været kontakt med andre befolkningsgrupper. Men i det sydvestlige Australien bosatte europæere sig først rigtigt for ca. hundrede år siden. *Se: Hvad betyder ordet "aboriginer"?
Test dig selv
Du kan tegne abonnement på BioNyt: Videnskabens verden her!
/Homo/DNA/Genom/Aborigin/Det korte svar er: Egentlig "fra begyndelsen"
Betegnelsen "aboriginer" kommer fra latin: ab origine, dvs. “fra begyndelsen”, og betyder “de oprindelige indbyggere”. (Betegnelsen bruges dog også om oprindelige folk i Canada og Taiwan).
Test dig selv
Du kan tegne abonnement på BioNyt: Videnskabens verden her!
/Homo/DNA/Genom/Aborigin/Det korte svar er: En hårtot af en ung aboriginer, indsamlet 1923 af etnolog Alfred Cort Haddon
Historiske optegnelser tyder på, at den britiske etnolog Alfred Cort Haddon fik håret fra en ung mand, som han stødte på under en togrejse fra Sydney til Perth. Ifølge hans nedskrevne noter kom ham på togrejsen igennem et guldgraver-område, som i dag kaldes Goldfields. I et område, som kaldes "Golden Ridge", mødte han på en lille togstation en ung aboriginer, som var traditionelt klædt. I håret havde han en masse ler, som har været brugt til at sætte håret. Alfred Cort Haddon fik en hårprøve, eller byttede sig til den. Der var næppe vold indblandet, for man var begyndt at få en noget mere human attitude (cirka i 1910 var man holdt op med at hugge hovederne af aboriginerne). *Se: Hvem var Alfred Cort Haddon?
Test dig selv
Du kan tegne abonnement på BioNyt: Videnskabens verden her!
/Homo/DNA/Genom/Aborigin/Det korte svar er: Britisk etnolog
Alfred Cort Haddon var en britisk etnolog, der er meget berømt inden for antropologi- kredse. Han samlede livet igennem hår fra hele verden, idet han mente, at hårets former kunne vise, hvordan befolkningsgrupper er beslægtet, og hvem der nedstammer fra hvem. Hele samlingen endte i Cambridge i England.
Test dig selv
Du kan tegne abonnement på BioNyt: Videnskabens verden her!
/Homo/DNA/Genom/Aborigin/Det korte svar er: I 1780'erne, dog over 100 år senere til visse øde egne
Europæerne kom til Australien i 1780'erne. I Nord-Australien kan der have været kontakt med andre befolkningsgrupper. Men i det sydvestlige Australien bosatte europæere sig først rigtigt for ca. hundrede år siden.
Test dig selv
Du kan tegne abonnement på BioNyt: Videnskabens verden her!
/Homo/DNA/Genom/Aborigin/Det korte svar er: Nej, det var uden europæisk indflydelse
Aboriginer-genomet fra hårprøven var uden genetiske spor efter de moderne immigranter til Australien. *Se: Hvilket materiale anvendtes til at finde aborigin-genomet?
Test dig selv
Du kan tegne abonnement på BioNyt: Videnskabens verden her!
/Homo/DNA/Genom/Aborigin/Det korte svar er: 0,6 gram af en hårtot af en ung aboriginer, indsamlet 1923 af etnolog Alfred Cort Haddon
Man anvendte 0,6 gram af en hårprøve, som i aug.-sep. 1923 var indsamlet af Alfred Cort Haddon, en britisk etnolog, der er meget berømt inden for antropologi-kredse. Han havde en slags hårfetich, og livet igennem samlede han hår fra hele verden, idet han mente, at hårets former kunne vise, hvordan befolkningsgrupper er beslægtet, og hvem der nedstammer fra hvem. Så han indsamlede hår og klippede hår overalt, og hele samlingen endte i Cambridge i England, i Leverhulme Centre for Human Evolutionary Studies ved Cambridge universitet. "Det er surrealistisk at gå rundt der i denne kæmpestore samling af hårprøver", fortæller Eske Willerslev. "Man åbner den ene skuffe efter den anden med hår fra alverdens folkeslag".
Historiske optegnelser tyder på, at Alfred Cort Haddon fik håret fra en ung mand, som han stødte på under en togrejse fra Sydney til Perth, hvor han deltog i en stor Pan-Pacific Conference, som blev afholdt i Sydney og Melbourne, men historikeren Craig Muller fra Australien fandt ud af, at Haddon havde sejlet til England fra Perth, og derfor havde krydset Australien med den transaustralske jernbane. Vi ved fra hans nedskrevne noter, at han på togrejsen kom igennem et guldgraver-område, som i dag kaldes Goldfields. Og der, i et område, som kaldes "Golden Ridge", står Alfred Cort Haddon på en lille togstation, og her møder han en ung aboriginer, som fanger hans interesse, fordi han er traditionelt klædt. I håret er en masse ler, som har været brugt til at sætte håret.
Alfred Cort Haddon får eller bytter sig til en hårprøve. (Eske Willerslev fortæller: "Da det er i 1920'erne, og Haddon er nær de 90 år, går vi ud fra, at det ikke var ved fysisk vold. Man var begyndt at få en noget mere human attitude - ca. 1910 var man holdt op med at hugge hovederne af aboriginerne. Hårets farve, tykkelse og mikrostruktur passer med at det fra en aboriginer fra dette tidspunkt.
Eske Willerslev har besøgt stedet, hvor den togstationen lå. Den findes ikke mere - man finder blot nogle flasker, tobaksdåser og den slags.
Test dig selv
Du kan tegne abonnement på BioNyt: Videnskabens verden her!
/Homo/DNA/Genom/Aborigin/Det korte svar er: Forsøg med menneskeligt væv, æg og celler skal godkendes af en videnskabsetisk komité
Alle biomedicinske forskningsprojekter, der involverer mennesker eller menneskeligt biologisk materiale såsom væv, æg og celler, skal godkendes af en videnskabsetisk komité, inden forskningsprojektet sættes i gang (ref.9574).
Test dig selv
Du kan tegne abonnement på BioNyt: Videnskabens verden her!
/Homo/DNA/Genom/Aborigin/Det korte svar er: Ikke i starten
Eske Willerslev fortæller, at da han startede projektet havde han aldrig været i Australien, men blot læst på teorierne om, hvor aboriginerne kom fra. Han vidste ikke meget om alle de problemer, som aboriginerne har haft, og hvordan de er blevet behandlet. For at sekvensbestemme genomet gik Eske Willerslev gennem de lovlige kanaler. Håret var ejet af England, og det var lovligt i forhold til British Tissue Act, at man kunne udlevere en sådan hårprøve til sekvensbestemmelse. Der findes ingen international standard for, hvilken forskning museernes arkæologiske samlinger må bruges til. Mange museer tilbagesender knogler, der er indsamlet fra indfødte stammer, men British Museum har den politik, at hår og negle ikke sendes retur. Eske Willerslev gik også til Videnskabsetisk Komité i Danmark. De sagde: "Jamen, her er jo ingen problemer - det er et arkæologisk fund, og derudover er det blot demografiske studier, som I kikker på, så I behøver overhovedet ikke at få en godkendelse. Bare gå i gang". (Alle biomedicinske forskningsprojekter, der involverer mennesker eller menneskeligt biologisk materiale såsom væv, æg og celler, skal godkendes af en videnskabsetisk komité, inden forskningsprojektet sættes i gang (ref.9574). Men den danske komité mente altså ikke, at det krævede en godkendelse i dette tilfælde, fordi der var tale om arkæologisk materiale)
I løbet af processen blev Eske Willerslev imidlertid opmærksom på, at det er et superfølsomt emne. Aboriginerne er blevet rigtig dårligt behandlet, også af videnskabsfolk. Det gik op for Eske Willerslev, at selv om han rent lovmæssigt ikke behøvede det, så behøvede han rent moralsk en godkendelse fra de mennesker, det drejede sig om. Problemet var så, hvordan man skulle gribe dette an. I Haddons noter nævnes intet stammenavn, men ved at tale med en arkæolog fik Eske Willerslev at vide, at Australien er delt op i regioner, hvor lokalråd, "Council Boards", repræsenterer de lokale stammer. "Vi vidste jo, at denne person levede ved Golden Ridge". Det vil sige, at det mest naturlige var at sige: "Ok, vi vil prøve at opsøge det råd, som dækker alle de stammer, som Golden Ridge er en del af. Med meget stor sandsynlighed har man derved opsøgt de biologiske efterkommere af de stammer, som den unge mand kan have tilhørt. Lokalrådet Goldfields Land and Sea Council i Kalgoorlie, Western Australia, repræsenterer de ca. 5000 aboriginere, der bor i det område, som Haddon havde hårprøven fra, og Eske Willerslev tog flyveren til Australien.
På dette tidspunkt var man langt i processen: "Vi havde faktisk investeret et års arbejde af cirka 40 mennesker. Der var brugt flere millioner kroner til dette projekt. Eske Willerslev fik - upfront - at vide, at det ville blive et nej. Aboriginerne bryder sig ikke om DNA-forskning. Ingen kunne erindre, at der nogensinde på stammeniveau var givet tilladelse til at udføre DNA-studier. "Men der var ikke andet at gøre - jeg måtte derned. Det gjorde jeg så. Jeg sendte et brev i forvejen om, hvem jeg var, og hvad jeg ville, samt hovedresultaterne af projektet, som man kendte dem på dette tidspunkt - og så spurgte jeg, om jeg måtte komme ned og fremlægge min sag for dem. Det fik jeg så at vide, at det måtte jeg gerne. Og jeg fløj så hele vejen til Perth. Der kunne jeg ikke komme videre med fly ud til disse skide Goldfields, som lå ad Helvede til ude i krakkemutten. Så vi måtte med bil - en ca. femten timers kørsel for at komme derud".
"Og så kommer jeg ind - og der må jeg sige, der havde jeg s'gu sved på panden - da jeg skulle ind til det møde, for jeg vidste jo, hvad der var på spil. Jeg vidste, at hvis de sagde nej til dette her, så var det end-of-story, altså. Så var alt det arbejde, vi havde lagt i det, det var færdigt. Vi ville aldrig gå ud og pu blicere det, og vi ville ikke gøre noget ved det." Eske Willerslev fik hjælp fra Craig Muller, der har med forskning for Goldfield-rådet at gøre. Craig Muller havde undersøgt hårprøvens historie og konkluderet, at manden havde doneret sin hårlok frivilligt til Haddon. "Men ham australieren, arkæologen, der var med mig, han sagde: Det kommer aldrig til at gå. De siger nej ".
"Men jeg lover dig for, at jeg blev mødt af det fuldstændigt modsatte. Det første, de sagde til mig, var, 'Hvorfor har du ikke medbragt de der redskaber, så du også kan tage DNA-prøver af os?' Nå, tænkte jeg så. Jo, sagde en, 'jeg er ved at være gammel, så du skal huske at få en DNA-prøve af mig'. Og de sagde så, at 'vi synes det er et fantastisk studie, og det er rart, at der endelig er nogen, som kan lave noget ordentlig videnskab. Det kan de nemlig ikke finde ud af her i Australien. Kommer du ikke herover, og slår dig ned, og arbejder'. Det var helt uforståeligt, hvad der foregik".
"Og hvad er det så, der gjorde, at det gik godt. Ja, det er der mange, der har spurgt om. Jeg fik tilladelsen - endda et dokument, sort på hvidt med stempel og underskrevet af hele moletjavsen om, at det her ønskede de blev publiceret, at det var frit tilgængeligt, og at der ikke var nogen restriktioner på det - man kan bruge det til al den videnskab, man ønsker. Jeg har talt med sidste 10 år kæmpet for at få lejlighed til at sekvensbestemme et aboriginergenom. Og så kommer jeg flyvende ind, tager genomet og flyver ud igen. Så de var rimeligt bitre. Det kan man roligt sige. De var faktisk pissebitre". "Hvad er det så, der gør det? Ja, altså, det er s'gu et godt spørgsmål. Jeg har ikke noget endeligt svar. Jeg tror, at der var nogle ting, som spillede en rolle - det fik jeg i hvert fald at vide. Det ene er, at jeg er dansker. Man kan sige, at som videnskabsmand plejer det altid at være skidegodt at komme fra USA eller Amerika eller sådan noget. Harvard for eksempel, men København, det ved de ikke, hvad er. Danmark i det hele taget - super harmløst. De tror, det er en by i Sverige. Vi har ikke nogen kolonihistorie, som folk i hvert fald ude i verden bliver meget ophidset over. Det betød noget. Det betød noget, at jeg ikke var australier, at jeg ikke var tysker og ikke var englænder." Og det betød noget, at jeg kom. De sagde til mig, at det havde de respekt for. At man flyver i 22 timer hver vej for at komme ned til et møde, der tager 1 time. For simpelthen at fremlægge min sag. Det betød virkelig meget."
"Og så vil jeg sige, så tror jeg faktisk, at en anden ting betød noget - det var, at jeg kunne fortælle dem, hvad resultatet var. Jeg ved godt, at hvis det skulle være gået fuldstændig efter bogen, så skulle jeg have været dernede og skulle have sagt, at jeg ville teste disse teorier. Men så havde jeg måske fået et nej. Det tror jeg i hvert fald. Men fordi jeg kunne komme og sige, at jeg ville teste disse teorier, og det, vi finder, er det her, så betød det faktisk noget - de vidste, hvad de fik".
Accepten fra de lokale aboriginere var langtfra en selvfølge. Selv det prestigefyldte Humangenom-projekt måtte opgive at få DNA fra aboriginere på grund af deres mistænksomhed over, om disse data ville kunne "udnyttes" som en slags "neokolonialisme". Aboriginernes mistænksomhed er så stor, at genetikeren Sheila van Holst Pellekaan ved Sydney universitet er stødt på lange forsinkelser med fortsat at få godkendelser til hendes genetiske studier af herkomst og sygdom blandt aborigin-australiere, og hun må kun dele begrænsede data med andre forskere. Hindringerne betyder, at aborigin- australiere, der som gruppe lider af forhøjet forekomst af diabetes og hjertesygdomme, kan gå glip af fremskridt inden for genetisk medicin (ref.9556)
Da artiklen blev frigivet fra Science og gik ud til medierne, tog Eske Willerslev igen til Australien og deltog i en pressekonference, hvor han inviterede to af Council-medlemmerne med. "Det var de også rigtig, rigtig glade for", fortæller han. "De sagde, at normalt, når de havde stødt på videnskabsfolk, så tager de bare, hvad de skal have, og så ser vi aldrig mere noget til dem. Her kom jeg ned, og de deltog ved præsentationen overfor pressen". Senere har Eske Willerslev inviteret to Council-medlemmer til Danmark, så de kan se, hvor arbejdet bliver lavet. Dette kan forhåbentlig tage noget af mystikken væk fra dette her DNA-sekvensbestemmelse, håber han.
"Vi har kørt dette studie fuldstændigt lovligt. Det er efter bogen - også efter australsk norm. Alligevel er der nogen, som brokker sig, og siger: "Jamen, vi skulle have spurgt nogle flere stammer". Og hvor er det så, de røster kommer fra? Det er fra kultur-antropologer samt jurister, som har specialiseret sig i at varetage disse folks interesser. Der har ikke været en eneste negativ røst fra en aboriginer. Derimod har vi fået email fra andre stammer, som siger: 'Kommer I ikke også over og laver studier hos os'. Det kan man så tænke lidt over. Hvem er det egentlig, der skaber problemerne?", siger Eske Willerslev.
Test dig selv
Du kan tegne abonnement på BioNyt: Videnskabens verden her!
/Homo/DNA/Genom/Aborigin/Det korte svar er: Har ikke haft gode erfaringer
Aboriginerne, urbefolkningen i Australien, har været meget mistænksomme mod DNA-sekvensbestemmelse og genom-forskning. Selv det prestigefyldte Humangenom-projekt måtte opgive at få DNA fra aboriginere på grund af deres mistænksomhed over, om disse data ville kunne "udnyttes" som en slags "neokolonialisme".
Aboriginernes mistænksomhed er så stor, at genetikeren Sheila van Holst Pellekaan ved Sydney universitet er stødt på lange forsinkelser med fortsat at få godkendelser til hendes genetiske studier af herkomst og sygdom blandt aborigin-australiere, og hun må kun dele begrænsede data med andre forskere. Hindringerne betyder, at aborigin-australiere, der som gruppe lider af forhøjet forekomst af diabetes og hjertesygdomme, kan gå glip af fremskridt inden for genetisk medicin (ref. 9556).
Eske Willerslev fortæller, at da han startede projektet havde han aldrig været i Australien, men blot læst på teorierne om, hvor aboriginerne kom fra. Han vidste ikke meget om alle de problemer, som aboriginerne har haft, og hvordan de er blevet behandlet.
For at sekvensbestemme genomet gik Eske Willerslev gennem de lovlige kanaler. Prøven var ejet af England, og det var lovligt i forhold til British Tissue Act, at man kunne udlevere en sådan hårprøve til sekvensbestemmelse.
Der findes ingen international standard for, hvilken forskning museernes arkæologiske samlinger må bruges til. Mange museer tilbagesender knogler, der er indsamlet fra indfødte stammer, men British Museum har den politik, at hår og negle ikke sendes retur.
Eske Willerslev gik også til Videnskabsetisk Komité i Danmark. De sagde: "Jamen, her er jo ingen problemer - det er et arkæologisk fund, og derudover er det blot demografiske studier, som I kikker på, så I behøver overhovedet ikke at få en godkendelse. Bare gå i gang". Den danske komité mente altså ikke, at det krævede en godkendelse i dette tilfælde, fordi der var tale om arkæologisk materiale.
I løbet af processen blev Eske Willerslev imidlertid opmærksom på, at det er et superfølsomt emne. Dette er ikke noget, som man bare gør. Aboriginerne er blevet rigtig dårligt behandlet. De bliver dårligt behandlet, og de er ikke mindst blevet dårligt behandlet af videnskabsfolk. Det gik op for Eske Willerslev, at selv om han rent lovmæssigt ikke behøvede det, så behøvede han rent moralsk en godkendelse fra de mennesker, det drejede sig om.
Problemet var så, hvordan man skulle gribe dette an. I Alfred Cort Haddons noter nævnes intet stammenavn, men ved at tale med en arkæolog fik Eske Willerslev at vide, at Australien er delt op i regioner, hvor lokalråd, "Council Boards", repræsenterer de lokale stammer. "Vi vidste jo, at denne person levede ved Golden Ridge". Det vil sige, at det mest naturlige var at sige: "Ok, vi vil prøve at opsøge det råd, som dækker alle de stammer, som Golden Ridge er en del af. Med meget stor sandsynlighed har man derved opsøgt de biologiske efterkommere af de stammer, som den unge mand kan have tilhørt. Lokalrådet Goldfields Land and Sea Council i Kalgoorlie, Western Australia, repræsenterer de ca. 5000 aborigin-australiere, der bor i det område, som Haddon havde hårprøven fra, og Eske Willerslev tog flyveren til Australien.
Det skal så siges, at på dette tidspunkt var de meget langt i processen. "Vi havde faktisk investeret et års arbejde af cirka 40 mennesker. Der var brugt flere millioner kroner til dette projekt. Eske Willerslev fik - upfront - at vide, at det ville blive et nej. Aboriginerne bryder sig ikke om DNA-forskning. Ingen kunne erindre, at der nogensinde på stammeniveau var givet tilladelse til at udføre DNA-studier. "Men der var ikke andet at gøre - jeg måtte derned. Det gjorde jeg så. Jeg sendte et brev i forvejen om, hvem jeg var, og hvad jeg ville, samt hovedresultaterne af projektet, som man kendte dem på dette tidspunkt - og så spurgte jeg, om jeg måtte komme ned og fremlægge min sag for dem. Det fik jeg så at vide, at det måtte jeg gerne. Og jeg fløj så hele vejen til Perth. Der kunne jeg ikke komme videre med fly, så vi måtte med bil - en ca. femten timers kørsel for at komme derud".
"Og så kommer jeg ind - og der må jeg sige, der havde jeg sved på panden - da jeg skulle ind til det møde, for jeg vidste jo, hvad der var på spil. Jeg vidste, at hvis de sagde nej til dette her, så var det end-of-story, altså. Så var alt det arbejde, vi havde lagt i det, det var færdigt. Vi ville aldrig gå ud og publicere det, og vi ville ikke gøre noget ved det."
Eske Willerslev fik hjælp fra Craig Muller, der har med forskning for Goldfield-rådet at gøre. Craig Muller havde undersøgt hårprøvens historie og konkluderet, at manden havde doneret sin hårlok frivilligt til Alfred Cort Haddon. Men denne arkæolog, der var sammen på rejsen med Eske Willerslev, sagde igen og igen: "Det kommer aldrig til at gå. De siger nej ".
"Men jeg skal love for, at jeg blev mødt af det fuldstændigt modsatte. Det første, de sagde til mig, var, 'Hvorfor har du ikke medbragt de der redskaber, så du også kan tage DNA-prøver af os?' Nå, tænkte jeg så. Jo, sagde en, 'jeg er ved at være gammel, så du skal huske at få en DNA-prøve af mig'. Og de sagde så, at 'vi synes det er et fantastisk studie, og det er rart, at der endelig er nogen, som kan lave noget ordentlig videnskab. Det kan de nemlig ikke finde ud af her i Australien. Kommer du ikke herover, og slår dig ned, og arbejder'. Det var helt uforståeligt, hvad der foregik".
"Jeg blev så inviteret ud i bushen, hvor de viste mig, hvordan man finder kænguruspor, jager osv. Jeg fandt ud af, at adskillige af medlemmerne af dette Council, hvor mange var midaldrende, og repræsenterede 13-14 stammer, havde fædre og mødre, som i virkeligheden var kommet ind fra bushen. Som barn var de blevet opdraget til at skulle fange kænguruer. De kan godt huske i dag, hvordan man fanger en kænguru, og hvilke bær og nødder, man kan spise".
Test dig selv
Du kan tegne abonnement på BioNyt: Videnskabens verden her!
/Homo/DNA/Genom/Aborigin/Det korte svar er: De fik en god forklaring, men også resultatet på forhånd
Mange har spurgt Eske Willerslev om, hvorfor han fik tilladelsen, når aboriginerne nu hidtil havde været så meget mod studier af deres genom. Eske Willerslev fik ikke alene en mundtlig accept, men kunne hjembringeet dokument, hvor det stod sort på hvidt med stempel og underskrevet af hele det lokale Council, at det her ønskede de blev publiceret, at det var frit tilgængeligt, og at der ikke var nogen restriktioner på det - man kan bruge det til al den videnskab, man ønsker. Jeg har talt med forskere fra Australien - de har i de sidste 10 år prøvet og kæmpet for at få et aboriginer-genom. Og så kommer jeg flyvende ind, tager genomet og flyver ud igen. Så de var rimeligt bitre. Det kan man roligt sige. De var faktisk rigtig bitre".
"Hvad er det så, der gør det? Jeg har ikke noget endeligt svar. Jeg tror, at der var nogle ting, som spillede en rolle - det fik jeg i hvert fald at vide. Det ene er, at jeg er dansker. Man kan sige, at som videnskabsmand plejer det altid at være rigtig godt at komme fra USA eller Amerika eller sådan noget. Harvard for eksempel, men København, det ved de ikke, hvad er. Danmark i det hele taget - super harmløst. Vi har ikke nogen kolonihistorie, som folk i hvert fald ude i verden bliver meget ophidset over. Og det betød noget. Det betød noget, at jeg ikke var australier, at jeg ikke var tysker og ikke var englænder." Og det betød noget, at jeg kom. De sagde til mig, at det havde de respekt for. At man flyver i 22 timer hver vej for at komme ned til et møde, der tager 1 time. For simpelthen at fremlægge min sag. Det betød virkelig meget."
"Og så vil jeg sige, så tror jeg faktisk, at en anden ting betød noget - det var, at jeg kunne fortælle dem, hvad resultatet var. Jeg ved godt, at hvis det skulle være gået fuldstændig efter bogen, så skulle jeg have været dernede og skulle have sagt, at jeg ville teste disse teorier. Men så havde jeg måske fået et nej. Det tror jeg i hvert fald. Men fordi jeg kunne komme og sige, at jeg ville teste disse teorier, og det, vi finder, er det her, så betød det faktisk noget - de vidste, hvad de fik".
Da artiklen blev frigivet fra Science og gik ud til medierne, tog Eske Willerslev igen til Australien og deltog i en pressekonference, hvor han inviterede to af Council-medlemmerne med. "Det var de også rigtig, rigtig glade for", fortæller han. "De sagde, at normalt, når de havde stødt på videnskabsfolk, så tager de bare, hvad de skal have, og så ser vi aldrig mere noget til dem. Her kom jeg ned, og de deltog ved præsentationen overfor pressen".
Senere har Eske Willerslev inviteret to Council-medlemmer til Danmark, så de kan se, hvor arbejdet bliver lavet. Dette kan forhåbentlig tage noget af mystikken væk fra dette her DNA-sekvensbestemmelse, håber han.
"Vi har kørt dette studie fuldstændigt lovligt. Det er efter bogen - også efter australsk norm. Alligevel er der nogen, som brokker sig, og siger: "Jamen, vi skulle have spurgt nogle flere stammer". Og hvor er det så, de røster kommer fra? Det er fra kultur-antropologer samt jurister, som har specialiseret sig i at varetage disse folks interesser. Der har ikke været en eneste negativ røst fra en aboriginer. Derimod har vi fået email fra andre stammer, som siger: 'Kommer I ikke også over og laver studier hos os'. Det kan man så tænke lidt over. Hvem er det egentlig, der skaber problemerne?", siger Eske Willerslev.
Test dig selv
Du kan tegne abonnement på BioNyt: Videnskabens verden her!
/Homo/DNA/Genom/Aborigin/Det korte svar er: Afrika eller mellemøsten
Nogle af teorierne om aboriginerne er så outdatede, at de ikke er værd at nævne i dag, såsom for eksempel, at de skulle være opstået helt uafhængigt af alting. "Den slags multiregional- teori holder simpelthen slet ikke, der er intet evidens for det", siger Eske Willerslev. "Men der har efter min mening været to teorier, som virkelig har været konkurrerende i de senere år", siger han.
Den teori, som i de senere år helt klart har været den mest dominerende, fordi den tilsyneladende har været understøttet af DNA-evidenser i form af mitokondrie-DNA og Y-kromosom-DNA, går ud på, at da det moderne menneske Homo sapiens spredte sig ud i verden fra Afrika, skete det kun én gang. Forskellige menneskegrupper poppede op undervejs fra den østgående bølge, og spredte sig ud over kontinentet - først europæerne, så asiaterne, og senere var der nogen inden for asiaternes gruppe, som vandrede ned i Australien, blev isoleret og udviklede sig til aboriginerne. Ifølge denne DNA-baserede en-bølge-teori er aboriginerne oprindelig asiater, ligesom indianerne stammer fra asiater. Det skønnede tidspunkt for den fælles forfader til europæerne og asiaterne (altså tidspunktet for adskillelsen mellem de europæiske og asiatiske forfædre)
Det stemmer imidlertid ikke overens med de arkæologiske beviser for en uafbrudt bosættelse i Australien siden meget tidlige tider. Hvis man skal tro de arkæologiske fund i Australien har aboriginerne været "for lang tid" i Australien i forhold til denne én-gangmigrationsbølge- teori. Ifølge arkæologiske fund har der været mennesker i Australien i mindst 44.000 år, og så kan aboriginerne ikke være opstået som udspaltning fra asiaterne, for europæerne og asiaterne blev spaltet ud fra hinanden for kun ca. 25.000 - 38.000 år siden, og hvis aboriginerne skulle være opstået fra asiaterne, må dette være sket senere. Hvis aboriginerne er spaltet ud fra asiaterne, så kan de ikke samtidig være direkte efterkommere af de første mennesker i Australien, og så ville aboriginerne ikke være "first nations", som de går rundt og siger. Dette er ret vigtigt - også politisk set for aboriginerne.
Der er heldigvis en anden teori. Den har hovedsagelig haft et arkæologisk fundament (men støttes nu af Eske Willerslev-gruppens genom-studie). Denne teori siger, at der har været to eller flere udvandringer fra Afrika, og at der i hvert fald har været flere ekspansioner østover ud i verden. Samt at der var en meget tidlig ekspansion, fra hvilken aboriginerne stammer. Den arkæologibaserede (nu genombaserede) flere-migrationsbølger-teori er blevet stort set fuldstændig latterliggjort. Man har sagt, at dette her er der overhovedet ikke noget i, og at man slet ikke kan finde noget genetisk evidens for det - indtil Eske Willerslevgruppen kom med deres genom. Ifølge denne teori opstod der fra denne tidlige ekspansion forskellige befolkningsgrupper, bl.a. aboriginerne og de såkaldte Negrito-grupper. (Se: "Hvad er nigrito-befolkningsgrupper?")
Et genom-studie af aborigineren kunne give en forklaring, og da Eske Willerslevs gruppe fra Københavns Universitet publicerede deres resultat i Science 22. september 2011, blev det da også bragt på forsiden som en nyfortolkning af menneskets forhistorie. Aborigin-studiet demonstrerede desuden, at hår, der har været opbevaret under dårlige forhold, alligevel kan bruges til genom-sekvensbestemmelse uden risiko for forurening fra nutidens mennesker (som det ellers ofte ses ved forsøg på DNA-studier af gamle fund af tænder og knogler). Fremover vil forskere derfor kunne studere den genetiske historie af mange oprindelige folk verden over ved at studere museum- samlinger samt samarbejde med grupper af efterkommere (også når flere befolkningsgrupper er iblandet).
61% af det DNA, som de fandt i hårlokken, var aborigin-DNA. Der sås ingen tegn på europæisk iblanding i aborigin-genomet. De vurderede forureningen til at være under 0,5%. Meget af bakterie-DNA'et var i øvrigt acne, så det var altså hyppigt i hovedbunden hos disse aboriginere.
Test dig selv
Du kan tegne abonnement på BioNyt: Videnskabens verden her!
/Homo/DNA/Genom/Aborigin/Det korte svar er: Efterkommere efter den første bølge ud fra Afrika eller mellemøsten
Ordet "negrito" kommer fra spansk og betyder "lille neger", hvilket hentyder til deres lave højde, under 155 cm. Man har kaldt dem pygmæer, men genetiske studier viser, at negritoer kun er fjernt beslægtede med de afrikanske pygmæer. Den lave vækst kan også tænkes at være en senere tilpasning til tropisk regnskov eller tilpasning til at klare sig ved begrænsede fødekilder.
De ligner afrikanere og findes forskellige steder i sydøstasien, bl.a. mørke folkeslag på fem Andaman-øer (Great Andamanese, Jangil, Jarawa, Onge og Sentinelese), seks Semang-befolkningsgrupper i Malaysia, Mani-folket i Thailand, samt Aeta-, Agta-, Ati-folket og 30 andre befolkningsgrupper på Philippinerne. Der skal også have været nigrito-befolkningsgrupper på Sarawak, Borneo og måske i Taiwan, hvor der stadig findes en festival, som kaldes "Ritual for det lille sorte folk" (ref.9563).
Antropologen Jared Diamond har i sin bog "Vejen til verden af i dag" ("Guns, Germs and Steel"), skrevet, at han forestiller sig, at negritoerne er mulige forfædre til aborigin-australierne og papuan-folket på New Guinea.
Test dig selv
Du kan tegne abonnement på BioNyt: Videnskabens verden her!
/Homo/DNA/Genom/Aborigin/Det korte svar er: Mindre forurenet, idet det lettere renses på overfladen
Aborigin-studiet demonstrerede, at hår, der har været opbevaret under dårlige forhold, alligevel kan bruges til genom-sekvensbestemmelse uden risiko for forurening fra nutidens mennesker (som det ellers ofte ses ved forsøg på DNA-studier af gamle fund af tænder og knogler). Fremover vil forskere derfor kunne studere den genetiske historie af mange oprindelige folk verden over ved at studere museum-samlinger samt samarbejde med grupper af efterkommere (også når flere befolkningsgrupper har blandet sig).
Test dig selv
Du kan tegne abonnement på BioNyt: Videnskabens verden her!
/Homo/DNA/Genom/Aborigin/Det korte svar er: 61%
61% af det DNA, som de fandt i hårlokken, var aborigin-DNA. Der sås ingen tegn på europæisk iblanding i aborigin-genomet. De vurderede forureningen til at være under 0,5%.
Test dig selv
Du kan tegne abonnement på BioNyt: Videnskabens verden her!
/Homo/DNA/Genom/Aborigin/Det korte svar er: Acne
Meget af bakterie-DNA'et var acne, så det var altså hyppigt i hovedbunden hos disse aboriginere.
Test dig selv
Du kan tegne abonnement på BioNyt: Videnskabens verden her!
/Homo/DNA/Genom/Aborigin/Det korte svar er: 60-70 basepar
Det meste DNA fra den 90 år gamle hårprøve af en aboriginer fra Sydvest-australien var fragmenteret ned til typisk kun 60-70 basepar. Hvis man til sammenligning tager DNA fra et nutidsmenneske, vil fragmenterne være fra 50 mill. til 250 mill. lange basekæder (ref.9576).
Test dig selv
Du kan tegne abonnement på BioNyt: Videnskabens verden her!
/Homo/DNA/Genom/Aborigin/Det korte svar er: Benny
Eske Willerslev giver altid sine DNA-sekvensbestemte prøver navne, så man kan tale hurtigt med hinanden på laboratoriet. Aborigineren kaldte Eske Willerslev "Benny".
Test dig selv
Du kan tegne abonnement på BioNyt: Videnskabens verden her!
/Homo/DNA/Genom/Aborigin/Det korte svar er: Forskernes navn til det første aboriginer-genom
Eske Willerslev giver altid sine DNA-sekvensbestemte prøver navne, så man kan tale hurtigt med hinanden på laboratoriet. Aborigineren, hvorfra man genom-bestemte DNA'et i en 90 år gammel hårprøve, kaldte han "Benny".
Test dig selv
Du kan tegne abonnement på BioNyt: Videnskabens verden her!
/Homo/DNA/Genom/Aborigin/Det korte svar er: Ingen overraskelser
På "Bennys" (den genom-sekventerede aboriginer fra 1923) mitokondrie- DNA og Y-kromosom var der ingen overraskelser. Det var af den slags, som man typisk finder i Australien og med samme grundstamme som mitokondrie-DNA og Y-kromosom-DNA, som man finder hos europæere og asiater.
Med grundstamme menes, at da de udvandrede fra Afrika havde de en bestemt type mitokondrie- DNA.
Test dig selv
Du kan tegne abonnement på BioNyt: Videnskabens verden her!
/Homo/DNA/Genom/Aborigin/Det korte svar er: bl.a. PCA-plot
Genom-sekvensen fra aboriginer-håret blev underkastet forskellige test, bl.a. en PCA-plot - hvor man sammenlignede aboriginerens DNA med DNA'et hos andre mennesker, både fra Afrika, Europa og Asien. På et PCA-plot lignede aborigineren lidt asiater og de amerikanske indianere (som er asiater, der er rejst til Amerika og blevet isoleret der). Så ved en umiddelbar betragtning lignede aborigineren en asiat, som på et eller andet tidspunkt er blevet isoleret i Australien - altså svarende til indianerne.
Test dig selv
Du kan tegne abonnement på BioNyt: Videnskabens verden her!
/Homo/DNA/Genom/Aborigin/Det korte svar er: Benny og asiater har visse ting tilfælles i genomet
Ved en umiddelbar betragtning ligner aborigin-DNA en asiat, som på et eller andet tidspunkt er blevet isoleret i Australien svarende til at indianerne blev isoleret i Amerika. Men det eneste, som dette viser , er, at det kun viser ligheden: Det viser ikke, hvordan denne lighed er opstået. Det viser ikke, hvordan det kan være, at Benny (det navn forskerne gav aborigineren fra 1923) ligner en asiat mere end Benny ligner en europæer. Er det, fordi han stammer fra asiater? Er det, fordi hans forfædre er spaltet ud fra asiaterne? Eller kunne der være en helt anden forklaring? Kunne det være et genflow, som ifølge den såkaldte "arkæologi-teori", således at der har været en udveksling af DNA mellem asiaternes forfædre og aboriginernes forfædre, hvorved asiaterne er kommet til at indeholde aboriginforfader-DNA? Det ville også give et sådant resultat med hensyn til lighed".
Mere omfattende og komplekse analyser var derfor nødvendige.
Test dig selv
Du kan tegne abonnement på BioNyt: Videnskabens verden her!
/Homo/DNA/Genom/Aborigin/Det korte svar er: For ca. 70.000 år siden
Meget veletablerede proportionsmodeller, der er baseret på kendte genomer viser, at for ca. 70.000 år siden spaltede aboriginerne ud og isolerede sig fra resten af Homo sapiens. Og ca. 24.000 år senere (altså for ca. 46.000 år siden) spaltede europæerne og asiaterne sig væk fra hinanden.
Test dig selv
Du kan tegne abonnement på BioNyt: Videnskabens verden her!
/Homo/DNA/Genom/Aborigin/Det korte svar er: Ja, der tyder ny forskning på
Formentlig var aboriginerne (indtil deres opblanding med europæerne i nyere tid) verdens mest direkte efterkommere efter denne tidlige udvandringsbølge, idet formentlig næsten alle de folkeslag, som ikke nåede helt til det isolerede Australien, efterhånden blev opblandet med senere udvandringsbølger.
Test dig selv
Du kan tegne abonnement på BioNyt: Videnskabens verden her!
/Homo/DNA/Genom/Aborigin/Det korte svar er: Negrito-grupperne
Der er næsten ikke nogen folkeslag tilbage med kruset hår i Asien. Kun Negrito-befolkningsgrupperne og aboriginerne i Australien, har kruset hår. Alle steder, undtagen i Australien, forsvinder disse træk, fordi de bliver opblandet. Aborigin-australierne er meget mørke i huden. Det afspejler deres oprindelse i Afrika, samt at de har vandret og levet i et område nær ækvator. Europæerne og asiaterne fik en lysere hud, som bedre kan optage sollys, der fører det dannelse af det meget vigtige vitamin-D.
Test dig selv
Du kan tegne abonnement på BioNyt: Videnskabens verden her!
/Homo/DNA/Genom/Aborigin/Det korte svar er: Får europæisk udseende
Selv om aboriginerne og europæerne er adskilt af tusinder af generationer, har de let ved at få børn sammen. Børnene kommer efter få generationer til at få et ret europæisk udseende (ref.9557).
Test dig selv
Du kan tegne abonnement på BioNyt: Videnskabens verden her!
/Homo/DNA/Genom/Aborigin/Det korte svar er: Ja, der tyder ny forskning på
Eske Willerslevs gruppe fra Københavns Universitet på Københavns Universitet fandt et resultat, der er i overensstemmelse med, at aboriginerne er direkte efterkommere efter de første mennesker i Australien. Aboriginerne kan være efterkommere af de første opdagelsesrejsende blandt Homo sapiens. Aboriginerne var før europæer-tiden den ældste, kontinuerlige menneskepopulation uden for Afrika (ref.9525), og måske den gruppe, som har den længste tilknytning til det land, de lever i den dag i dag. De er direkte efterkommere af de første mennesker, der kom til Australien, og har haft mere.
Test dig selv
Du kan tegne abonnement på BioNyt: Videnskabens verden her!
/Homo/DNA/Genom/Aborigin/Det korte svar er: Langs kysten via Indien
Vandringsvejen til Australien ses bl.a. af de over 50.000 år gamle stenredskaber, der er fundet i Indien og andre steder. Det tyder som forventet på en migration via Indien og Sydøstasien.
Test dig selv
Du kan tegne abonnement på BioNyt: Videnskabens verden her!
/Homo/DNA/Genom/Aborigin/Det korte svar er: 44000 år gamle, simple stenredskaber
De tidlige stenredskaber, der er blevet fundet i Australien (44.000 år gamle (ref.9558), er meget simplere end stenredskaberne fra den øvre del af ældre stenalder i Europa fra samme tid (og som må være fra den tidligste indvandring til Europa).
Test dig selv
Du kan tegne abonnement på BioNyt: Videnskabens verden her!
/Homo/DNA/Genom/Aborigin/Det korte svar er: For 44.000 år siden eller tidligere
Hvornår de ankom vides ikke - det var mindst for 44000 år siden, men kan være sket meget tidligere, for da havde de haft ca. 26.000 år til at gøre rejsen fra Afrika/Mellemøsten, De tidligste stenredskaber, der er blevet fundet i Australien (44.000 år gamle (ref.9558), er meget simplere end stenredskaberne fra den øvre del af ældre stenalder i Europa fra samme tid (og som må være fra den tidligste indvandring til Europa).
Test dig selv
Du kan tegne abonnement på BioNyt: Videnskabens verden her!
/Homo/DNA/Genom/Aborigin/Det korte svar er: Via en kortere rejse over havet, da Australien var tæt på Asien via øgrupper
De første indbyggere i Australien må have haft viden om at bygge avancerede både til at krydse fra det nærmeste punkt i Asien til Sahul, det gamle kontinent, hvori Australien/ New Guinea/Tasmanien indgik indtil havniveau-stigningen ved slutningen af den sidste istid for 10.000 år siden (se fig.). Der er ingen arkæologiske beviser for sådanne aborigin-både (ref.9558).
Der var dog talrige øbroer på vej til Australien, for havet var sunket og delvis blevet til is ved polerne. Men der var dog åbne vandstrækninger på op til 90 km (ref.9650s.98). Rejsen gik delvis via Andaman-øerne (hvor indbyggerne i dag har afrikansk mitokondrie- DNA), og disse øer har nok været landfaste dengang (ref.9650s.98).
Test dig selv
Du kan tegne abonnement på BioNyt: Videnskabens verden her!
/Homo/DNA/Genom/Aborigin/Det korte svar er: Uvist
Hvornår de ankom vides ikke. Man har gættet på en marchhastighed mod øst på 2 km pr. år, så rejsen til Australien kan have taget 5000 - 6000 år (ref.9650s.903). De kan måske have været fremme for 60.000 år siden. Da aboriginernes forfædre ankom til Australien var dette landfast med New Guinea - men kun frem til for 8000 år siden, så efter dette tidspunkt var urbefolkningerne på Australien og New Guinea formentlig uden kontakt med hinanden (ref.9650s.114).
Test dig selv
Du kan tegne abonnement på BioNyt: Videnskabens verden her!
/Homo/DNA/Genom/Aborigin/Det korte svar er: De var isolerede, men der må have været kontakter
Det er stadig uklart, hvor stor grad af isolation, der har været, efter at aboriginerne ankom til Australien. Der har været spekulationer, om der i Australien har været forskellige grupper af aboriginere, som har blandet sig. Hos skeletter af tidlige australierne finder man i hvert fald to typer, hvor nogle har mere robuste kranier, og andre har mere spinkle kranier. Der er blevet foreslået mange ting, f.eks. at det repræsenterer to forskellige indvandringer, eller bare almindelig variation - eller måske blot mænd og kvinder. Eske Willerslev har fået tilladelser til at lave DNA-studier af disse skeletter og skal samarbejde med prof. David Lambert fra Griffith universitet i Australien, der forsker i gammel-DNA, molekylær evolution, evolutionær biologi og evolutionær teori. Der er taget prøver af disse forskellige skeletter. Når man slår på dem, lyder det som porcelæn, hvilket ikke er så lovende, men måske kan man også her finde noget DNA.
På trods af aboriginernes genetiske isolation, er der tegn på væsentlige kulturelle udvekslinger. Der skete noget bemærkelsesværdigt i Australien for 6.000 - 4.000 år siden. Stenredskaberne blev mere sofistikerede, og befolkningstallet øgedes. Man mangler forklaring på disse kulturelle ændringer, der begyndte på tidspunktet for dingoens ankomst eller kort derefter. Aboriginerne har ikke dyrket planter eller tæmmet dyr, men den vilde hund, dingoen, dukkede op i de arkæologiske fund fra dette tidspunkt. Aboriginaustralierne synes på en eller anden måde at have beholdt Australien for sig selv igennem ca. 50.000 år (ref.9558). Men dingoen tyder på, at folk sivede til Australien på forskellige tidspunkter.
Test dig selv
Du kan tegne abonnement på BioNyt: Videnskabens verden her!
/Homo/DNA/Genom/Aborigin/Det korte svar er: De var isolerede, men der må have været kontakter
Aborigin-australierne synes på en eller anden måde at have beholdt Australien for sig selv igennem ca. 50.000 år (ref.9558). Men Alan Redd, der forsker i biologisk antropologi ved University of Kansas i Lawrence, mener dog, at indvandringen til Australien kan have været kompliceret. F.eks. blev dingoen, den vilde hund, bragt til Australien af mennesker, der ankom inden for de sidste ca. 5.000 år. “Det er helt sikkert muligt, at folk sivede til Australien", mener han.
Test dig selv
Du kan tegne abonnement på BioNyt: Videnskabens verden her!
/Homo/DNA/Genom/Aborigin/Det korte svar er: Intet landbrug
Aboriginerne har ikke dyrket planter eller tæmmet dyr.
Test dig selv
Du kan tegne abonnement på BioNyt: Videnskabens verden her!
/Homo/DNA/Genom/Aborigin/Hund/Dingo/Det korte svar er: En vild hund
Dingoen er en vild hund. Forskere under ledelse af Peter Savolainen fra det kongelige teknologiske institut i Stockholm har sporet spredningen af dingoen på tværs af øerne i Stillehavet ved at analysere gammelt DNA i knoglerne i polynesiske hunde (ref.9778, Proceedings of the Royal Society B).
Test dig selv
Du kan tegne abonnement på BioNyt: Videnskabens verden her!
/Homo/DNA/Genom/Aborigin/Hund/Dingo/Det korte svar er: I Asien
Dingoen opstod på det asiatiske fastland og blev en del af de polynesiske husdyr sammen med gris, høns - og rotter. Denne påvirkning nåede New Zealand ca. år 1250, bortset fra dingoen, der ikke nåede til New Zealand. Derimod er det en gåde, hvordan dingoen ankom til Australien, fordi ingen af de andre elementer i den polynesiske kultur findes i Australien. Og man har endnu ikke fundet tegn på polynesiske gener i aborigin-befolkningen.
Test dig selv
Du kan tegne abonnement på BioNyt: Videnskabens verden her!
/Homo/DNA/Genom/Aborigin/Test/D/Det korte svar er: Statistisk test til påvisning af beslægtethed
Den første test, som blev lavet af Benny's DNA (DNA fra aboriginer-hårprøven fra 1923) var en såkaldt D-test. D-testen blev brugt på SNP-chipdata og genomer til at lede efter fælles forfædre mellem aborigin-australiere og andre befolkningsgrupper. D-testen afprøver 2 simple teorier:
Hvis man spørger D-testen: "Er aboriginerne gået ud af Afrika før asiatere og europæere?" svarer D-testen: "Nej, det er ikke nødvendigvis rigtigt".
Hvis man så spørger D-testen: "Er aboriginerne gået ud af Afrika sammen med asiatere?" svarer D-testen: "Nej, det er heller ikke rigtigt".
Ifølge D-testen er der derfor ingen af de mest simple teorier, der er rigtige. Vores forklaring er så, forklarer Eske Willerslev, at når de første mennesker er gået fra Afrika til Australien, så har de jo efterladt populationer hele vejen. Så når den næste bølge kommer, kan den sagtens have blandet sig. Men dem, der er gået op i Europa, har ikke blandet sig. Derfor vil folk i Asien se ud til at være lidt tættere beslægtet med aboriginerne, end med folk i Europa. Så det, at aboriginerne med en test ligner asiaterne mere, behøver ikke at betyde, at de stammer derfra. D-testen bruges til at vise, at ingen af de to helt simple forklaringer er korrekt - det er mere komplekst.
"Man tager f.eks. tre genomer, og så bruger man D-testen til at finde ud af, hvem af de to genomer, der er tættest på Benny. Det viser sig så, at ifølge D-testen kan ingen af de to teorier være rigtig, medmindre der er sket et genflow mellem populationerne.
Den arkæologi-baserede flere-migrationsbølger-teori kan ifølge D-testen kun være rigtig, hvis der efterfølgende er sket en masse genflow mellem Benny'erne (aboriginerne) og asiatere - og det er faktisk netop, hvad man ville forvente ifølge denne teori
Vi brugte D-testen på SNP-chipdata og genomer til at lede efter fælles forfædre mellem aborigin-australiere og andre befolkningsgrupper." D-testen afprøver 2 simple teorier: 1) Hvis man spørger D-testen: "Er aboriginerne gået ud af Afrika sammen med asiatere?" svarer D-testen: "Nej, det er ikke rigtigt". Hvis man spørger D-testen: "Er aboriginerne gået ud af Afrika før asiatere og europæere?" svarer D-testen: "Nej, det er ikke præcis rigtigt". Ifølge D-testen er der derfor ingen af de mest simple teorier, der er rigtige. Vores forklaring er så, at når de første mennesker er gået fra Afrika til Australien, så har de jo efterladt populationer hele vejen. Så når den næste bølge kommer, kan den sagtens have blandet sig. Men dem, der er gået op i Europa, har ikke blandet sig. Derfor vil folk i Asien se ud til at være lidt tættere beslægtet med aboriginerne, end med folk i Europa. Så det, at aboriginerne med en test ligner asiaterne mere, behøver ikke at betyde, at de stammer derfra. D-testen bruges til at vise, at ingen af de to helt simple forklaringer er korrekt - det er mere komplekst. "Man tager f.eks. tre genomer, og så bruger man D-testen til at finde ud af, hvem af de tre genomer, der er tættest på Benny. Det viser sig så, at ifølge D-testen kan ingen af de to teorier være rigtig, medmindre der er sket et genflow mellem populationerne. Den arkæologi-baserede flere-migrationsbølger- teori kan ifølge D-testen kun være rigtig, hvis der efterfølgende er sket en masse genflow mellem Benny'erne (aboriginerne) og asiatere - og det er faktisk netop, hvad man ville forvente ifølge denne teori.
Eske Willerslevs gruppe fra Københavns Universitet supplerede med en såkaldt D4P-test, som er tæt forbundet med D-testen.
Test dig selv
Du kan tegne abonnement på BioNyt: Videnskabens verden her!
/Homo/DNA/Genom/Aborigin/Test/D4P/Det korte svar er: Test hvormed man kan afprøve forskellige scenarier for DNA-slægtskab
Eske Willerslevs gruppe fra Københavns Universitet på Københavns Universitet supplerede studiet af aborigin-genomet med en såkaldt D4P-test, som er tæt forbundet med D-testen. Ved D4P-testen sætter man et scenarie op, hvor man spørger: Hvor mange positioner i genomet støtter en situation, hvor asiatere og aboriginere er tættest på hinanden, og hvor europæere og afrikanere er tættest på hinanden? Man kan altså tælle antallet af positioner, og så gå ind og lave simuleringer om, hvilke teorier, der passer bedst. Er det den teori, hvor der er to migrationsbølger med efterfølgende genflow mellem asiater og Benny, eller er det den nævnte én-gang-migrationsbølge-teori. Det, som man ser hver eneste gang, er at det, som passer bedst til dataene, er dobbelt-ekspansionsteorien: Altså at Benny'erne (aboriginerne) vandrer ud og spalter ud tidligere".
Test dig selv
Du kan tegne abonnement på BioNyt: Videnskabens verden her!
/Homo/DNA/Genom/Aborigin/Test/D4P/Det korte svar er: Slægtskab mellem genom-grupper
Ved D4P-testen (af aborigin-genomet) sætter man et scenarie op, hvor man spørger: Hvor mange positioner i genomet støtter en situation, hvor asiatere og aboriginere er tættest på hinanden, og hvor europæere og afrikanere er tættest på hinanden? Man kan altså tælle antallet af positioner, og så gå ind og lave simuleringer om, hvilke teorier, der passer bedst. Er det den teori, hvor der er to migrationsbølger med efterfølgende genflow mellem asiater og Benny, eller er det den nævnte én-gangmigrationsbølge- teori. Det, som man ser hver eneste gang, er at det, som passer bedst til dataene, er dobbelt-ekspansionsteorien: Altså at aboriginerne vandrer ud og spalter ud tidligere".
Til sidst tog vi nogle meget veletablerede proportionsmodeller, der er baseret på kendte genomer, og spurgte: Hvor passer Benny bedst? Og hver gang spaltede Benny ud tidligere end europæerne og asiaterne, hvilket passer til flere-migrationsbølger-teorien. De nævnte proportionsmodeller kan også svare på spørgsmålet: Hvor mange generationer siden er det, at Benny spaltede ud? Hvis man f.eks. siger, at generationstiden er 35 år, eller 25 år, så får man et tal som svar. Tallet viser, at for ca. 70.000 år siden (62-75.000 år siden) spaltede aboriginerne ud og isolerede sig fra resten af Homo sapiens. Og at det var mindst ca. 24.000 år senere (for omkr. 25-38.000 år siden), at europæerne og asiaterne spaltede sig væk fra hinanden." Forskerne fandt tegn på, at asiaterne havde optaget DNA (ved genflow) fra den første udvandringsbølge, som indeholdt aborigin-forfædrenes DNA, - og at dette genflow mellem befolkningsgrupperne i de to spredningsbølger skete forud for udspaltningen til de indfødte indianer-amerikanere fra disses moderne asiatiske forfædre (ref.9566).
Test dig selv
Du kan tegne abonnement på BioNyt: Videnskabens verden her!
/Homo/DNA/Genom/Aborigin/Test/D4P/Det korte svar er: Test for at finde hvor en genomgruppe passer bedst slægtsmæssigt
Proportionsmodeller er baseret på kendte genomer og kan bruges til at udregne antallet af generationer, siden noget skete - altså svare på spørgsmålet: Hvor mange generationer siden er det, at "Benny" (aborigin-genomet) spaltede ud? Og hvor mange generationer er det siden, at asiaterne og europæerne spaltede væk fra hinanden? Hvis man f.eks. siger, at generationstiden er 35 år, eller 25 år, så får man et tal som svar. Proportionsmodeller kan også svare på "Hvor passer Benny bedst?" Benny's DNA-linie viste, at hans forfædre spaltede ud tidligere fra de oprindelige (afrikanske) linier end europæerne og asiaterne, hvilket passer til den arkæologibaserede "flere-migrationsbølger"-teori.
Test dig selv
Du kan tegne abonnement på BioNyt: Videnskabens verden her!
/Homo/DNA/Genom/Aborigin/Test/PCA/Det korte svar er: At "Benny" og asiater har visse ting tilfælles i genomet
På et såkaldt PCA-plot sammenlignedes Benny's DNA med DNA'et hos andre mennesker, både fra Afrika, Europa og Asien. På et PCA-plot ligner Benny lidt asiater og de amerikanske indianere (som er asiater, der er rejst til Amerika og blevet isoleret der). Så ved en umiddelbar betragtning ligner Benny en asiat, som på et eller andet tidspunkt er blevet isoleret i Australien - altså svarende til indianerne. (PCA-analysen kan bruges til at besvare spørgsmålet: "Hvis man prøver at gøre det simpelt, hvor er der så mest variation?" PCA-analysen er derfor god til at adskille meget komplekse og store datasæt).
Test dig selv
Du kan tegne abonnement på BioNyt: Videnskabens verden her!
/Homo/DNA/Genom/Aborigin/Test/PCA/Det korte svar er: Hvor der er mest variation
PCA-analysen kan bruges til at besvare spørgsmålet: Hvis man prøver at gøre det simpelt, hvor er der så mest variation? PCA-analysen er derfor god til at adskille meget komplekse og store datasæt) .
Test dig selv
Du kan tegne abonnement på BioNyt: Videnskabens verden her!
/Homo/DNA/Genom/Aborigin/Test/PCA/Det korte svar er: Det er ikke tilstrækkeligt
På et PCA-plot lignede aborigineren lidt asiater og de amerikanske indianere (som er asiater, der er rejst til Amerika og blevet isoleret der). Så ved en umiddelbar betragtning lignede aborigineren en asiat, som på et eller andet tidspunkt er blevet isoleret i Australien - altså svarende til indianerne. Men det viser ikke, hvordan denne lighed er opstået. Det viser ikke, hvordan det kan være, at Benny ligner en asiat mere end Benny ligner en europæer. Er det, fordi han stammer fra asiater? Er det, fordi hans forfædre er spaltet ud fra asiaterne? Eller kunne der være en helt anden forklaring? Kunne det være et genflow , som ifølge den såkaldte "arkæologi-teori", således at der har været en udveksling af DNA mellem asiaternes forfædre og aboriginernes forfædre, hvorved asiaterne er kommet til at indeholde aboriginforfader- DNA? Det ville også give et sådant resultat med hensyn til lighed". "Det er altså vigtigt, at man ikke stopper her - som de fleste gør - og det gjorde vi altså ikke", siger Eske Willerslev. "Vi begyndte på meget omfattende og komplekse analyser, bl.a. en "D-test".
Test dig selv
Du kan tegne abonnement på BioNyt: Videnskabens verden her!
/Homo/DNA/Genom/Aborigin/Test/PCA/Det korte svar er: Test, der bl.a. bruges når geners aktivitet (ekspression) sammenlignes i form af pletter på geler
PCA-plot (Principal Component Analysis) bruges til forskellige studier. Blandt andet til "spot-ekspression-niveauer" i geler, hvor geners aktivitet (ekspression) sammenlignes i form af pletter på geler. Afbildning af ekspressionsdata giver mulighed for at separere gelprøverne efter deres ekspression-variation. Dette er nyttigt til at identificere geler, der har forskellige egenskaber i forhold til andre geler (“gel outliers”).
Hvis man i et simpelt eksperiment bruger 2 geler og har 15 pletter på hver gel, så kan man afbilde pletterne i en 2-dimensionel graf. Det første skridt i PCA-metoden er at tegne en ny akse, der repræsenterer retningen af den maksimale variation i de fundne data (det vil sige den retning, som gør rede for og står for så meget af variabiliteten i data-sættet som muligt). Dette kaldes “den første principale komponent” (the first principal component).
Dernæst bliver en anden akse tilføjet, men vinkelret på den første, dvs. ukorreleret med den første. (De ukorrelerede variable kaldes altså “principale komponenter” (“hovedkomponenter”) i denne matematiske procedure (ref.9564).ref.9565). Den anden akse bliver placeret, så den kommer til at repræsentere den næsthøjeste variation af de fundne data. Dette kaldes “den anden principale komponent” (the second principal component).
Dataene transformeres derefter (roteres), så man kan se punkterne på den nye akse i grafen. Naturligvis ville dette være temmelig meningsløst, med kun 2 geler, da vores hjerner nemt kan se relationerne mellem punkterne i et to-dimensionelt rum. Men med 3 geler afbildes punkterne i et 3-dimensionelt rum, og med 4 geler afbildes punkterne i et 4-dimensionelt rum, og så videre. I disse tilfælde fortsætter processen med at tilføje flere “principale” komponenter, som hver gang afsættes vinkelret på den foregående, og som hver omfatter mindre og mindre af variansen i datasættet.
Resultatet af dette er, at man kan visualisere pletter (og geler) i to- eller tre-dimensionelle rum på en sådan måde, at pletter, der er “tæt sammen” (dvs. som ikke viser megen variation) vil blive vist sammen på et PCA-plot og omvendt. Ved at afbilde geler såvel som pletter på den samme graf (kaldet et “biplot”), bliver det lettere at se, hvilke pletter (spots), der bidrager til forskellen mellem gelerne. Dette kan så bruges til at afgøre, hvilke pletter, der er vigtigst for at skelne en bestemt gel eller en gruppe fra de andre geler eller grupper.
Man kan afbilde både transformede plet-data og gel-data på et biplot. Biplot’et indeholder en masse oplysninger og kan være nyttig i forbindelse med fortolkningen af relationer mellem grupper og pletter. Desuden kan det hjælpe til at identificere “outlier-geler”, dvs. geler, der har forskellige egenskaber i forhold til andre geler i de samme grupper.
Test dig selv
Du kan tegne abonnement på BioNyt: Videnskabens verden her!
/Homo/DNA/Genom/Inuit/Det korte svar er: Nej
Det var ikke inuitter, der først kom til Grønland. Det var heller ikke indianere. Derimod var det folk fra Sibirien, som krydsede Beringstrædet og via Alaska og Canada som de første nåede Grønland. Vandringen startede for ca. 5500 år sidenref.9550). Saqqaq-manden for 4000 år siden er i dag tættest beslægtet med folk, som nu 200 generationer senere lever i Sibirien. Genomet viser, at manden var gene-tisk tilpasset til at leve i kulden. Eske Willerslev havde brune øjne, brun hud, skovlformede fortænder, tørt ørevoks (hvilket øger risikoen for øreinfektioner), tendens til skaldethed (og da der jo var tale om en ret stor hårtot, var Eske Willerslev derfor sikkert ret ung, da Eske Willerslev døde). Saqqaq-kulturen eksisterede i ca. 1700 år i Grønland. Den uddøde, måske ca. 800 år før vor tidsregning. Håret og 4 knoglestykker er de ene-ste vævsrester, som man har fundet af disse mennesker fra de 1700 år. Det var ikke Columbus, der opdagede Amerika. Det er sket mange gange, bl.a. for 14500 og for 5500 år siden (ref.9552).
Test dig selv
Du kan tegne abonnement på BioNyt: Videnskabens verden her!
/Homo/DNA/Genom/Inuit/Det korte svar er: En kultur, der levede fra ca. år -2500 til ca. år -800 i Grønland
De palæoeskimoiske kulturer, som kaldes Saqqaq-kulturen og Independence-I-kulturen, kendes fra arkæologiske fund i det nordlige Canada og Grønland. Deres oprindelse og genetiske relationer til senere kulturer er ukendt. Saqqaq-kulturen eksisterede i ca. 1700 år i Grønland (fra ca. år -2500 til ca. år -800). Den uddøde, måske ca. 800 år før vor tidsregning. Håret og 4 knoglestykker er de eneste menneskevævsrester, som man har fundet fra de 1700 år.
Test dig selv
Du kan tegne abonnement på BioNyt: Videnskabens verden her!
/Homo/DNA/Genom/Inuit/Det korte svar er: 4000-5000 nordboere
Gården under sandet kaldes de rester efter en nordbo-gård, som man fandt velbevaret af permafrost. Gården blev forladt i ro og orden, men dyrene var efterladt, eftersom man fandt rester af en ged under en sammenfaldet mur. DNA-studier har kunnet påvise den senere historie på stedet med hensyn til husdyrenes fortsatte tilstedeværelse på stedet. Kirkegård-udgravninger tyder på, at der i Grønland boede 4000-5000 nordboere, de fleste i Østerbygden, på over 300 gårde. Man har fundet 22 kirker, et nonnekloster, og mange kirkegårde. Nordbosamfundet blomstrede med institutioner og kultur og forblev europæisk, og opførelsen af kunstfærdige kirker viser, at den kristne tro nok har vokset sig stærkere. Da Hans Egede ankom til Grønland i 1721 var nordbosamfundet imidlertid forlængst uddød ved gradvis tilbagegang som følge af klimatisk afkøling, overbefolkning og økonomisk stress, mener man (ref.9642).
Den 27. maj 2011 forsvarede Eske Willerslev sin ph.d-afhandling om “anvendelser af 2.-generationssekventering til studier af gammelt DNA”).
Test dig selv
Du kan tegne abonnement på BioNyt: Videnskabens verden her!
/Homo/DNA/Genom/Inuit/Det korte svar er: Fra en hårtot på Nationalmuseet i København af en forhistorisk mand fra Arktis
Eske Willerslev har i modsætning til andre DNA-forskere en baggrund, hvor det ikke afskrækker ham at gå langt for at finde velbevaret, gammelt DNA, - og han går gerne langt i jagten på uberørt DNA, skriver Nature. Det bedste bevaringssted er is, hvor DNA er bevaret af kulden og bedst beskyttet mod forurening. Eske Willerslev ønskede DNA fra et fortidsmenneske i arktis, f.eks. noget hår, et ben, en tand. Med dette for øje fløj han i 2006 med sin gruppe til Grønlands mest øde, nordlige område, til den militære Station Nord, blot for at erfare, at helikopteren, der skulle flyve hans gruppe til et arkæologisk interessant sted, var styrtet ned i en storm kort forinden. Han fik dog et fly til at lande holdet på en klippefyldt “landingsbane”. Her ledte de i 6 uger uden at finde rester efter mennesker.
Det var frygten for forurening, der fik hans hold til at risikere turen til det nordlige Grønland - til et sted, der er så ugæstfrit, at de håbede, at ingen moderne arkæologer have nået det.
To år senere blev det eftersøgte fundet - en hårtot af en forhistorisk mand fra Arktis - på Nationalmuseet i København, hvor Eske Willerslev havde spurgt tidligere, men hvor man måske havde gået fejl af hinanden, idet arkæologerne måske havde tænkt "knogler" og ikke "hår" som DNA-interessant.
Det var direktøren for Statens Naturhistoriske Museum, Morten Meldgård, som fastholdt, at Nationalmuseet måtte have noget, som hans far havde hjembragt fra permafrost ca. 20 år tidligere. Man fandt så håret, som lå opbevaret i en kælder. Håret var fundet på en arkæologisk ekspedition i 1986 i Disko Bugt området på vestkysten af Grønland, som var ledet af Jørgen Meldgaard. Håret havde været opbevaret ved stuetemperatur, men var blevet håndteret meget lidt. En gruppe bestående af bl.a. Eske Willerslev og evolutionsbiologen Thomas Gilbert sekvensbestemte de cirka 16.000 basepar af det mitokondrielle genom, som kunne udvindes af håret, og publicerede i 2008 (ref.9577).
Test dig selv
Du kan tegne abonnement på BioNyt: Videnskabens verden her!
/Homo/DNA/Genom/Inuit/Det korte svar er: Ved omfattende sekvensanalyse (i Kina) af en hårtot på Nationalmuseet i København af en forhistorisk mand fra Arktis
Tre uger efter publiceringen af det mitokondrielle genom af "den første grønlænder" i Science vågnede Eske Willerslev op midt om natten, og tænkte: “Også cellekerne-DNA'et var godt bevaret - og vi kan gøre det på hele genomet,” fortalte han senere til Nature.
Sekvensbestemmelsen af cellekerne- DNA'et fra håret af Saqqaq-manden blev allerede afsluttet i løbet af 2,5 måned, men kostede 500.000 dollar (godt 2½ mill. kr.). Der var tale om et samarbejde med Jun Wang, der er vicedirektør på Beijing Genomics Institute (BGI) i Shenzen, Kina. Eske Willerslev havde kontaktet instituttet efter at have erfaret, at de havde 120 af de nyeste og hurtigste Illumina-sekvensbestemmelsesmaskiner. Samarbejdet er fortsat som et dansk-kinesisk Genomics-center på Beijing Genomics Institute. Det gamle genom blev i første omgang sekvensbestemt ca. 10 gange, som man normalt ville gøre i humangenom- projekter. Men man opdagede, at der kom fejl-sekvenser fra selve sekvensbestemmelsesprocessen. Det offentliggjorte genom er derfor sekvensbestemt 20 gange i gennemsnit over de ca. 79% af genomet, som lod sig lokalisere. Hele genomet blev sekvensbestemt - man kan bare ikke lægge de sidste brikker med blå himmel på plads i puslespillet: DNA fra nulevende mennesker kan kun sekvensbestemmes for 85% af genomet, idet resten er ensarte de base-gentagelser, som ikke kan stedfæstnes.
Der blev fundet 353.151 snip'er (SNP single-nucleotide polymorphisms), hvoraf 6,8% var hidtil ukendte. Det var en bedrift - for det var kun et årti siden det første genom fra et levende menneske blev tydet (ref.9551), og man kæmpede stadig for at undgå forurening af gammelt DNA i prøverne. (Da man brugte klorin-toiletrens til håret fra Saqqaqmanden, var 84% af alt det DNA, der kom ud, menneske-DNA) . Desuden var endnu kun ca. 8 humane genomer blevet sekvensbestemt, da "Inuk" blev sekvensbestemt. (Nu er tusinder af human-genomer sekventeret) . Dette var det første genom fra et forhistorisk menneske overhovedet. For at identificere oprindelsen sammenlignedes med DNA-sekvenser fra nulevende populationer. Man inddrog til dette arbejde Richard Villems, en befolkningsgenetiker ved Tartu Universitet i Estland, der har frysere fyldt op med menneskeprøver, fordi han studerer folkevandringer på verdensplan.
Test dig selv
Du kan tegne abonnement på BioNyt: Videnskabens verden her!
/Homo/DNA/Genom/Inuit/Det korte svar er: Ved at koble signal-sekvenser på DNA-stumpernes ender
Ved studiet af hårtotten af "den første grønlænder" valgte man at beskytte mod forurening ved at Rasmus Nielsen (Købh.univ. og Berkeley Univ.) mærkede hvert fragment af det udvundne DNA med en "stregkode" (dvs. en sekvens af baser, der påsættes DNA-fragmenterne, medens disse stadig er i renrums-laboratoriet, så de senere under sekvensbestemmelsen kan skelnes fra eventuelt omstrejfende DNA fra moderne mennesker, der jo ikke vil have denne stregkode-sekvensstart på fragmenterne).
Test dig selv
Du kan tegne abonnement på BioNyt: Videnskabens verden her!
/Homo/DNA/Genom/Inuit/Det korte svar er: Ca. 85%
Maksimumgrænsen afhænger af længden på de DNA-fragmenter, man får ud. Jo længere de er, jo mere kan man rekonstruere - og hvis de var meget lange, kunne man dække næsten alt (der er stadig dele såsom centromerer og andet, der er tæt på umulige). Det skyldes, at jo længere stykkerne er, jo mere overlap vil der være, og jo nemmere bliver det at løse puslespillet og få placeret alle brikkerne. Hvis stykkerne er for små, kan man ikke klare de repetitive områder. Selv om hele genomet i princippet bliver sekvensbestemt, kan man på grund af de områder, der ligner hinanden, ikke lægge de sidste brikker med blå himmel på plads i puslespillet: DNA fra nulevende mennesker kan kun sekvensbestemmes for 85% af genomet, idet resten er ensarte- de base-gentagelser, som ikke kan stedfæstnes.
Test dig selv
Du kan tegne abonnement på BioNyt: Videnskabens verden her!
/Homo/DNA/Genom/Inuit/Det korte svar er: For 4000 år siden
Den palæo-eskimoiske mand gav man navnet Inuk ("menneske" på grønlandsk). Han levede for 4000 år siden(3400-4500 år) som medlem af Saqqaq-kulturen - den måske første kultur, der bosatte sig i Grønland (ref.9551), men som uddøde i både Alaska, Canada og Grønland uden at efterlade sig efterkommere i dag i den nye verden. De palæo-eskimoiske kulturer, som kaldes Saqqaqkulturen (fra år -2500 til -800) , og den delvis samtidige Independence-I-kultur (fra år -2400? til -1300) , som måske var meget lig med Saqqaq-kulturen (bortset fra at disse mennesker ved at bo i Nordgrønland ikke havde adgang til de samme ressourcer (ref.9653) kendes fra arkæologiske fund i det nordlige Canada og Grønland. Genomet af "Inuk" var forskelligt fra både de moderne indfødte amerikanere og fra de neo-eskimoer, der nu findes i området. Mitokondrie-genomet, som blev publiceret i 2008 i Science, kunne henføres til en haplogruppe D2a1, som kendes fra moderne Aleut-folk og sibiriske Sireniki Yuit, som hverken er direkte beslægtet med indfødte amerikanere (“indianerne”) eller de senere neo-eskimoer (ref.9537). I 2010 lavede Eske Willerslevs afdeling et komplet kernegenom af "Inuk" af Saqqaq-folket. Hvis man sammenligner kerne-DNA'et med nulevende befolkningsgrupper ligner "Inuk" mest folk fra det østlige Sibirien, etniske grupper (Chukchi-, Koryak- og Naganasan- folk), som også har samme A+ blodtype. (Cellekernens genom gav denne oplysning om blodtypen). Det var ikke inuitter, der først kom til Grønland. Det var heller ikke indianere. Derimod var det folk fra Sibirien, som krydsede Beringstrædet og via Alaska og Canada som de måske første nåede Grønland. Vandringen startede for ca. 5500 år siden (ref.9550). Saqqaq-manden for 4000 år siden er i dag tættest beslægtet med folk, som nu 200 generationer senere lever i Sibirien. Genomet viser, at manden var genetisk tilpasset til at leve i kulden. Han havde brune øjne, brun hud, skovlformede fortænder, tørt ørevoks (hvilket øger risikoen for øreinfektioner, men er en fordel for folk, der lever i kolde egne, fordi det samme gen også nedsætter sveddannelsen (ref.9654), tendens til skaldethed (og da der var tale om en ret stor hårtot, var han derfor sikkert ret ung, da han døde).
Test dig selv
Du kan tegne abonnement på BioNyt: Videnskabens verden her!
/Homo/DNA/Genom/Inuit/Det korte svar er: Nej
Det var ikke inuitter, der først kom til Grønland. Det var heller ikke indianere. Derimod var det folk fra Sibirien, som krydsede Beringstrædet og via Alaska og Canada som de første nåede Grønland. Vandringen startede for ca. 5500 år sidenref.9550). Saqqaq-manden for 4000 år siden er i dag tættest beslægtet med folk, som nu 200 generationer senere lever i Sibirien. Genomet viser, at manden var gene-tisk tilpasset til at leve i kulden. Eske Willerslev havde brune øjne, brun hud, skovlformede fortænder, tørt ørevoks (hvilket øger risikoen for øreinfektioner), tendens til skaldethed (og da der jo var tale om en ret stor hårtot, var Eske Willerslev derfor sikkert ret ung, da Eske Willerslev døde). Saqqaq-kulturen eksisterede i ca. 1700 år i Grønland. Den uddøde, måske ca. 800 år før vor tidsregning. Håret og 4 knoglestykker er de ene-ste vævsrester, som man har fundet af disse mennesker fra de 1700 år. Det var ikke Columbus, der opdagede Amerika. Det er sket mange gange, bl.a. for 14500 og for 5500 år siden (ref.9552).
Test dig selv
Du kan tegne abonnement på BioNyt: Videnskabens verden her!
/Homo/DNA/Genom/Inuit/Det korte svar er: En kultur i Grønland der uddøde måske år 800 f.v.t.
De palæoeskimoiske kulturer, som kaldes Saqqaq-kulturen og Independence-I-kulturen, kendes fra arkæologiske fund i det nordlige Canada og Grønland. Deres oprindelse og genetiske relationer til senere kulturer er ukendt. Saqqaq-kulturen eksisterede i ca. 1700 år i Grønland (fra ca. år -2500 til ca. år -800). Den uddøde, måske ca. 800 år før vor tidsregning. Håret og 4 knoglestykker er de eneste menneskevævsrester, som man har fundet fra de 1700 år.
Test dig selv
Du kan tegne abonnement på BioNyt: Videnskabens verden her!
/Homo/DNA/Genom/Inuit/Det korte svar er: Nej
Nej, genomet var forskelligt fra både de moderne indfødte amerikanere og fra de neo-eskimoer, der nu findes i området.
Test dig selv
Du kan tegne abonnement på BioNyt: Videnskabens verden her!
/Homo/Forældre/Mitokondrie/Det korte svar er: Variationen er lille - kun en femtedel af variationen hos chimpansers mitokondrier
Meget tyder på, at de nulevende mennesker er meget ens. Et eksempel herpå er mitokondrierne. Mitokondrier er cellens energifabrik. Mitokondrierne arves kun fra moderen. Mandens sædcelle overfører ikke en eneste mitokondrie til ægcellen. Menneskets mitokondrier indeholder et ringformet DNA-molekyle. Variationen på dette er lille - kun en femtedel af variationen hos chimpansers mitokondrier (ref.9707). Det skyldes nok dels, at mennesket er en ret ny art, dels at der har været flaskehalse, som kun få individer overlevede. Af de mitokondrier, der har været hos mennesketyperne for 200.000 år siden, overlevede kun én type helt til nutiden (ref.9708). Fænomenet kan forklares ved en analogi til efternavne: Hvis en befolkning på 5000 forældrepar, hver med et særligt efternavn, der følger drengene, i hver generation får 2 børn (25% 2 piger, 25% 2 drenge og 50% 1 dreng og 1 pige), så vil 25% af efternavnene altså tabes i hver generation. Efter 100 generationer, f.eks. 2000 år, vil der kun være ét efternavn tilbage. I Kina er nogle efternavne over 4000 år gamle, og folk har i nogle småbyer alle samme efternavn. Om mange tusinde år vil alle i Kina have samme efternavn (ref.9709) med denne navnepolitik.
Test dig selv
Du kan tegne abonnement på BioNyt: Videnskabens verden her!
/Homo/Forældre/Y-kromosom/Det korte svar er: De ældste Y-kromosom-haplogrupper (kaldet A og B) findes kun i Afrika syd for Sahara. Den ældste Y-kromosom- mutation, M91, opstod for i størrelsesordenen 60.000 år siden hos en mand, der levede kort efter stamfaderens oprindelse. Denne mand vandrede mod syd i Østafrika
En mand kan kun arve sit Y-kromosom fra sin far, og det nedarves uforandret. Y-kromosomerne inddeles i talrige haplotyper, som samles i 153 "haplogrupper", og disses slægtskab kan kortlægges ud fra mutationer (betegnet M og et tal). De ældste Y-kromosom-haplogrupper (kaldet A og B) findes kun i Afrika syd for Sahara. Den ældste Y-kromosom- mutation, M91, opstod for i størrelsesordenen 60.000 år siden hos en mand, der levede kort efter stamfaderens oprindelse. Denne mand vandrede mod syd i Østafrika, og etablerede haplogruppe A, der blev dannet ved M91-mutationen (ref.9710). M91-mutationen findes i dag i befolkninger, der taler de såkaldte kliksprog (bl.a. Hadza-folket i Tanzania og !Kung San folket ("buskmænd") i Kalahari (Botswana i det sydlige Afrika), og kliklyde anses derfor for sprogets oprindelse. Alle andre haplogrupper er afledt fra en mutation, som kaldes M168. Alle nulevende ikke-afrikanere bærer uden undtagelse M168 i Y-kromosomet(ref.9711). (Nogle afrikanere har også M168-mutationen i Y-kromosomet).
Test dig selv
Du kan tegne abonnement på BioNyt: Videnskabens verden her!
/Homo/Klima/Det korte svar er: Præget af istiders skiftende klima
Det moderne menneske er udviklet inden for de seneste 1/4 mill. år, hvor der har været to istider, kaldet Saale- istiden og Weichsel-istiden, samt Eem-mellemistiden og den nuværende varmetid (Holocæn), der hidtil har varet 10.000 år. Weichsel-istiden indledtes ret pludseligt for godt 70.000 år siden, og lagde det meste af Europa øde, og i Afrika opstod tørke, der måske reducerede arten Homo sapiens til nogle få tusinde individer (ref.9712).
Test dig selv
Du kan tegne abonnement på BioNyt: Videnskabens verden her!
/Homo/Moderne/Det korte svar er: Ifølge en teori var det en udvikling af den afrikanske population af Homo heidelbergensis
Den afrikanske population af Homo heidelbergensis var blevet isoleret fra den europæiske population af Homo heidelbergensis (idet denne art var opstået for 600.000 år siden i Afrika, hvor istider ikke forekom (ref.9695). Derfor gik den afrikanske populations udvikling i en anden retning: Mod Homo sapiens! (Alternativt var det ifølge en anden teori Homo antecessor, der udviklede sig til Homo sapiens).
Test dig selv
Du kan tegne abonnement på BioNyt: Videnskabens verden her!
/Homo/Moderne/Det korte svar er: Måske for 150.000 år siden
Tidlig Homo sapiens i Afrika udvikledes til moderne Homo sapiens for 150.000 år siden, og ankom til Europa for omkr. 40.000 år siden som CroMagnon-mennesket (ref.9696), der var mere robust end nutidens menneske.
Test dig selv
Du kan tegne abonnement på BioNyt: Videnskabens verden her!
/Homo/Moderne/Det korte svar er: Fladt ansigt og markant hageparti
Homo sapiens har et markant anderledes ansigt: Dels et markant hageparti (der kun ses hos vores art), og dels er ansigtsskelettet fladt og trukket tilbage under hjernekassen, der er opret og ikke vigende.
Test dig selv
Du kan tegne abonnement på BioNyt: Videnskabens verden her!
/Homo/Moderne/Det korte svar er: For ca. 30.000 år siden
Endnu for 30.000 år siden havde verden flere menneskearter.
Test dig selv
Du kan tegne abonnement på BioNyt: Videnskabens verden her!
/Homo/Moderne/Det korte svar er: Kun i 25-30.000 år
Kun i 25-30.000 år har Homo sapiens hersket alene på Jorden uden andre Homo-arters tilstedeværelse.
Test dig selv
Du kan tegne abonnement på BioNyt: Videnskabens verden her!
/Homo/Moderne/Det korte svar er: Fra Afrika
Det har været diskuteret, hvor de moderne mennesketyper kom fra. Ifølge den nu forkastede "multiregionale teori" skulle europæerne være opstået fra de europæiske Neandertalere, asiaterne fra de asiatiske Homo erectus og afrikanerne fra afrikanske Homo heidelbergensis/ergaster/ erectus (ref.9698). Denne teori forsøgte så at forklare opretholdelsen af én art ved, at der skulle være sket en tilstrækkelig opblanding af de forskellige Homo sapiens-typer på tværs af kontinenterne. Til erstatning af denne nu forkastede "multiregionale teori" har man "Ud af Afrika"-teorien, også kaldet "Fortrængningsteorien". Ifølge denne skete udviklingen mod Homo sapiens kun ét sted på Jorden, nemlig i Afrika (ref.9699). En variation af "Ud-af-Afrika"-teorien går på, at der skete opblandinger med lokal menneske-typer, så f.eks. den europæiske type modtog gener fra de europæiske Neandertalere, og tilsvarende i Asien. Forskellen på disse 2-3 teorier er dybden af forskellene mellem "racerne". Faktisk kan begrebet "race" vanskeligt defineres. Ser man f.eks. på hudfarve opdager man, at cellernes hudpigmenter er ens, og at farvevariationen kun skyldes et eller to mutationer - ikke nok til at definere en race.
Test dig selv
Du kan tegne abonnement på BioNyt: Videnskabens verden her!
/Homo/Moderne/Det korte svar er: Bl.a. abstrakt tænkning og et komplekst talesprog
Mennesket er unikt på en række måder. Vi er i stand til at forestille os, hvad andre tænker og føler. Der findes mennesker, som ikke kan dette, men det er et sjældent handicap. Ingen andre dyr end mennesket har evnen til abstrakt tænkning og et komplekst talesprog med symbolske udtryksformer. Der findes mennesker, som ikke forstår omskrivninger og vendinger (såsom "vi tager lige hjem", "give ham fingeren", "tage sig til hovedet" osv.), og som opfatter det bogstaveligt, som børn kan gøre. Men det er meget få, som har dette handicap, og de har bevaret andre evner. Der findes over 5000 forskellige sprog på Jorden (ref.9758). Erhvert barn kan lære ethvert af disse 5000 sprog. Intet menneske behøver at lære, hvordan man bruger et sprog. Alle kulturer har et avanceret sprog. Selv om deres teknologi og organisation er primitiv, er deres sprog ikke primitivt (ref.9759). Næsten alle mennesker mestrer sproget.
Test dig selv
Du kan tegne abonnement på BioNyt: Videnskabens verden her!
/Homo/Moderne/Det korte svar er: Det afhænger af definitionerne på hvilke der er arter.
Der er ikke længere sikkerhed for identiteten af de mennesker, der frembragte Aurignacian-kulturen, selv om den formodede spredning i vestlig retning af de anatomisk moderne mennesker (anatomically modern humans, AMH) på tværs af Europa stadig er baseret på de kontroversielle tidligst daterede fund af Aurignacian-kulturen.
I øjeblikket er den ældste europæiske, anatomisk moderne Homo sapiens repræsenteret ved en robust moderne-menneske underkæbe, som blev opdaget på Pestera cu Oase (sydvestlige Rumænien), dateret til 34,000-36,000 år siden.
Menneskelige skeletrester fra den tyske side af Vogelherd, som indtil videre betragtes som den bedste forbindelse mellem det anatomisk moderne Homo sapiens og Aurignacian-kulturen, viste sig at indeholde stenalder-begravelser i Aurignacian-niveauet og som følge heraf er alle de vigtigste Vogelherd fossiler nu i stedet dateret til at være fra blot 3900 - 5.000 år siden.
Udbredelsen af de første anatomisk moderne mennesker i Europa kan ikke lokaliseres ved hjælp af diagnostiske og godt daterede anatomisk-moderne-menneskers fossiler “vest for Iron Gates i Donau”, før for 32.000 år siden.
Den nøjagtige karakter af biologisk og kulturel interaktion mellem neandertalerne og andre grupper af mennesker for mellem 50.000 og 30.000 år siden, er i øjeblikket meget omstridt.
Et nyt forslag bestræber sig på at løse problemet ved at foreslå, at Gravettians snarere end Aurignacians var de anatomisk moderne mennesker, der har bidraget til den eurasiske genetiske puljen efter for 30.000 år siden.
Tilsvarende blev det menneskelige kraniefragment, der var fundet ved Elbens flodbred ved Hahnöfersand nær Hamburg engang kulstof14-dateret til at være 36.000 år gammel, og blev anset for at være et muligt bevis for krydsning mellem Neanderthalerne og anatomisk moderne mennesker. Det er nu dateret til den nyere mesolitiske periode. .
Test dig selv
Du kan tegne abonnement på BioNyt: Videnskabens verden her!
/Homo/Moderne/Det korte svar er: For ca. 100.000 - 200.000 år siden.
I Afrika opstod Homo sapiens sapiens for ca. 100.000 - 200.000 år siden.
Test dig selv
Du kan tegne abonnement på BioNyt: Videnskabens verden her!
/Homo/Moderne/Blod/Det korte svar er: Som reaktion på sygdomme
Blodtyperne skyldes forskelle på overfladeproteiner på blodlegemerne. Nogle mikroorganismer har kemiske forbindelser på deres overflade, der i den kemiske struktur minder meget om den kemiske struktur af blodtypemolekylerne på overfladen af de røde blodlegemer. Hvis mikroorganismers proteiner har stor lighed med blodlegemernes egne overfladeproteiner, så vil immunsystemet næppe danne sygdomsbeskyttende antistoffer mod mikroorganismen. Koppevirus har stor kemisk lighed med A-blodtypen. Det medfører, at folk med A-blodtypen har 4 gange højere risiko for at dø af kopper (ifølge undersøgelser af den indiske befolkning). I Indien er A-blodtypen netop meget sjældnere end i Europa, hvor det er meget længere siden, at der fandtes kopper (ref.9766). Pestbakterien minder kemisk om 0- blodtypen (nul-typen). De dele i verden, hvor pest har været mest udbredt, er 0-blodtypen mest sjælden (ref.9767). På steder, hvor både kopper og pest forekom, blev B-blodtypen mest udbredt (som i Indien, der har verdens højeste forekomst af B-allelen) (ref.9768). B-allelen kan være opstået efter, at havniveauet steg ved slutningen af sidste istid for 10.000 år siden. Det ville kunne forklare, hvorfor B-allelen ikke findes hos Australiens abo riginere og heller ikke findes hos de oprindelige folk i Amerika, - hvilket altså kan forklares med at begge steder blev afskåret fra yderligere indvandring på grund af havstigningerne (ref.9769). De store epidemier af infektionssygdomme blev formentlig først et stort problem, da mennesker blev fastboende landbrugere. Derfor er den nuværende geografiske fordeling af AB0-blodtyperne formentlig af nyere dato (ref.9770).
Test dig selv
Du kan tegne abonnement på BioNyt: Videnskabens verden her!
/Homo/Moderne/Blod/Det korte svar er: Genetisk anlæg for blod med abnorme hæmoglobinmolekyler
Indførelsen af landbruget medførte også, at malariaparasitten fik alt for fordelagtige vilkår. Nogle mennesker har blod med abnorme hæmoglobinmolekyler, der ændrer struktur, når ilten skal afgives ude i vævene - og strukturændringen i molekylet medfører deformering af de røde blodlegemer, så de får halvmåne-form som et segl. De går også hurtigt til grunde, så der opstår blodmangel. Sådanne personer dør, inden de når den fødedygtige alder. Gen-varianten bæres derfor kun af raske anlægsbærere, der kun har gen-varianten fra en af forældrene. Når gen-varianten alligevel findes på 10-20% af befolkningen i Centralafrika skyldes det, at malariaparasitten ikke kan formere sig i personer med seglcelleanæmi-anlægget, så raske anlægsbærere er beskyttede mod malaria (ref.9771).
Test dig selv
Du kan tegne abonnement på BioNyt: Videnskabens verden her!
/Homo/Moderne/Cro_magnon/Det korte svar er: Det første moderne menneske i Europa
Trods menneskets lange historie i Afrika var Homo sapiens pludselig blevet alene i hele Afrika. Men på grænsen til Europa mødte de overraskende et menneske, som var meget lig dem, men også meget anderledes - neandertaleren (ref.9727), præget af rutine, forudsigelighed, uden evne til at forbedre deres redskaber og med ringe mobilitet og aktionsradius. Neandertaleren levede især af vildoksen ved at ligge på lur og stikke et spyd i den - ikke ved fælder eller kasteskyts (ref.9728). CroMagnon-mennesket var en Homo sapiens, der fangede laks, jagede rensdyr, organiserede sig i store grupper, udvekslede ideer, og hentede materialer langvejs fra til varierede redskaber. Vores art levede sammen med neanderalere for blot 1000 generationer siden (ref.9729). Homo sapiens nåede Sibirien for 35.000 år siden (ref.9730), men på dette tidspunkt var havene delvis blevet til is ved polerne, og under istiden var havniveauet 100-120 m lavere end nu. Beringsstrædet, der i dag er 70 km bredt og 60 meter dybt, var tørlagt. Navnet CroMagnon skyldes det første fund (i 1868) af "de første moderne mennesker i Europa", i en klippehylde kaldet Cro Magnon, ved en lille by Les Eyzies i Dordogne-regionen i det sydvestlige Frankrig, dateret til at være 30.000 år gammelt. Evolutionsmæssigt er 30.000 år ikke meget. Intellektuelt var de sikkert i stand til at flyve F16-fly, men de havde altså kun sten og ben til at lave redskaber af (ref.9740). Benfløjter blev fremstillet for 30.000 år siden (ref.9741). De døde begravedes med gravgods i form af særlige genstande af sten eller ben (ref.9742) ; i en 26.000 år gammel grav fandt man over 10.000 omhyggeligt polerede perler af mammut-elfenben, tilpasset (med mindst en times forbrug pr. perle) i størrelsesfordeling med hinanden, så de sad pænt, når de blev trukket på en snor (ref.9743). Man har fundet over 200 huler med over 1000 hulemaleri-kunstværker, lavet for 18.000-12.000 år siden (ref.9744). Nogle er sammensatte figurer af mennesker og dyr. Nogle kan være syner, oplevet i trance af en shaman påvirket af narkotiske plantestoffer (ref.9745). I de huler, hvor der findes billeder, er der en særlig stærk resonans, så måske har musik, sang eller tilmed dans været brugt i ritualer (ref.9746).
Test dig selv
Du kan tegne abonnement på BioNyt: Videnskabens verden her!
/Homo/Moderne/Hudfarve/Det korte svar er: Ved variation i melano-cellernes stofskifte
Intuitivt opdeler vi befolkninger efter hudfarve og kalder det "racer". Hudfarve, hårfarve og øjenfarve skyldes pigmentet melanin. Det findes i overhud, hårsække (gråhårethed skyldes, at hårsække med alderen ophører med at danne melanin). Pigmentet findes også i øjets regnbuehinde (samt i visse dele af hjernen og i det indre øre). Spredt blandt overhudens celler findes de pigmentdannende celler, som kaldes melanocytter. Alle mennesker har - uanset deres hudfarve - samme antal melanocytter i huden. Men melanocytternes stofskifte kan være forskelligt, hvorved der ændres på sammensætningen, antallet, størrelsen og udseendet af de pigmentkorn, som melanocytterne danner. Pigmentet i mørk hud er fortrinsvis af en type, som kaldes eumelanin (egl.: "sandt melanin"). Pigmentet i lys hud kaldes fæomelanin (gul melanin). Se: "Hvorfor udvikledes hudfarverne hos den moderne Homo sapiens?"
Test dig selv
Du kan tegne abonnement på BioNyt: Videnskabens verden her!
/Homo/Moderne/Hudfarve/Det korte svar er: Selektion for forskellig UV-beskyttelsesbehov
Den mørke eumelanin giver bedre UV-beskyttelse. I mørk hud er eumelanin samlet i mange, store, dybsorte pigmentkorn, spredt enkeltvis rundt i hudcellerne. Den lyse fæomelanin er derimod samlet i mindre grupper, og dette ses især hos europæere og nordlige asiater. Det indebærer en fordel at have lys hud på steder, hvor der er mindre lys, da sollyset ved at trænge ind i huden danner det for bl.a. immunsystemet vigtige D-vitamin. Hudens farve skyldes altså balancen mellem eumelanin og fæomelanin, altså de to former for melanin. Denne balance reguleres af et hormon, som hedder "det melanocytstimulerende hormon, MSH). Det dannes i hypofysen. Dette hormon sendes med blodet ud i kroppen, hvor det kan binde sig til et receptormolekyle på melanocyttens overflade. Når hormonet bindes, stimuleres dannelsen af eumelanin inde i cellen (ref.9700). Alle mennesker danner samme mængde MSH. Men der er hos folk med lys hud (europæere og asiater) stor forskel på opbygningen af receptormolekylet. Receptormolekylet er et protein, og der er forskel på rækkefølgen af proteinets aminosyrer. Derimod er der ingen variation hos negrider fra Centralafrika og andre ækvatoriale egne. Variationen skyldes mutationer i det gen (MC1R-genet), som koder for receptormolekylet. Alle europæere med meget lys hud, fregner og rødt hår har mutationer i genet og de har denne mutation i begge de arvede kopier af genet. Men også generelt for nordeuropæere er mutationerne i genet almindelige (findes hos 75%). Folk med en hud, som let blive brun i solen, har sjældnere mutationer i genet. Asiater har også hyppigt mutationer i genet, men det er andre slags mutationer end europæerne har. Mennesket har et gen (kaldet SLC24A5-genet), som forekommer i to versioner. 98% af europæerne har en muteret variant (kaldet A) af dette gen. Næsten alle andre har det ikke-muterede gen (kaldet G, eller vildtypen). Personer, der har to kopier af mutant-genet, hører til de mest lyshudede, og personer, der har to kopier af vildtype-genet, hører til de mest mørkhudede. Personer med kun en kopi af mutant-genet har en hudfarve imellem de to ekstremer. Det ses hyppigt hos blandingsbefolkninger, såsom afro-caribiske og afroamerikanske. SLC24A5-genet koder for et protein, som sidder i den membran, der omgiver melanosomet. Funktionen af proteinet er at stimulere transporten af calcium ind i cellen, hvilket igen stimulerer dannelsen af melanin. Genet er altså en slags speeder for melanin- dannelsen. I det muterede gen kan speederen ikke aktiveres. Når MSH-hormonet bindes til MC1R-receptoren på melanocyttens overflade stimuleres netop transporten af calcium ind i cellen, hvilket altså stimulerer dannelsen af melanin. Hvor meget calcium, der bliver aktiveret afhænger altså dels af SLC24A5-genet og dels af MC1R-genet. Det meste af variationen i menneskets hudfarve kan således forklares ved variationen i blot to gener !!! (Det kan ikke udelukkes, at flere gener måske er involveret i hudfarve-variationen). Se: "Hvordan udvikledes hudfarverne hos den moderne Homo sapiens?". Se: Hvad skyldes den lyse hudfarve hos europæere?"
Test dig selv
Du kan tegne abonnement på BioNyt: Videnskabens verden her!
/Homo/Moderne/Hudfarve/Det korte svar er: En fejlfunktion i to gener
De hvides lysere hudfarve skyldes en fejlfunktion i to gener (selv om denne fejlfunktion er en fordel i et land med ringe solmængde). Selv om hudfarve er det mest tydelige, er der også andre synlige træk, som populært bruges til at identificere "racer", men de skyldes formentlig alle tilsvarende meget enkle mekanismer, der styres af et meget begrænset antal gener (ref.9701). Det er tankevækkende, at mutationer i blot 2 ud af menneskets ca. 25.000 gener har haft og stadig har så store, tragiske konsekvenser baseret på racefordomme. Årsagen til variationen i hudfarven skyldes tilpasninger til det lokale steds sollys. UV-lys er skadeligt for mennesker ved at ødelægge svedkirtlerne i huden, samt ved at nedbryde vigtige stoffer i huden, foruden at det medfører hudkræft. UV-lys har en enkelt positiv virkning, idet det omdanner et forstadium i huden til aktivt D-vitamin. Befolkningsgrupper, der lever tættere på polerne, hvor sollyset er svagere, er lysere i huden - dels behøves ikke UV-beskyttelsen, og dels øges dannelsen af D-vitamin i den lysere hud. Den nordeuropæiske befolkning er i særlig grad lyshudede, og det passer med, at der her i Nordeuropa dels ikke er meget sollys og dels ofte er meget overskyet (ref.9702). På høje breddegrader vil den bedst tilpassede hud blive hurtig brun i sollys, og hurtig bleg om vinteren, hvor der er mangel på sollys. Eskimoerne er mørkere i huden, - dels medfører isen stor sollysrefleksion, og dels indeholder deres oprindelige kost meget D-vitamin (ref.9703). Melanin hindrer nedbrydning (fotolyse- nedbrydning) af B-vitaminet folinsyre, der er oplagret som depot i huden. Se: "Hvordan udvikledes hudfarverne hos den moderne Homo sapiens?". Se: "Hvorfor udvikledes hudfarverne hos den moderne Homo sapiens?".
Test dig selv
Du kan tegne abonnement på BioNyt: Videnskabens verden her!
/Homo/Moderne/Hudfarve/Det korte svar er: Øget risiko for hudkræft
I Afrika forekommer mangel på pigmenter hos mennesker, der er albinoer. De har tusinde gange forhøjet risiko for hudkræft. I Nigeria dør 9 ud af 10 albinoer inden de er 30 år (ref.9705).
Test dig selv
Du kan tegne abonnement på BioNyt: Videnskabens verden her!
/Homo/Moderne/Sprog/Det korte svar er: Hjerneudvikling eller strubehovedudvikling, eller begge dele
Der er ikke enighed om, hvornår sproget opstod. Medens nogle forskere mener, at det skete for 35.000 år siden - dvs. omkring det tidspunkt, hvor der er de første beviser på en avanceret kultur, mener andre forskere, at det kan være opstået for 2 mill. år siden, hvor der var en markant stigning af hjernens rumfang (ref.9760). Begge dele kan være rigtigt, nemlig en tidlig start, men først en spurt for måske 35.000 år siden for sprogevnen. Strubehovedet er lavt placeret (men højt placeret hos en menneskeabe; det nyfødte barns strubehoved er formet som hos en menneskeabe, men indtager gradvis den voksne placering). Strubehovedets placering kan ses på kraniet - ved vinklingen af kraniets underside (flad hos menneskeaber, stærkt vinklet hos mennesket) (ref.9761). I modsætning til hos de fleste dyr er struben og svælget hos mennesket udformet sådan, at det ikke er muligt at synke maden og trække vejret samtidigt. Disse anatomiske forudsætninger for et avanceret sprog var allerede til stede for 300.000 år siden hos Homo heidelbergensis (ref.9762). Denne art kendte til ild og kunne bygge hytter og redskaber, men om det havde et sprog vides ikke, og der kendes ikke spor efter symbolsk aktivitet (ref.9763). Sprog har afgørende betydning for tankevirksomheden. Det er næsten umuligt at forestille sig tankevirksomhed uden sprog, idet sprog er det medium, hvormed vi forklarer vores tanker for andre. Uden ord forbliver meget udvisket. Man har påvist et "sprog-gen" (der kan være flere) som ved mutationer medfører sprogforstyrrelser. Måske styrer det muskler, der har betydning for sproget eller dele af hjernen. Genet synes at have udviklet sig meget hurtigt siden menneskelinien opstod (ref.9764). Det lidt usikre bud på alderen af de ændringer, som genet har undergået på menneskelinien, er "mindre end 200.000 år" (ref.9765). Det passer med alderen for Homo sapiens.
Test dig selv
Du kan tegne abonnement på BioNyt: Videnskabens verden her!
/Homo/Moderne/Vandring/Afrika/Det korte svar er: Homo ergaster/erectus
Homo ergaster/erectus var den første Homo (det første menneske), som udvandrede fra Afrika, nemlig til Asien og Europa (ref.9676), og dermed den første Homo, som levede uden for den tropiske zone: Homo erectus levede ved det kolde Peking i Kina ("Peking-mennesket") for ½ mill. år siden (ref.9677), og de var de første mennesker, der kom til Europa for 800.000 år siden (ref.9678). Homo erectus var udbredt over meget af Asien, og kan være vandret tilbage til Afrika (ref.9679). Denne art gav formentlig ophav til dværgmennesket Homo floresiensis på øen Flores i Indonesien (ref.9680).
Test dig selv
Du kan tegne abonnement på BioNyt: Videnskabens verden her!
/Homo/Moderne/Vandring/Afrika/Det korte svar er: For måske 2 mill. år siden
Den første udvandring fra Afrika skete måske allerede for 2 mill. år siden, det moderne menneske opstod i Afrika for 150.000 år siden, Cro-Magnon kom til Europa for omkr. 40.000 år siden (ref.9697). Mange arter uddøde undervejs i dette tidsforløb.
Test dig selv
Du kan tegne abonnement på BioNyt: Videnskabens verden her!
/Homo/Moderne/Vandring/Afrika/Det korte svar er: Flere udvandringsbølger, langs kystegne
Der var en tidlig og en sen udvandring fra Afrika af Homo sapiens (ref.9713). Den tidlige for ca. 60.000-70.000 år siden blev ifølge genetiske undersøgelser måske højst foretaget af 5000 mennesker. Formentlig klarede de sig bedst langs floder og i kystegne, hvor de kunne samle muslinger og andet fra havets tidevandszone. Det er netop fra disse kystegne ud for Østafrika, at den første udvandringsbølge for ca. 60-70.000 år siden startede med retning mod Asien, via Yemen (ref.9714), og nåede helt til Australien - hvilket den næste udvandringsbølge faktisk ikke nåede. Denne første bølge nåede ikke Europa, hvilket passer med at Europa ikke lå på vejen, hvis den gik via Yemen. Homo sapiens opstod for 150.000 (eller 150.000 - 200.000 år) siden (ref.9715). Det skete i Afrika.
Test dig selv
Du kan tegne abonnement på BioNyt: Videnskabens verden her!
/Homo/Moderne/Vandring/Afrika/Det korte svar er: Mindst to
Man har påvist to udvandringsbølger ud af Afrika af det modene menneske. Eske Willerslev siger, at personligt forekommer det ham at være sandsynligt, at der har været mange flere ekspansionsbølger. Det må tiden vise, når man får sekvensbestemt mange flere genomer fra alle mulige forskellige folkeslag rundt i verden.
For ca. 70.000 år siden sad folk i Afrika, eller måske i Mellemøsten, som er aboriginernes forfædre, og som tog en beslutning, og sagde: 'Ved du hvad, vi skal ud og kigge på verden. Verden er stor og forunderlig, og vi skal ud at kigge og undersøge den', og så sagde de farvel-og-tobak og begyndte at vandre østover. Europæernes og asiaternes forfædre var på dette tidspunkt "lidt af nogle piveskider", der sad hjemme og sagde, altså, verden er s'gu stor og farlig, uha, vi må hellere blive hjemme. Og så sad vi der og blev hængende. Mens aboriginerne vandrede afsted. " Først mindst 24.000 år efter begynder europæernes og asiaternes forfædre endelig "at tage sig sammen, og siger: 'Vi må også hellere se lidt på verden'. Og så vandrer de også afsted - nogle når op i Europa og bliver til europæere, og der er nogen, der går østover, og bliver til det, som vi kalder asiatere i dag". De møder så efterkommerne fra den første migration. Altså i virkeligheden aboriginernes forfædre. Og de blander sig så med dem - i hvert fald uden for Australien. Og derved kommer asiaterne til at indeholde noget DNA, som fandtes hos aboriginernes forfædre og altså findes den dag i dag hos aboriginerne. Det er det, der gør, at aboriginerne og asiaterne i dag ligner hinanden mere end aboriginerne og europæerne ligner hinanden - indtil den moderne opblanding med europæisk DNA. Det har altså ikke noget at gøre med, at aboriginerne er opstået ved udspaltning fra asiaterne. Det har tværtimod noget at gøre med, at der er sket et sekundært genflow mellem dem. Det er ikke aboriginerne, der ligner asiaterne - men omvendt asiaterne, der via denne indblanding med aborigin-DNA, er kommet til at ligne aboriginerne. Og det giver det resultat, som aboriginerne sikkert godt kunne lide, da Eske Willerslev kom og fremlagde resultaterne. Det betyder, at i virkeligheden kan de godt være efterkommere af de allerførste mennesker, som kom ind i Australien. De isolerede sig nemlig meget tidligere end det, der ligger i den "tilsyneladende DNA-baserede teori om én-gang-migrationsbølgen". "Hvis man skal konkludere på det her, så kan man sige, at frem for den generelle opfattelse, nemlig at der har været én enkelt ekspansion af moderne Homo sapiens mennesker ud i verden, så har der i hvert fald været to", siger Eske Willerslev. Australiens urbefolkning, aboriginerne, er efterkommere efter den første udvandringsbølge, der begyndte i Afrika eller Mellemøsten for 62.000 - 75.000 år siden, og som nåede Australien for omkring 50.000 år siden (mindst 44.000 år siden).
Test dig selv
Du kan tegne abonnement på BioNyt: Videnskabens verden her!
/Homo/Moderne/Vandring/Afrika/Det korte svar er: Aboriginerne i Australien
Aboriginerne i det sydlige Australien var - indtil opblandingen med europæere - formentlig den ældste, kontinuerlige menneskepopulation uden for Afrika (ref.9525). Måske er aboriginerne den menneskegruppe i verden, som har den længste tilknytning til det land, de lever i den dag i dag.
Test dig selv
Du kan tegne abonnement på BioNyt: Videnskabens verden her!
/Homo/Moderne/Vandring/Afrika/Det korte svar er: Ja, relativt meget forskellige - omend alle mennesker også kan siges at være ret ens genetisk set
Mennesker i Afrika er relativt meget forskellige, men det afspejler, at Homo sapiens opstod i Afrika. Dog kan alle mennesker også siges at være ret ens genetisk set.
Test dig selv
Du kan tegne abonnement på BioNyt: Videnskabens verden her!
/Homo/Moderne/Vandring/Afrika/Det korte svar er: Da der har været to udvandringsbølger er der en vis forskel
Studierne af aboriginernes genom viser, at mennesker uden for Afrika er mere forskellige end hidtil antaget.
Test dig selv
Du kan tegne abonnement på BioNyt: Videnskabens verden her!
/Homo/Moderne/Vandring/Afrika/Det korte svar er: Via den arabiske halvø, senere via Levanten
Den første kendte udvandring fra Afrika skete via den sydlige del af den arabiske halvø. Den senere udvandring som nåede Europa skete via Ægypten/ Levanten.
Test dig selv
Du kan tegne abonnement på BioNyt: Videnskabens verden her!
/Homo/Moderne/Vandring/Amerika/Det korte svar er: For 14.300 år siden eller tidligere
Der har været en følelsesladet debat om en lort var fra et menneske, hund eller kamel. Bag dette ligger en hed diskussion i USA om Clovis-folket som det ældste folk i Amerika. Eske Willerslevs gruppe fra Københavns Universitet bestemte lorten fra en hule i USA til at være en menneskelort, og den blev aldersbestemt på to uafhængige laboratorier til at være 14.300 år gammel. Det var næsten 1500 år før det hidtil tidligste sikre bevis for menneskers eksistens i Amerika. Det var dermed før Clovis-folket, og man havde ikke tidligere kendt til nogen sikker før-Clovis kultur. [Der er to måder at angive alder på - en ikke-kalibreret carbon-14 og en kalibreret carbon-14 metode. Hvis et tal, der er kalibreret, sammenlignes med et andet tal, der er ukalibreret, kommer der mere forskel, end der skal være]. Vurderet ud fra sprogene hos "oprindelige amerikanere" er der sket talrige indvandringer til Amerika, hvor de seneste var Na-Dene-indianere for 8-10.000 år siden og inuitter (eskimo-aleuterne) for 5000-5500 år siden (ref.9732). 9733), dog ifølge arkæologiske fund og en særlig M242-mutation (opstået i Asien) er indvandringen næppe sket tidligere end for 18.000 år siden (ref.9734). Den første koloniseringsbølge til Amerika omfattede mindre end 100 individer, ifølge genetiske undersøgelser (ref.9735). 97% af alle nulevende indianere nedstammer fra 4 haplotyper af mitokondrier (A-D), der ikke findes hos nulevende afrikanere eller europæere - fordi disse mitokondrie- mutationer er opstået i Asien. Mod slutningen af sidste istid indvandrede mammutjægere (Clovis-folket) til Amerika. De lavede en spydspids, som var rendeformet udhulet på den side, som skulle vende ind mod spydtræet. Måske ankom kun 100 individer via Bering-broen for 13.500 år siden. Med en generationstid på 25 år, og med 2-3% årlig befolkningsvækst (idet der var masser af jagtdyr, som mammutter, mastodonter, bison, kæmpedovendyr, vildheste, tre kamelarter, kæmpegnavere, tapirer og flere antilope-arter) kunne Clovis-folket på bare 300 år vokse til 1 million, hvilket de arkæologiske fund også tyder på (ref.9736), - selv om der også var farlige dyr, som løver, bjørne, sabelkatte og ulve. Efter ca. 500 succesfulde år uddøde Clovis-folket pludselig for 12.800 år siden, men da var der i Nordamerika også kun 20% af den mængde storvildt, som der havde været, da de ankom (ref.9737). Mange arter var uddøde, bl.a. mammutten, mastodonten, kæmpedovendyret, vildhesten, flere bjørnearter og sabelkattene. Clovis-folket afløstes af Folsom-folket, som brugte en anden jagtteknik: De jagtede bl.a. flokke af bison, det eneste tilbageværende storvildt. Visse steder kunne de med ild skræmme en flok på op til 50 dyr til at løbe ud over en klippeskrænt. Måske stammede de fra rester af Clovis-folket. Over de næste 1000 år befolkede Folsom-folket hele Amerika, ned til sydspidsen af Sydamerika, en distance på over 8000 km (ref.9738). Da Columbus ankom, var der mange millioner mennesker i Amerika. Nogle Mayaog Aztec-byer havde over 100.000 indbyggere. Der var opstået over 1000 sprog og kulturer i Amerika i løbet af de 1000 år (ref.9739).
Eske Willerslevs gruppe fra Københavns Universitet har i Science okt. 2011 vist, at en mastodont var blevet dræbt af et spyd ca. 1000 år tidligere end det, som man kalder Clovis- kulturen. Mastodonten var en kæmpeelefant, Mammut americanum, ca. 3 meter høj, med mere lige stødtænder end mammutten, ikke hældende ryg som en mammut, længere hoved, på størrelse med en Indisk Elefant, små ører, tykt hår, den åd buske og småtræer, ikke især urter som mammut.
Morten Rasmussen er ved at sekvensbestemme et knoglefund fra Clovis-kulturen for at finde ud af, hvem der er de nærmeste slægtninge i Asien og Nordamerika. Han er “postdoc” efter afsluttet ph.d.-projekt om at få de nye teknikker til at virke på gammelt DNA.
Test dig selv
Du kan tegne abonnement på BioNyt: Videnskabens verden her!
/Homo/Moderne/Vandring/Amerika/Det korte svar er: Langs kysten via Beringsstrædet formentlig
Under Istiden var der isdækket hele vejen til Nordamerika. For ca. 12000 år siden åbnede der sig en isfri passage mellem to iskapper, og man har gættet på, at mennesker kom denne vej. Men en alternativ teori om, at de første mennesker kom ad kystvejen via Beringsstrædet, passer bedre med, at Eske Willerslevs gruppe på Københavns Universitet har fundet ud af, at der var mennesker i Nordamerika ca. 1000-1500 år tidligere end Clovis. Dels kunne de i Science okt. 2011 rapportere om en mastodont (kæmpeelefant), som var blevet dræbt af et spyd - og som var dateret til ca. tusinde år tidligere end det, som man kalder Clovis-kulturen, som ellers blev betragtet som det ældste bevis på menneskelig tilstedeværelse i Amerika. Dels gjorde man følgende opdagelse:
I USA har det blandt arkæologer været ivrigt debatteret, hvilken befolkningsgruppe, der først koloniserede Amerika, og hvornår. Diskussionen kan nu være blevet afgjort af et fund i Oregon-ørkenen, hvor man i hulerne "Paisley Caves", fandt fossile ekskrementer, samt knogler og stenredskaber. I 2002 fandt Paisley Jenkins fra Oregon State Museum of Natural and Cultural History og hans 26 studerende rester af forhistorisk liv på stedet: Knogler af heste, kameler, bjergfår mv., nogle med snitmærker, samt stenredskaber og et dusin ovale, organiske emner, som var udtørrede ekskrementer (koprolitter). Eske Willerslev, som i et amerikansk, arkæologisk tidsskrift beskrives som “an enthusiastic Dane known among his colleagues for his friendly personality, foul mouth and outstanding research”, fløj til Oregon i 2004 for at tage prøver. Nogle af knoglerne havde stadig blødt væv i sig. Ekskrementerne så ud som menneskelorte. De lå så i en fryser i København, medens Eske Willerslev ledte efter DNA fra bunden af Grøn-lands indlandsis, og udtrak mammut-DNA af den sibiriske permafrost.
I 2006 fik Jenkins pludselig en email fra Eske Willerslev, der spurgte, hvor gammel koprolitten var. I København var man nemlig nu kommet til menneskelorten, og havde fundet menneske-DNA, som havde genetiske træk, som kun kendes hos ameri-kanske indianere. Da alderen ikke var fastlagt, blev laboratorier i Oxford og Florida sat til at lave en datering ud for kulstof-14, betalt for penge, som Eske Willerslev skaffede. Begge laboratorier nåede frem til, at menneskelortene var 14.300 år gamle. Det var næsten 1500 år før det hidtil tidligste sikre bevis for menneskers eksistens i Amerika. Det var altså før-Clovis, og man havde ikke tidligere kendt til nogen før-Clovis kultur. [Der er to måder at angive alder på - en ikke-kalibreret carbon-14 og en kalibreret carbon-14. Hvis man får de to tal blandet, kommer der mere forskel, end der skal være]. I hundrede år havde arkæologer skrevet i deres bøger, at det første menneske i Amerika levede for ca. 13.000 år siden - baseret på fund af stenredskaber nær Clovis, New Mexico, i 1930’erne. Efterhånden var Clovis-kom-først teorien blevet den almindelige antagelse. Menneskelorte er et bedre bevis end stenredskaber, som jo kan være med-bragt fra andre steder. [Retsgenetikere anser dog ekskrementer for et usikkert materiale at lave DNA-studier på]. (Der var seks lorte - men tre var ikke menneske-lorte. To af dem var spændende, fordi de ifølge kulstof-14 datering var før Clovis-kulturen(ref.9576). Danske DNA-studier omskrev altså den amerikanske forhistorie(ref.9547). Der vil dog altid være nogle modstandere, der siger, at det bare er forurening. Det er Eske Willerslev nu ret sikker på, at det ikke var. Bl.a. fordi man tog DNA-prøver fra alle, der havde været med til udgravningen.
Man har antaget, at Amerika var uopdaget af mennesker indtil for 13.000 år siden - og at det første menneske i Amerika var en mammut-jagende asiat. Man har fundet menneskeknogler, der er dateret til at være 12000 år gamle, tilhørende Clovis-kulturen, som er defineret ved en speciel pilespids og stenværktøj, hvor flagerne er slået af på en bestemt måde. Der er debat i Amerika om, hvor Clovis-folket kom fra. Var de oprindeligt europæere, der senere blev fortrængt af de amerikanske indianeres forfædre? Eller var de forfædre til indianerne?
Test dig selv
Du kan tegne abonnement på BioNyt: Videnskabens verden her!
/Homo/Moderne/Vandring/Europa/Det korte svar er: Over en lang periode fra for 100.000 år siden til blot for 700 år siden.
Vores art nåede Sydafrika for 100.000 år siden, Australien for 50.000 år siden, Nordeuropa for 40.000 år siden, Alaska for måske 18.000 år siden, Sydamerika for måske 14.000 år, Japan fra nord for 12.000 år siden og fra syd for 2300 år siden (ref.9716), Madagaskar (fra Asien!!) for 3200 år siden. Koloniseringen af Stillehavets øer er ikke mindre imponerende. Det måtte ske i små både. Fiji blev nået for 3500 år siden, Tahiti for 2200 år siden, Marquesa (mellem Tahiti og Hawaii) for 1700 år siden, Hawaii for 1500 år siden og Rapanui (også kaldet Påskeøen, midt mellem Tahiti og Sydamerika) for måske 800 år siden (ref.9717) eller måske tidligere. New Zealand blev også nået for ca. 750-800 år siden, idet polynesere bosatte sig i New Zealand omkring år 1250–1300 [europæere ankom først i år 1642]. Udriggerkanoen, der blev brugt til at kolonisere Stillehavet, blev udviklet i Taiwan, hvor også den austronesiske sproggruppe opstod, og som i dag omfatter 1200 forskellige sprog, der tales af 200 millioner oprindelige folk i Thailand, central-Sydvietnam, Malaysia, Filippinerne, Indonesien, Taiwan og mange Stillehavsøer - og Madagaskar (denne ø ud for Afrika er oprindelig ikke befolket af folk fra Afrika-kontinentet!) (ref.9718). Taiwan blev koloniseret af negritofolk, rekoloniseret af risdyrkere (for 6000 år siden) fra dalen ved Yangtzefloden og igen rekoloniseret af Hantalende kinesere (i 1600-tallet) (ref.9719).
Test dig selv
Du kan tegne abonnement på BioNyt: Videnskabens verden her!
/Homo/Moderne/Vandring/Europa/Det korte svar er: For 45000 år siden
Indvandringen til Europa skete for ca. 45000 år siden, i den såkaldte Hengelo-varmeperiode midt i Weichel-istiden (ref.9720), idet mennesket fulgte i hælene på vildtet nordpå, via Levanten og derfra via Centralasien ind i Europa. CroMagnon-menneskets genetiske kendetegn er mitokondriemutationen M173, der er afledt af M45-mutationen, der opstod i Centralasien omkring dette tidspunkt (ref.9721). En mindre indvandring er sket via Balkan for mindst 40.000 år siden ifølge de ældste fossiler i Europa af Homo sapiens (ref.9722). I Europa etablerede Homo sapiens den kultur, som kaldes Aurignacien (40.000-28.000 år før nu (ref.9723), og som markerer menneskets "store spring fremad" (ref.9723), formentlig styret af en markant udvikling af hjernen og dermed sprog og teknologi - samt den midlertidige klimaforbedring og dermed konkurrencefordel overfor neandertalerne. Befolkningseksplosionen hos Homo sapiens medførte neandertalernes uddøen, da de blev presset af den næste kuldeperiode, der var den værste kuldeperiode i 130.000 år (ref.9724). Neandertalerne uddøde kort før kulde-kulminationen, der indtraf for 21.000 til 18.000 år siden. Derefter ændredes klimaet dramatisk. Den varmeste periode siden sidste istid var mellem 8000 og 5000 år før nu (Atlantisk tid), og i denne periode spredtes landbruget op gennem Europa (ref.9725).
Test dig selv
Du kan tegne abonnement på BioNyt: Videnskabens verden her!
/Homo/Moderne/Vandring/Europa/Det korte svar er: På grund af teknologiske forskelle og adgang til ressourcer
Europæerne koloniserede Amerika og Australien. Dette skyldtes ikke biologiske forskelle, men teknologiske og politiske forskelle, bl.a. som følge af adgang til ressourcer. Europa brugte jernredskaber, havde skriftsprog, landbrug, centralstyrede stater og skibsfart. Befolkningerne i Amerika og Australien manglede centralstyrede stater, jern, skibe, skriftsprog (ref.9772). I Europa og Asien kunne befolkningstallet stige voldsomt, og tætte befolkninger er en forudsætning for statsdannelser. Dette øgede infektionssygdomme, som befolkningerne så med tiden blev modstandsdygtige overfor (de ikke modstandsdygtige nåede ikke at få børn). Ved koloniseringerne var medbragte sygdomme en del af årsagen til, at invasionen gik let. Våben, skibe og skriftsprog var også medvirkende hertil. Læs kilder og mere om bladets
Test dig selv
Du kan tegne abonnement på BioNyt: Videnskabens verden her!
/Homo/Moderne/Ørevoks/Det korte svar er: To tyder, den ene genetisk dominerende, den anden vigende (recessiv)
Der er to typer ørevoks (cerumen) : Den våde type, som er genetisk dominerende, og den tørre type, som er genetisk recessiv (vigende). Sorte og hvide mennesker er mere tilbøjelige til at have den våde type (honningbrun-mørkebrun og fugtig). Ørevokstypen har været anvendt af antropologer til at spore befolkningers vandringsveje. Den våde type er f.eks. mere udbredt blandt Ainu i Nordjapan, i modsætning til sydjapanere. Den våde ørevoks skyldes højere koncentration af lipid og pigmentkorn (50% lipid), hvorimod den tørre type kun indeholder 30% lipid. Forskellen skyldes en enkelt DNA-baseændring i et gen, som er kendt som “ATP-bindende kassette C11 gen.” Ud over at påvirke ørevokstypen, reducerer denne mutation også svedproduktionen - hvilket man antager har været til gavn for forfædrene til østasiaterne og de indfødte amerikanere, som menes at have levet i et koldt klima. Asiater, almindelige japanere og indfødte amerikanere er mere tilbøjelige til at have den tørre type (grå og skællet), dvs. den genetisk recessive type. Saqqaq-manden “Inuk”, der levede i Vestgrønland for 4000 år siden, havde også den tørre ørevokstype (ref.9654).
Test dig selv
Du kan tegne abonnement på BioNyt: Videnskabens verden her!
/Homo/Neanderthal/Det korte svar er: Ikke endelig bevist, men en mulighed ifølge nogle data
Er der Neandertal-DNA i det moderne menneske (Homo sapiens)? Mange steder kan man læse - som var det et fastslået faktum - at der er ca. 2% Neandertal-DNA i alle moderne mennesker uden for Afrika, eller 1%-4%. "Det kan man ikke sige", mener Eske Willerslev. "Vi ved det faktisk ikke - og det har noget at gøre med, at det, som man kalder nandertal-signalet (altså DNAligheder mellem neandertaler-DNA og vores DNA) kan være kommet ved et såkaldt genflow. I den nævnte 2%-teori går man ud fra, at det moderne menneske, der vandrer ud af Afrika, møder Neandertalere og har sex med dem, inden de spreder sig videre ud i verden. Problemet er bare, at man faktisk også kan forklare DNA-lighederne mellem Neandertalere og os på en anden måde: "At Neandertaleren og det moderne menneske i Afrika har et fælles udspring," siger Eske Willerslev. "Med de data, der i dag er til rådighed, kan man ikke skelne mellem de to teorier. Neandertal-2%-sex-teorien går verden rundt som et forsknings- resultat, fordi det er en meget bedre historie. Det er så simpelt - videnskab er ikke mere hellig end som så. Det vil sige, at det er den teori, der bliver favoriseret. Men i virkeligheden kan man ikke sige det, baseret på de data, der er til rådighed," siger han. Ligesom andre befolkninger uden for Afrika, har den australske aboriginalmand måske visse små stumper af sit genom fra neandertalerne, - eller disse små genom-stykker stammer fra før Neandertal-udskillelsen.
Test dig selv
Du kan tegne abonnement på BioNyt: Videnskabens verden her!
/Homo/Neanderthal/Det korte svar er: Udviklet fra Homo heidelbergensis eller Homo rhodesiensis
Den første proto-Neandertaler dukkede op i Europa så tidligt som for 600,000-350,000 år siden.
Proto-neanderthalerne angives undertiden som arter: Homo heidelbergensis eller Homo rhodesiensis.
Det er blevet foreslået ud fra genetiske data, at der fandt krydsning sted med anatomisk moderne mennesker på et tidspunkt mellem ca. 80.000 og 50.000 år siden i Mellemøsten, hvilket resulterede i, at 1-4% af genomet i folk fra Eurasien skyldes DNA-bidrag fra neandertalerne.Men dette “neanderthal-DNA” kan også være DNA, som blot fandtes i den fælles stamform mellem neanderthaleren og det moderne menneske. Med den nuværende viden kan man ikke afgøre, hvilken af disse to teorier, der er korrekt.
Det yngste Neanderthal-fund er fra Hyaena Den (England), og anses for ældre end 30.000 år siden, mens Vindija (Kroatien) neandertalere er blevet re-dateret til stadig at leve på et tidspunkt for 33.000 - 32.000 år siden. Ingen fund er yngre end 30.000 år. men tegn på anvendelse af ild kan betyde, at der har levet neandertalere på Gibraltar indtil for kun 24000 år siden.
Cro-Magnon mennesket, dvs. tidlige moderne mennesker med ‘Neanderthal træk’ blev fundet i Lagar Velho (Portugal), dateret til for 24.500 år siden. Det er blevet tolket som tegn på en blanding mellem disse mennesketyper.
Flere kulturelle fund har været knyttet til neandertalere i Europa. De tidligste, Mousterian stenværktøjkultur, er blevet dateret til for ca. 300.000 år siden. Senere Mousterian kultur ses også i Asien og i Afrika dateret til at være efter 150.000 år siden på et sted, kaldet Jebel Irhoud, der ligger 620 km syd for Gibraltar.
Sene Mousterian-fund blev fundet i Gorham Hulen på den afsides beliggende, sydvendte kyst ved Gibraltar.
Andre værktøjskulturer, der er blevet sat i forbindelse med Neanderthal-mennesket, omfatter Châtelperronian, Aurignacian, og Gravettian, der synes at være udviklet med gradvis kontinuitet.
Test dig selv
Du kan tegne abonnement på BioNyt: Videnskabens verden her!
/Homo/Neanderthal/Det korte svar er: Som hos Homo sapiens
Neanderthalerens kraniekapacitet menes at have været lige så stor som en Homo sapiens, måske større, hvilket tyder på at deres hjernestørrelse kan have været af sammenlignelig størrelse, eller større.
I 2008 udførte en gruppe forskere et studie med tre-dimensionel computer-assisteret rekonstruktion af Neanderthal-spædbørn baseret på fossiler, der var fundet i Rusland og Syrien.
Undersøgelsen viste, at Neanderthalerens hjerne og det moderne menneskers hjerner var af samme størrelse ved fødslen, men ved voksenalderen blev Neandertalhjernen større end det moderne menneskes hjerne.
Test dig selv
Du kan tegne abonnement på BioNyt: Videnskabens verden her!
/Homo/Neanderthal/Det korte svar er: Meget stærkere end Homo sapiens
Neanderthaleren var meget stærkere end Homo sapiens, især havde de særligt stærke arme og hænder. Neanderthaler-manden var 168-164 cm høj og kvinden var ca. 152-156 cm høj.
Test dig selv
Du kan tegne abonnement på BioNyt: Videnskabens verden her!
/Homo/Neanderthal/Det korte svar er: Kød- og plantespisende, kendskab til varmetilberedning af kosten
I 2010 publicerede en amerikansk forsker en rapport om fund af kogt plantemateriale i tænderne på et neanderthal-kranium, hvilket modsiger den tidligere antagelse, at de udelukkende (eller næsten udelukkende) var kødspisende og i toppen af predatorkæden.
Test dig selv
Du kan tegne abonnement på BioNyt: Videnskabens verden her!
/Homo/Neanderthal/Det korte svar er: Neanderthaleren er opkaldt efter Neander-dalen, der ligger ca. 12 km øst for Düsseldorf, Tyskland
Neanderthaleren er opkaldt efter Neander-dalen, der ligger ca. 12 km øst for Düsseldorf, Tyskland. Denne kløft er blevet dannet af floden Düssel, og kløften er blevet udvidet på grund af minedrift.
Kløften blev kaldt Neanderthal i begyndelsen af det 19. århundrede for at ære en præst og salmedigter, Joachim Neander: “Tal” er det tyske ord for “dal”. . (det blev stavet “Thal” indtil den tyske stavereform i 1901), og stavningen af dalen blev også ændret i overensstemmelse hermed til Neandertal. Den tidligere stavning er imidlertid ofte bevaret på engelsk for hominider. Stavemåden med th er derudover altid brugt i videnskabelige navne i hele verden. På tysk er den moderne stavemåde med T i stedet for Th dog i brug, når der henvises til både hominiden og dalen.
Fossilet blev opdaget i Feldhofer-hulen i kalkstensklipper i Neanderthal i 1856,
Test dig selv
Du kan tegne abonnement på BioNyt: Videnskabens verden her!
/Homo/Neanderthal/Det korte svar er: Inden for de sidste 200.000 år udviklede Homo heidelbergensis sig til Homo neanderthalensis ifølge en teori
Neanderthalerne udviklede sig fra tidlige Homo-typer ad en vej, der ligner den vej, som Homo sapiens udviklede sig. Begge stammer fra en chimpanse-lignende forfader, der levede for mellem 5 og 10 millioner år siden.
Ligesom Homo sapiens er neandertalerne beslægtet med Australopithecus, Homo habilis og Homo ergaster; men den præcise afstamning er fortsat usikker. Den sidste fælles forfader mellem det anatomisk moderne Homo sapiens og neandertaleren synes at være Homo rhodesiensis, opkaldt efter en gammel Homo-fossil, Broken Hill 1 (Kabwe 1), der blev opdaget i et område af Rhodesia i 1921.;.
Homo rhodesiensis opstod i Afrika for - skønnet - cirka 700.000 til 1.000.000 år siden. Det tidligste skøn for, hvornår Homo rhodesiensis nåede Europa, er for ca. 800.000 år siden, da en form for menneske, benævnt Homo antecessor eller Homo cepranensis, allerede beboede i regionen. Disse to mennesketyper kan være forløbere til den europæiske Homo heidelbergensis, men stenredskaber, der stammer fra for 1,2 til 1,56 millioner år siden, af en ukendt oprindelse er blevet opdaget i det sydvestlige Europa. Fundet Sima de los Huesos (i Atapuerca-hulesystemet på Den Iberiske Halvø) antyder, at Homo heidelbergensis allerede var i Europa for 600.000 år siden.
Molekylære fylogenetiske analyser tyder på, at Homo rhodesiensis og Homo heidelbergensis fortsatte med at blande sig indtil for 350.000 år siden, hvorefter de blev til adskilte arter, og engang inden for de sidste 200.000 år udviklede Homo heidelbergensis sig til Homo neanderthalensis, det klassiske Neanderthal-menneske.
Det ser ud til, at den oprindelige Neanderthal-befolkning i virkeligheden var mere fjernt beslægtet med nutidens menneske, end Homo heidelbergensis var. Dette forudsætter så, at Neanderthalmennesket faktisk ikke krydsede med det moderne menneske, som nogle påstår, men som dataene ikke er gode nok til at bekræfte ifølge Eske Willerslev.
Test dig selv
Du kan tegne abonnement på BioNyt: Videnskabens verden her!
/Homo/Neanderthal/Det korte svar er: Man har fundet knogler fra over 400 neandertalere
Neanderthal-kranier blev først opdaget i Engis-hulerne, i hvad der nu er Belgien (1829), af Philippe-Charles Schmerling og i Forbes ‘Quarry, Gibraltar (1848), begge forud for det “typefund”, der blev gjort i et kalkbrud i Neander-dalen i Düsseldorf i august 1856, tre år før Charles Darwins udgav Arternes Oprindelse.
Typefundet, som blev kaldt Neanderthal 1, bestod af en hovedskal, to lårbensknogler, tre knogler fra højre arm, to fra venstre arm, en del af venstre hofteben (ilium), fragmenter af et skulderblad, og ribben.
De arbejdere, der fandt dette materiale, troede oprindeligt, at det kunne være resterne af en bjørn. De gav materialet til amatør-naturforskeren Johann Carl Fuhlrott, der viderebragte fossilerne til anatomen Hermann Schaaffhausen. Opdagelsen blev publiceret i 1857.;.
Det oprindelige Neanderthal-fund betragtes som begyndelsen på palæoantropologien. Disse og andre fund førte til ideen om, at disse jordiske rester var fra gamle europæere, der havde spillet en vigtig rolle i det moderne menneskes oprindelse. Siden har man fundet knogler fra over 400 neandertalere.
Tidslinje
1829: Neanderthal kranier blev opdaget i Engis, i nutidens Belgien.
1848: Neandertalkranium fundet i Forbes ‘Quarry, Gibraltar. Kaldes “et ældgammelt menneske” på det tidspunkt.
1856: Johann Karl Fuhlrott anerkendte fossilerne kaldet “neanderthal-manden”, som blev opdaget i Neanderthal, en dal nær Mettmann i, hvad der nu er Nordrhein-Westfalen, Tyskland.
1880: underkæben af et Neanderthal-barn blev fundet i forbindelse med kulturelle rester, herunder ildsteder, Mousterian-redskaber og knogler af uddøde dyr.
1886: To næsten perfekte skeletter af en mand og en kvinde blev fundet ved Spy i Belgien i en dybde af 16 meter med mange redskaber af Mousterian-type.
1899: Hundredvis af Neanderthal-knogler blev beskrevet i stratigrafisk position i forbindelse med kulturelle rester og uddøde dyreknogler.
1908: Et næsten komplet Neanderthal skelet blev opdaget i forbindelse med Mousterian-redskaber og knogler af uddøde dyr.
1925: Francis Turville-Petre finder ‘Galilæa manden’ eller ‘Galilæa hovedbunden’ i Zuttiyeh hulen i Wadi Amud i Palæstina (nu Israel).
1953-1957: Ralph Solecki finder ni Neandertaler-skeletter i Shanidar hulen i det nordlige Irak.
1975: Erik Trinkaus undersøgelse af Neanderthal-menneskets fødder bekræftede, at de gik som moderne mennesker.
1987: Thermoluminescence-resultater fra israelske fossiler daterer neandertalerne på Kebara til at leve for 60.000 år siden og mennesker ved Qafzeh til at leve ved 90.000 år siden. Disse datoer blev bekræftet af elektronspin-resonans (ESR) datering for Qafzeh (90.000 år) og Es Skhul (80.000 år).
1991: Elektronspin-resonanselektronspin-resonans (ESR) datoer viste at Tabun-Neanderthaleren var samtidig med de moderne mennesker fra Skhul og Qafzeh.
1993: 127.000 år gammelt DNA blev fundet på et barn fra Sclayn, der findes i Scladina (fr), Belgien.
1997: Matthias Krings et al. er de første, som det lykkes at opformere Neanderthal-mitokondrie-DNA (mtDNA) ved hjælp af materiale fra Feldhofer grotten i Neander-dalen.
2000: Igor Ovchinnikov, Kirsten Liden, William Goodman et al. henter DNA fra et sen-neanderthal-spædbarn (29.000 år siden) fra Mezmaiskaya hulen i Kaukasus. .
2005: Max Planck Instituttet for Evolutionær Antropologi lancerer et projekt til at rekonstruere Neanderthal-genomet.
2006: Max Planck Instituttet for Evolutionær Antropologi annoncerer at det vil arbejde med Connecticut-baserede 454 Life Sciences men henblik på at rekonstruere Neanderthal-genomet.
2009:. Max Planck Instituttet for Evolutionær Antropologi annoncerer at de nu har det “første udkast” af et komplet Neanderthal-genom.
2010: Sammenligning af Neanderthal-genomet med det moderne menneske fra Afrika og Eurasien tyder på, at 1-4% af moderne ikke-afrikanske menneskelige genom måske kunne komme fra neandertalere (eller være .fra fælles stamformer)
Test dig selv
Du kan tegne abonnement på BioNyt: Videnskabens verden her!
/Homo/Neanderthal/Det korte svar er: I den sidste istid, over et tidsrum på ca. 100.000 år over det meste af Europa syd for israndslinien, omtrent langs den 50. nordlige breddegrad, herunder det meste af Vesteuropa, den sydlige kyst af Storbritannien, Centraleuropa og Balkan-landene, nogle steder i Ukraine og i det vestlige Rusland, frem til Sibirien til Altai-bjergene og syd gennem Levanten til Indus-floden.
Tidlige Neanderthalere levede i den sidste istid, over et tidsrum på ca. 100.000 år. På grund af de ødelæggende virkninger, som istiden havde på de steder, hvor Neanderthalerne engang boede, kender man ikke meget til de tidlige typer af neanderthalere.
Lande, hvor der er gjort neanderthal-und, omfatter det meste af Europa syd for israndslinien, omtrent langs den 50. nordlige breddegrad, herunder det meste af Vesteuropa, den sydlige kyst af Storbritannien, Centraleuropa og Balkan-landene, nogle steder i Ukraine og i det vestlige Rusland, frem til Sibirien til Altai-bjergene og syd gennem Levanten til Indus-floden. Det anslås, at den samlede Neanderthal-befolkning over dette store område var på op til 70.000 på sit højeste.
Neanderthal-fossiler er ikke indtil nu blevet fundet i Afrika, men der har været fund temmelig tæt på Afrika, både i Gibraltar og i Levanten. På nogle Levantinske steder, daterer Neandertal-resterne sig efter et tidspunkt, hvor Homo sapiens var flyttet fra de samme steder.
Pattedyrfossiler fra den samme periode viser, at dyr, der var tilpasset et koldt klima, levede samme steder som neanderthalere i det østlige Middelhav. Dette indebærer, at Neanderthalerne var bedre biologisk-tilpasset til koldt vejr end Homo sapiens og at neanderthalerne til tider fordrev Homo sapiens i dele af Mellemøsten, når klimaet blev koldt nok. Homo sapiens synes at have været den eneste menneskelig type i dalen ca. Nilen i disse perioder, og Neanderthalerne er ikke kendt for nogensinde at have levet sydvest for det moderne Israel.
Når yderligere klimaændringer forårsagede varmere temperaturer, fortrak Neanderthalerne formentlig til nordligere områder sammen med de pattedyr, der var tilpasset kulde. Tilsyneladende skete disse klimafremkaldte migrationer før det “moderne” menneske sikrede sig konkurrencefordele overfor Neanderthalerne, da disse skift i udbredelse fandt sted over godt titusind år før det moderne menneske helt erstattede Neanderthalerne.
Der var forskellige udviklinger i menneskelinje i andre regioner, såsom i det sydlige Afrika, som i nogen grad ligner neanderthalerne i Europa og det vestlige / centralasiatiske område, men disse mennesker var ikke rigtige neandertalere. Et eksempel herpå er Rhodesian-mennesket (Homo rhodesiensis), der eksisterede længe før, der fandtes nogen klassiske, europæiske neandertalere, men de havde et mere moderne sæt tænder, og velsagtens var nogle Homo rhodesiensis populationer var på vej til at udvikle sig i retning af det moderne Homo sapiens sapiens.
Endnu har man ikke fundet nogen intim sammenhæng mellem disse menneskelige typer og de vestlige og central-eurasiske neandertalere, i det mindste i den tidsperiode, hvor de klassisk eurasiske neandertalere eksisterede, og Homo rhodesiensis synes at have udviklet sig for sig selv og tidligere end de klassiske neandertalere i en konvergent evolution.;.
Det ser ud til at være forkert, baseret på nuværende forskning og kendte fossilfund, at henføre noget som helst fossilfund uden for Europa eller Vest- og Centralasien, til at være Neanderthalere. De egentlige Neanderthalere havde en udbredelse, som muligvis strakte sig så langt mod øst som til Altai-bjergene, men ikke længere mod øst ellermod syd, og tilsyneladende ikke ind i Afrika. I hvert fald var landet umiddelbart syd for Neanderthal området i Afrika, optaget af “moderne” Homo sapiens i mindst 160.000 år før nutiden.
Klassiske Neanderthal-fossiler er blevet fundet over et stort område, fra det nordlige Tyskland til Israel og Middelhavslande som Spanien og Italien mod syd og fra England og Portugal mod vest og til Usbekistan mod øst. Hele dette område var formentlig ikke i brug for neanderthalerne på samme tid. Den nordlige grænse af deres udbredelsesområde må i særdeleshed være blevet formindsket i kolde perioder. På den anden side behøver den nordlige grænse af deres udbredelse, som repræsenteres af fossilfundene, ikke at være den egentlige nordgrænse af det område, de besatte, eftersom fund af mellem-palæolitisk udseende er blevet fundet endnu længere mod nord, op til 60 ° N, på den russiske slette. De seneste oplysninger har udvidet Neanderthal-området med ca. 2010 km østover ind i det sydlige Sibiriens Altai-bjerge.
Neanderthalernes anatomi var mere robust end det anatomisk moderne menneske .
Neanderthalerne forblev næsten udelukkende kødspisere og derfor øverst på fødepyramiden;. Men nye undersøgelser tyder på, at de kogte grøntsager i deres kost .
De lavede avancerede redskaber, de havde et sprog (hvis karakter dog debatteres) og de levede i komplekse sociale grupper.
Tidlige DNA-undersøgelser koncentrerede sig om mitokondrie-DNA (mtDNA), som på grund af strengt matrilineal arv (nedarvning via moderen) og deraf følgende sårbarhed overfor genetisk drift, er af begrænset værdi for vurderingen af muligheden for krydsning mellem neandertalere og Cro-Magnon mennesker. Med "genetisk drift" menes, at afvigende mitokondrier kan tabes, når de fordeles videre fra generation til generation.
I 1997 var genetikere i stand til at udtrække en kort sekvens af DNA fra Neanderthal knogler, der var 30.000 år gamle. .;.
I juli 2006 bekendtgjorde Max Planck Instituttet for Evolutionær Antropologi og 454 Life Sciences, at de ville sekvensbestemme hele Neanderthal-genomet i løbet af de næste to år.
Svante Pääbo har testet mere end 70 Neanderthal prøver. DNA-sekvensbestemmelse af et 38.000 år gammel knoglefragment af en lårbensknogle, der blev fundet i Vindija hulen, Kroatien, i 1980, viste, at Homo neanderthalensis og Homo sapiens deler ca. 99,5% af deres DNA.
Ud fra mtDNA analyserne anslår man, at de to arter havde en fælles forfader for ca. 500.000 år siden. I en artikel i tidsskriftet Nature har man beregne, at de to arter udskilte sig fra hinanden for ca. 516.000 år siden, hvorimod fossilfundene viser et tidspunkt på ca. 400.000 år siden.
Et studie fra 2007 skubber spaltningstidspunktet tilbage til for ca. 800.000 år siden. .Edward Rubin fra Lawrence Berkeley National Laboratory i Berkeley, Californien, har oplyst, at genomet hos neandertalere tyder på, at menneskelig-DNA og neanderthal-DNA er 99,5% - 99,9% identiske. ;.
Den 16. november 2006 udsendte Lawrence Berkeley National Laboratory en pressemeddelelse, hvor man foreslog, at neandertalerne og gamle mennesketyper formentlig ikke krydsedes (ikke havde sex med hinanden).
Edward M. Rubin, der er direktør for det amerikanske energiministeriums Lawrence Berkeley National Laboratory og Joint Genome Institute (JGI) , sekvensbestemte en brøkdel (0,00002) af nukleært-DNA (nDNA) fra en 38.000 år gammel lårbensknogle fra Vindia-Neanderthal. De beregnede den fælles forfader til at have levet for ca. 353.000 år siden, og en fuldstændig adskillelse af forfædrene til arterne kunne dateres tilbage til ca. 188.000 år siden. Deres resultater viser, at genomet fra moderne mennesker og fra neandertalere er mindst 99,5% identiske, men på trods af denne genetiske lighed, og på trods af, at de to arter levede i det samme geografiske område i tusinder af år, fandt Rubin og hans hold ingen tegn på nogen signifikant krydsning mellem de to. Rubin sagde: “Mens vi er ude af stand til endeligt at kunne konkludere, at krydsning mellem de to menneske-arter ikke skete, tyder analyser af det nukleare DNA fra Neanderthal tyder på, at der kun er ringe sandsynlighed for, at det skete i nogen nævneværdig grad.” ;.
I 2008 publicerede Richard E. Green et al. fra Max Planck Instituttet for Evolutionær Antropologi i Leipzig, Tyskland, hele sekvensen af Neanderthal mitokondrie-DNA (mtDNA) og foreslog, at “neandertalerne havde en langvarig, effektiv populationsstørrelse, der var mindre end populationsstørrelsen for det moderne menneske”.
James Morgan hævdede i en artikel i Nature om Green et al.‘s resultater, at mtDNA-sekvensen indeholdt tegn på, at neandertalerne levede i “små og isolerede populationer, og sandsynligvis ikke krydsede (havde sex) med deres menneskelige naboer.” .;.
I samme publikation blev det af Svante Pääbo oplyst, at i det tidligere arbejde ved Max Planck Instituttet “var forurening et problem,” og at de til sidst indså, at 11% af deres prøve var DNA fra moderne mennesker.
Siden da er mere af præparationsarbejdet blevet udført i rene omgivelser, og 4-basepar ‘tags’ er blevet tilføjet til DNA’et, så snart den er udvundet i ren-rummet i laboratoriet, således at Neanderthal-DNA kan identificeres.
Ud af de ca. 3 milliarder sekvensbestemte nukleotider viste analyser af ca. 1/3 ingen tegn på blanding mellem moderne mennesker og neandertalerne, ifølge Pääbo. Det var i overensstemmelse med et studie, som Noonan havde publiceret to år tidligere.
Den variant af microcephalin, der er almindelig uden for Afrika, og som var blevet foreslået at være af Neanderthal-oprindelse og ansvarlig for den hurtige hjernevækst hos mennesker, blev ikke fundet i neandertalerne.
Ej heller blev MAPT-varianten, som er en meget gammel variant, som primært findes i europæerne, fundet i neanderthalerne. ;.
Men en analyse af et første udkast til Neanderthal-genomet af den samme forskergruppe, og som blev publiceret i maj 2010, angav, at krydsning kan have fundet sted.
“De af os, der bor uden for Afrika, bærer en lille smule neandertal-DNA i os,” sagde Pääbo, der ledede studiet. “Andelen af Neanderthal-arvet genetisk materiale er ca. 1 til 4%.Det er en lille, men meget reel del af herkomsten i ikke-afrikanere i dag,” siger David Reich fra Harvard Medical School i Boston, som arbejdede på studiet. Denne forskning sammenlignede genomet i neandertalere med fem moderne mennesker fra Kina, Frankrig, Afrika syd for Sahara, og Papua Ny Guinea. Konklusionen var, at ca. 1 til 4% af generne hos ikke-afrikanerne kom fra neandertalerne, i forhold til basislinien, som blev defineret ved de to afrikanere.
Dette indikerer et genflow fra neandertalere til moderne mennesker, dvs. krydsning (sex) mellem de to populationer. Da de tre ikke-afrikanske genomer viser samme andel af Neanderthal sekvenser, skal krydsning i så fald have fundet sted tidligt i det moderne menneskers migration ud af Afrika, måske i Mellemøsten.
Intet bevis for genflow i retning fra moderne mennesker til neandertalerne blev fundet. Sidstnævnte resultat ville dog heller ikke være uventet, hvis kontakten fandt sted mellem en lille koloniserende bestand af moderne mennesker og en meget større fastboende befolkning af neandertalerne.
En meget begrænset mængde af krydsning kunne forklare resultaterne, hvis den indtraf tidligt nok i koloniseringsprocessen. ;.
Mens krydsning betragtes som den mest enkle fortolkning af de genetiske opdagelser, påpeger forfatterne, at de ikke endeligt kan udelukke et alternativt scenario, hvor kildebefolkningen af ikke-afrikanske moderne mennesker allerede var mere nært beslægtet med neandertalere end andre afrikanere var, på grund af gamle genetiske udspaltninger i Afrika. ;.
Blandt de gener, der har vist sig at variere mellem nutidens mennesker og neandertalerne, er RPTN, SPAG17, CAN15, TTF1 og PCD16. .;.
Neanderthal-fossiler kendes ikke fra tiden efter for 25.000 år siden. De sidste spor af Mousterian-kulturen (uden menneske-fund) er fundet i Gorham Hulen på afsides beliggende sydvendte kyst på Gibraltar, dateret til 30.000 - 24.500 år siden. Mulige scenarier er:.
Neanderthalerne var en selvstændig art fra moderne mennesker, og uddøde (på grund af klimaforandringer eller samspil med mennesker). De uddøde måske fordi Homo sapiens flyttede ind på neanderthalernes levested fra for ca. 80.000 år siden. Konkurrence fra Homo sapiens bidrog sandsynligvis til Neanderthalernes uddøen. Jared Diamond har foreslået et scenarie med voldelig konflikt og fordrivelse. ;
Det er muligt, at Neanderthalerne var en underart, der krydsede med Homo sapiens og forsvandt gennem absorption (krydsningsteori, interbreeding theory).
Vulkanske superudbrud for ca. 40.000 år siden, efterfulgt af endnu et vulkansk superudbrud et par tusinde år senere, er blevet foreslået som medvirkende årsag til Neanderthalernes tilbagegang, baseret på data fra Mezmaiskaya hulen i Kaukasus-bjergene i det sydlige Rusland
Mitokondrie-DNA (mtDNA) analyse af en neanderthaler fra Mezmaiskaya-hulen er dateret med kulstof-14 til at være ca. 29.000 år gammel. og dermed en af de senest levende Neanderthal individer.
Fylogenetisk analyse placerer de to neandertalere fra Kaukasus og vest-Tyskland sammen i en stamme, der er forskellig fra det moderne menneske, hvilket tyder på, at deres mtDNA-typer ikke har bidraget til det moderne menneskes mtDNA pool. .;.
I en mtDNA-baseret simulation af det moderne menneskes ekspansion i Europa med start for 1600 generationer siden (Neandertalerne angivet med lys grå farve), viser tilbagetrængning af neanderthalerne fra mellemøsten, senere fra østeuropa, endnu senere fra midteuropa og afslutning på den iberiske halvø. .
Som Paul Jordan noterer: “En naturlig sympati for underhunden og de dårligst stillede giver Neanderthal-folket en trist skæbne, uanset, hvor det skete.” Paul Jordan anfører, at der måske forekom krydsning til en vis grad, men at befolkninger, der forblev helt som Neanderthalere sandsynligvis blev udkonkurreret og marginaliseret til udryddelse af Aurignacians (en kultur, som fandtes for 45,000 - 35,000 år siden, karakteriseret ved hulemalerier, brug af smykker og bedre - for perioden - forarbejdede stenredskaber. De er navngivet efter Aurignac i Haute-Garonne området af Frankrig.) . .
For ca. 55.000 år siden begyndte klimaet at svinge vildt fra ekstrem kulde til mild kulde og tilbage i løbet af få årtier. Neanderthalernes krop var velegnet til at overleve i et koldt klima - deres relativ store brystkasse og tætbyggede lemmer opbevarede kropsvarme bedre end det var tilfældet hos Cro-Magnon.
Men de hurtige udsving i klimaet forårsagede økologiske ændringer, som neandertalerne ikke kunne tilpasse sig til. Klimaet skiftede så hurtigt, at de planter og dyr, som neanderthaleren som barn voksede op med, blev erstattet af helt andre planter og dyr i løbet af individets levetid. Neanderthalere, der havde udviklet en teknik med jagt ved at ligge i skjul, ville f.eks. have svigtende jagtheld, hvis skovene blev erstattet af græssletter. Et stort antal neandertalere døde formentlig i løbet af disse klimaudsving, der toppede for ca. 30.000 år siden. ;.
Undersøgelser af Neanderthalerens kropstrukturer har vist, at de havde brug for mere energi til at overleve end andre arter. Deres energibehov var op til 100 til 350 kalorier mere om dagen sammenlignet med et anatomisk moderne menneske (manden vejede i gennemsnit 68,5 kg, kvinder, den 59,2 kg).
Når maden blev knap, kan denne forskel have spillet en stor rolle for neandertalernes uddøen.
Test dig selv
Du kan tegne abonnement på BioNyt: Videnskabens verden her!
/Homo/Neanderthal/Det korte svar er: En række fund i Tyskland og Frankrig
Ferrassie-kraniet
• Neanderthal 1: Første Neanderthal eksemplar blev fundet under en arkæologisk udgravning i august 1856. Opdaget i et kalkbrud på Feldhofer grotten i Neanderthal i Tyskland. Fundet bestod af et kraniums hovedskal, to lårbensknogler, de tre højre armknogler, to af den venstre arms knogler, hofteben (ilium), og fragmenter af en scapula og ribben.
• La Chapelle-aux-Saints 1: Kaldet “den gamle mand”, et forstenet kranium fundet i La Chapelle-aux-Saints, Frankrig, af A. og J. Bouyssonie, og L. Bardon i 1908. Blandt andet et lavt hvælvet kranium og store øjebrynsbuer, der er typisk for neandertalerne.
Skønnes at være ca. 60.000 år gammelt. Individet havde alvorlig gigt og havde mistet alle sine tænder, med tegn på helbredelse. For at Eske Willerslev kunne leve videre sådan kræves, at nogen har lavet mad for ham, en af de tidligste eksempler på Neanderthal altruisme (svarende til Shanidar I.)
• La Ferrassie 1: et forstenet kranium fundet i La Ferrassie, Frankrig, af R. Capitan i 1909. Det anslås at være 70 tusind år gammel. Dens egenskaber omfatter en stor bule (occipital bun) i bagkraniet ud for lillehjernen), lavt hvælvet kranium og stærkt slidte tænder.
• Le Moustier: En forstenet kranium, som blev opdaget i 1909, i det arkæologiske område i Peyzac-le-Moustier, Dordogne, Frankrig. Mousterian-redskabskulturen er opkaldt efter Le Moustier. Kraniet, der skønnes at være mindre end 45.000 år gammelt, har en stor næsehule og en noget mindre udviklet øjebryns-hvælvning og bule (occipital bun) i bagkraniet ud for lillehjernen”, som man kunne forvente hos et ungt individ.
• Shanidar 1: Fundet i Zagros bjergene i det nordlige Irak, i alt ni skeletter, som menes at have levet i Mellem Ældre stenalder (middle paleolithic). En af de ni fund manglede en del af sin højre arm, og kan have været brækket eller amputeret. Fundet er også betydningsfuldt, fordi det viser, at stenredskaber var en del af kulturen her. Den ene var blevet begravet med blomster, hvilket viser, at en vis form for begravelsesceremoni kan være sket.
Kronologisk rækkefølge
Knogler med Neandertaler-træk i kronologisk rækkefølge. (Sorteret efter tid)
Blandet med H. heidelbergensis træk
•> 350.000 år siden: Sima de los Huesos c. 500-350.000 år siden
• 350-200.000 år siden: Pontnewydd 225.000 år siden.
• 200-135.000 år siden: Atapuerca, Vértesszöllos, Ehringsdorf, Casal de’Pazzi, Biache, La Chaise, Montmaurin, Prince, Lazaret, Fontéchevade
Typiske H. neanderthalensis træk
• 135-45.000 år siden: Krapina, Saccopastore, Malarnaud, Altamura, Gánovce, Denisova, Okladnikov Altai, Pech de l’Aze, Tabun 120.000 år siden - 100 ± 5.000 år siden, Qafzeh9 100, Shanidar 1 til 9 fra 80000 år siden til 60000 år siden , La Ferrassie 1 70.000 år siden, Kebara 60.000 år siden, Régourdou, Mt. Circeo, Combe Grenal, Erd 50.000 år siden, La Chapelle-aux Saints 1 60.000 år siden, Amud I 53 ± 8.000 år siden, Teshik-Tash.
• 45-35000 år siden : Le Moustier 45.000 år siden, Feldhofer 42.000 år siden, La Quina, l’Horus, Hortus, Kulna, Šipka, Saint Cesaire, Bacho Kiro, El Castillo, Bñnolas, Arcy-sur-Cure
• <35.000 siden Chatelperron, Figueria Brava, Zafaraya 30.000 år siden, Vogelherd 3, Vindija 32.400 ± 800 14C BP (Vi-208 31.390 ± 220, Vi-207 32.400 ± 1.800 14C BP) , Velika PECINA,
Blandet med træk fra det anatomisk moderne menneske
• <35.000 år siden_ Pestera cu Oase 35.000 år siden, Mladec 31.000 år siden, Pestera Muierii 30.000 år siden, Lagar Velho 24.500 år siden.
Test dig selv
Du kan tegne abonnement på BioNyt: Videnskabens verden her!
/Homo/Neanderthal/Denisovan/Sex/Det korte svar er: Måske
Eske Willerslevs gruppe fra Københavns Universitet har i aboriginerens genom fundet evidens for, at aborigin-forfædrene på vejen mødte den mennesketype, som kaldes "Denisovans" eller "Denisova-homininer" (ref.9567).
Test dig selv
Du kan tegne abonnement på BioNyt: Videnskabens verden her!
/Homo/Neanderthal/Denisovan/Sex/Det korte svar er: En slags neanderthaler
Det er en slags Neandertalere, som må have forladt Afrika langt tidligere end det moderne menneske udvandrede fra Afrika. Denisovan-mennesket er kun identificeret ud fra noget 30.000-50.000-årigt DNA, der stammer fra én fingerknogle fundet i en hule i Sibirien. Denisovans var en slags basal-Neandertalere. Ifølge hvad man foreløbig ved om deres DNA var der genetisk ikke større forskel på Denisovans og en klassisk Neandertaler, end der i dag er mellem en "Han-talende kineser" og en "buskmand". Det er blevet fremlagt, som om Denisovans er mere forskellige fra Neandertalere, og at de derfor måske skal opfattes som en særlig art, men der gik medie-spin i den historie, mener Eske Willerslev. Hvis man laver et genetisk træ over Neandertalerne, så falder Denisovans meget tæt på Neandertalerne ifølge David Reich's studier i Nature. Splittet mellem Denisovans og Neandertalerne er cirka den samme, som mellem afrikanere og alle ikke-afrikanere blandt de nulevende mennesker. De har været væk fra hinanden i mange år, men det er svært at sige, at de var noget andet end Neandertalere. I marts 2010 blev det offentliggjort, at benrester (blot et enkelt knogle-fragment) af et ungt individ, der levede for ca. 41.000 år siden, var blevet fundet i Denisova-hulen (Altai Krai, Rusland, tæt på Mongoliet), som er et område, hvor der på cirka samme tid også boede Neandertalere og det moderne menneske (arboriginernes forfædre) . Denisova-homininerne er efterkommere af en udvandring fra Afrika, der hidtil har været ukendt. Derudover kender man mindst 4 kendte udvandringer, som blev foretaget af henholdsvis 1) Homo erectus, 2) Homo heidelbergensis / neandertaleren og (i mindst to udvandringsbølger) , det moderne menneske 3) ,4) Homo sapiens (hvoraf de første europæere fra for 35.000 år siden traditionelt kaldes CroMagnon). Denisova- homininerne kan være endnu en udvandringsbølge, der måske skete for ca. 1 mill. år siden (ref.9568, ref.9567), og som nåede helt til Sibirien, før den uddøde.
Eske Willerslevs gruppe fra Københavns Universitet på Københavns Universitet har i abori-ginerens genom fundet evidens for, at aborigin-forfædrene på vejen mødte den mennesketype, som kaldes Denisovans eller "Denisova-homininer" (ref.9567). Det er en slags neanderthalere, som allerede var derude, og som må have forladt Afrika langt tidligere.
Denisovan-mennesket er kun identificeret ud fra noget 30.000-50.000-årigt DNA, der stammer fra én fingerknogle fundet i en hule i Sibirien.
Denisovans er en slags basalneanderthalere. Ifølge hvad man foreløbig ved om deres DNA var der genetisk ikke større forskel på en denisovans og en klassisk neanderthaler, end der i dag er mellem en "hankineser" og en "busk-mand". Det er blevet fremlagt, som om denisovans er mere forskellige fra neanderthalere, og at de derfor måske skal opfattes som en særlig art, men der gik medie-spin i den historie, mener Eske Willerslev.
Hvis man laver et genetisk træ over neanderthalerne, så falder Denisovans meget tæt på neanderthalerne ifølge David Reich's studier i Nature. Splittet mellem Denisovans og neander-thalerne er cirka den samme, som mellem afrikanere og alle ikke-afri-kanere blandt de nulevende menne-sker. De har været væk fra hinanden i mange år, men det er svært at sige, at de var noget andet end neander-thalere.
I marts 2010 blev det offentliggjort, at benrester (blot et enkelt knogle-fragment) af et ungt individ, der levede for ca. 41.000 år siden, var blevet fundet i Denisova-hulen (Altai Krai, Rusland, tæt på Mongoliet), som er et område, hvor der på ca. samme tid også boede neanderthalere og det moderne menneske (arboriginernes forfædre).
Denisova-homininerne er efterkommere af en udvandring fra Afrika, der hidtil har været ukendt. Derudover kender man fem kendte udvandrin-ger, som blev foretaget af henholdsvis Homo erectus, Homo heidelbergensis, neanderthaleren og (i mindst to udvandringsbølger) det moderne menneske Homo sapiens sapiens (hvoraf de første europæere fra for 35000 år siden traditionelt kaldes CroMagnon). Denisova-homininerne kan være endnu en udvandringsbølge, der måske skete for ca. 1 mill. år siden (ref.9568), og som nåede helt til Sibirien, før den uddøde.
Test dig selv
Du kan tegne abonnement på BioNyt: Videnskabens verden her!
/Homo/Neanderthal/Klima/Det korte svar er: Vildt svingende i temperatur
For ca. 55.000 år siden begyndte klimaet at svinge vildt fra ekstrem kulde til mild kulde og tilbage i løbet af få årtier. Neandertalernes krop var egnet til at leve i et koldt klima: Stor brystkasse og tætbyggede lemmer holdt bedre på kropsvarmen end hos Cro-Magnon. Men udsvingene i klimaet skiftede så hurtigt, at de planter og dyr, som Neandertaleren som barn voksede op med, blev erstattet af helt andre planter og dyr i løbet af individets levetid på stedet. Neandertalere, der havde udviklet en teknik med jagt ved at ligge i skjul, ville f.eks. have svigtende jagtheld, hvis skovene blev erstattet af græssletter. Et stort antal neandertalere døde formentlig i løbet af disse klimaudsving, der toppede for ca. 30.000 år siden (ref.9569 note 71). Undersøgelser af Neandertalerens kropstrukturer har vist, at de havde brug for mere energi til at overleve end andre arter. Deres energibehov var op til 100 til 350 kalorier mere om dagen sammenlignet med et anatomisk moderne menneske (Neandertal-manden vejede 68,5 kg, kvinden vejede 59,2 kg, i gennemsnit). (ref.9569 note 72). Når der blev knaphed på føde kan denne forskel have spillet en stor rolle for neandertalernes uddøen (ref.9569note 71).
Test dig selv
Du kan tegne abonnement på BioNyt: Videnskabens verden her!
/Homo/Neanderthal/Sex/Det korte svar er: Det er en mulighed, men ikke endelig bevist
Et alternativ til teorien om neandertalernes uddøen er at neandertalerne blev absorberet i Cro-Magnon befolkningen ved krydsning.
Dette vil være i modstrid med strenge versioner af teorien om “seneste afrikanske oprindelse” (Recent African Origin,), da det ville betyde, at i det mindste en del af genomet af europæerne ville nedstamme fra neandertalere.;.
De mest højlydte fortalere for hybridiseringshypotesen er Erik Trinkaus fra Washington University.
Erik Trinkaus antager, at forskellige fossiler er hybrid-enkeltpersoner, heriblandt “ Lagar Velho barnet” (“child of Lagar Velho), et skelet fundet ved Lagar Velho i Portugal og dateret til at være ca. 24000 år gammelt.
De undersøgte genomer af ikke-afrikanere omfatter dele, som er blevet beskrevet som “af Neanderthal-oprindelse”, på grund af krydsning mellem neandertalerne og forfædre til eurasiater i det nordlige Afrika eller Mellemøsten før deres spredning.
I stedet for absorption af Neanderthalpopulationerne, synes denne genflow at have været af begrænset varighed og af begrænset omfang.
Det anslås til 1 til 4% af DNA’et i europæere og asiater (i studiet fransk, kinesisk og Papua) er ikke-moderne DNA, som deles med det gamle neanderthal-DNA i stedet for med Sub-Sahara afrikanere (i studiet Yoruba og San).
Test dig selv
Du kan tegne abonnement på BioNyt: Videnskabens verden her!
/Homo/Neanderthal/Sex/Det korte svar er: Det er en mulighed, men ikke endelig bevist
Navnet Homo neanderthalensis, er en angivelse som en selvstændig art, blev foreslået af den britisk-irske geolog William King i 1864. Dette har derfor forrang for et forslag fremsat i 1866 af Ernst Haeckel, nemlig Homo stupidus. .Den praksis, blot at omtale arten som “neandertalerne”, opstod i populærlitteratur i 1920’erne.
Forskere har diskuteret, om neandertalerne skal klassificeres som Homo neanderthalensis eller “Homo sapiens neanderthalensis”, sidstnævnte navn ville placere neandertalere som en underart af Homo sapiens. Nogle morfologiske undersøgelser støtter den opfattelse, at Homo neanderthalensis er en selvstændig art og ikke en underart.
Hvis man antager, at der har været krydsning mellem de to og at det har givet frugtbart afkom, kan det tale for at klassificere neanderthaleren som en underart af Homo sapiens, idet arter er defineret ved reproduktiv isolation fra andre arter.
Test dig selv
Du kan tegne abonnement på BioNyt: Videnskabens verden her!
/Homo/Neanderthal/Sex/Det korte svar er: Måske
Mange steder kan man læse - som var det et fastslået faktum - at der er ca. 2% neanderthal-DNA i alle moderne mennesker, eller 1-4%.
"Det kan man ikke sige", mener Eske Willerslev. "Vi ved det faktisk ikke - og det har noget at gøre med, at det, som man kalder neanderthal-signalet (altså DNA-ligheder mellem neanderthaler-DNA og vores DNA) kan være kommet ved et genflow.
I den nævnte 2%-teori går man ud fra, at det moderne menneske, der vandrer ud af Afrika, møder neander-thalere og har sex med dem, inden de spreder sig videre ud i verden. Problemet er bare, at man faktisk også kan forklare DNA-lighederne mellem neanderthalere og os på en anden måde: At neanderthaleren og det moderne menneske i Afrika har et fælles udspring," siger Eske Willerslev.
"Med de data, der i dag er til rådighed, kan man ikke skelne mellem de to teorier. Neanderthal-2%-sex-teorien går verden rundt som et forskningsresultat, fordi det er en meget bedre historie. Det er så simpelt - videnskab er ikke mere hellig end som så. Det vil sige, at det er den teori, der bliver favoriseret. Men i virkeligheden kan man ikke sige det, baseret på de data, der er til rådighed," siger han.
Ligesom andre befolkninger uden for Afrika, har den australske aboriginal-mand måske visse små stumper af sit genom fra neandertalerne, - eller disse små genom-stykker stammer fra før neanderthal-udskillelsen.
Et alternativ til teorien om neandertalernes uddøen er at neandertalerne blev absorberet i Cro-Magnon befolkningen ved krydsning.
Dette vil være i modstrid med strenge versioner af teorien om “seneste afrikanske oprindelse” (Recent African Origin,), da det ville betyde, at i det mindste en del af genomet af europæerne ville nedstamme fra neandertalere.
De mest højlydte fortalere for hybridiseringshypotesen er Erik Trinkaus fra Washington University.
Erik Trinkaus antager, at forskellige fossiler er hybrid-enkeltpersoner, heriblandt “ Lagar Velho barnet” (“child of Lagar Velho), et skelet fundet ved Lagar Velho i Portugal og dateret til at være ca. 24000 år gammelt.
De undersøgte genomer af ikke-afrikanere omfatter dele, som er blevet beskrevet som “af Neanderthal-oprindelse”, på grund af krydsning mellem neandertalerne og forfædre til eurasiater i det nordlige Afrika eller Mellemøsten før deres spredning.
I stedet for absorption af Neanderthalpopulationerne, synes denne genflow at have været af begrænset varighed og af begrænset omfang.
Det anslås til 1 til 4% af DNA’et i europæere og asiater (i studiet fransk, kinesisk og Papua) er ikke-moderne DNA, som deles med det gamle neanderthal-DNA i stedet for med Sub-Sahara afrikanere (i studiet Yoruba og San).
Test dig selv
Du kan tegne abonnement på BioNyt: Videnskabens verden her!
/Homo/Redskab/Det korte svar er: Øget hjerne og reduceret tandstørrelse
Homo har reduceret tandstørrelse - som kombineret med øget hjernestørrelse er tilpasset mere varieret, omnivor ("altædende") fødeindtagelse, af høj næringsværdi, under skiftende miljøforhold - og afhængig af socialt familie/gruppe-liv. Homo rudolfensis (opkaldt efter Rudolf-søen, en ældre betegnelse for Turkana-søen i nord-Kenya) anses for at være stamform til senere Homo-arter på grund af sit større hjernerumfang i forhold til Homo habilis (ref.9664).
Test dig selv
Du kan tegne abonnement på BioNyt: Videnskabens verden her!
/Homo/Redskab/Det korte svar er: For 2½ mill. år siden
De "tidlige Homo'er" var de første redskabsmagere (ref.9665). Redskaberne var allerede i brug for 2,5 mill. år siden samtidig med indskrænkningen af skoven (ref.9666). (Man har forsøgt at lære verdens måske mest intelligente chimpanse, dværgchimpansen Kanzi, at fremstille stenredskaber som i den simple Oldowan-kultur, men det kan den ikke lære - selv om den var i stand til at lære et omfattende engelsk ordforråd (ref.9667). Evnen til at lave redskaber kan være udviklet i konkurrence med de "robuste sydaber", der satsede på store tænder i stedet for store hjerner (ref.9668).
Test dig selv
Du kan tegne abonnement på BioNyt: Videnskabens verden her!
/Homo/Redskab/Det korte svar er: Nye fødeemner
Redskaber betød adgang til nye fødeemner, såsom kød og knoglemarv. Hjernen hos nyfødte lægger beslag på 75% af organismens energiproduktion (20% hos voksne) (ref.9669). Kød var derfor en nødvendighed, men også adgang til dyr i vand (muslinger, krebsdyr, skildpadder, fisk og fugleæg) (ref.9670). Fedt udgør 60% af hjernens vægt, og skaldyr og fisk er fedtrige, og med en fedtsammensætning, som minder om hjernens. (Kilden til disse fedtstoffer var delvis alger).
Test dig selv
Du kan tegne abonnement på BioNyt: Videnskabens verden her!
/Homo/Redskab/Det korte svar er: Forsinket modning
Når Homo-arterne kunne udvikle en stor hjerne skyldtes det dels hurtig hjernevækst i fosterlivet, dels fortsat hjernevækst længe efter fødslen - ledsaget af forsinket modning, og hjælpeløshed i længere tid end hos alle andre pattedyr. Dette var muligt, fordi Homo'erne ikke var afhængige af træer som sikkerhed. Hjerneudviklingen kompenserede for disse problemer, bl.a. ved udvikling af sprog og en samarbejdskultur. Når kvinder i dag lever længere kan det være en tilpasning til, at bedstemødrene skulle kunne hjælpe de unge mødre.
Test dig selv
Du kan tegne abonnement på BioNyt: Videnskabens verden her!
/Homo/Redskab/Det korte svar er: Chimpanserne
I dag er chimpanse-arterne tættere beslægtet med mennesket end med de andre store menneskeaber (gorilla'er) (ref.9671). Genetisk er 99,2% af de aktive gener ens hos chimpansen og mennesket (ref.9672). Hvis hele DNA'et sammenlignes, er der 98,75% overensstemmelse(ref.9673).
Test dig selv
Du kan tegne abonnement på BioNyt: Videnskabens verden her!
/Homo/Redskab/Det korte svar er: Bevarer ungdomstræk længe. øget hjerne
De fleste af de træk, der adskiller mennesket, er anatomisk unikke karakterer hos mennesket. Den store forskel er i aktiviteten af gener, som styrer væksten af forskellige legemsdele i barndommen, og som bevarer ungdommelige træk i det voksne menneske (neoteni). Visse gener ændrede sig hurtigt ved menneskes udvikling, f.eks. gener af betydning for hjernens funktion, sprogudviklingen, kønscelledannelsen, immunforsvaret, lugtesansen, høresansen, proteinnedbrydningen (så fødens proteinnedbrydning i tarmen blev hurtigere, da kød blev føde for 2,5 mill. år siden) og kæbemuskelstyrken svækkedes for 2,4 mill. år siden, da hjernen øgedes (ref.9674). . Genetisk er 99,2% af de aktive gener ens hos chimpansen og mennesket (ref.9672). Hvis hele DNA'et sammenlignes, er der 98,75% overensstemmelse(ref.9673)
Test dig selv
Du kan tegne abonnement på BioNyt: Videnskabens verden her!
/Homo/Opret_gang/Det korte svar er: For over 3 mill. år siden
I Østafrika eller måske i Chad 2500 km længere mod vest opstod en ny type primat, der gik på to ben, for derved var den mere egnet til de nu mere spredte fødekilder, idet den nu kunne bevæge sig hurtigere (ref.9656). Eksempler er "Lucy" og "Lucy's baby" for henholdsvis 3,2 og 3,3 mill. år siden; de opfattes begge som sydabearten Australopithecus afarensis. Syd Sydaberne var afhængige af træer for at komme i sikkerhed (og de uddøde, da træerne blev sjældne på grund af tørke). Sydaberne brugte ikke stenredskaber, kendte ikke til ild, havde svage hjørnetænder, korte ben og ringe løbeevne. Dog var australopithecinere (dvs. slægterne Ardipithecus og Australopithecus) ofte nede på jorden - de gik på to ben (ref.9657) : I 1978 fandt Mary Leakey 3,6 mill. år gamle fodspor af 3 individer, der kan følges over 25 meter, opfattet som en far med barn forrest og en efterfølgende kone.
Test dig selv
Du kan tegne abonnement på BioNyt: Videnskabens verden her!
/Homo/Opret_gang/Det korte svar er: Ændring rygradstilling, bækken, fod, lemmer
I forhold til bavianer, der går på alle fire ben og de afrikanske menneskeaber, der bruger kno-gang, kræver oprejst gang meget væsentlige anatomiske ændringer i udformningen af foden (tabt fod-gribefunktion), bækkenet (øget vægtbærende evne), rygsøjlen (S-formet og mere robust) og kraniet (fremad-forskudt nakkehul) og i længdeforholdet mellem (kortere) arme og (længere) ben (ref.9658).
Test dig selv
Du kan tegne abonnement på BioNyt: Videnskabens verden her!
/Homo/Opret_gang/Det korte svar er: Det dobbelte af mennesket
En chimpanse, der går, bruger dobbelt så meget energi som et menneske, der går (ref.9659).
Test dig selv
Du kan tegne abonnement på BioNyt: Videnskabens verden her!
/Homo/Opret_gang/Det korte svar er: Af flere grunde, bl.a. varmeregulering, bevægelsesfrihed mv.
Opret holdning kan være opstået af en eller flere af disse grunde: For at kunne nå frugter i småbuske (ref.9660), få bedre udsyn mod farer og føde, opnå større dominans i gruppen, bedre regulering af legemstemperaturen (idet opret gang giver mindre solbeskinnet kropsareal og i 1-2 meters højde over jordoverfladen kan temperaturen være 20 grader lavere, foruden at det blæser mere i denne højde og derved mindskes behovet for vandtabende svedafgivelse (ref.9661). Evnen til at regulere varmen via sved blev dog udviklet dramatisk hos den fuldt savannetilpassede Homo ergaster, kombineret med slank, høj kropsbygning og tab af behåring, som ellers ville hindre svedfordampningen (ref.9662). (En udviklingsretning som reaktion på tørt klima for 2½ mill. år siden var arter med lille hjerne og kraftigt tyggeapparat tilpasset seje, hårde plantedele. Disse "robuste sydaber" klarede ikke den tiltagende tørke for 1 mill. år siden). Hvornår udvikledes Homo-slægten? Homo-slægten med den voksende hjerne opstod for 2,5 mill. år siden omkring det tidspunkt, hvor Jorden som følge af kontinentaldrift med bjergkædedannelser var blevet så kold, at der i perioder opstod istider med iskapper ved polerne, bl.a. Antarktis- nedisningen for 3 mill. år siden, og hvor der i tidsmæssige ryk skete savannedannelser i den østafrikanske riftzone, der dengang var løftet 1 km højere end i dag, og derfor køligere og mere fugtig (ref.9663).
Test dig selv
Du kan tegne abonnement på BioNyt: Videnskabens verden her!
/Homo/Opret_gang/Det korte svar er: For måske 2 mill. år siden
For omkring 2 mill. år siden dukkede arten Homo ergaster ("det arbejdsomme menneske") op i Østafrika - med en markant større hjerne end "de tidlige Homo'er", og mere tydelig asymmetri i hjernen end hos "tidlig Homo" (ref.9675), kropsvægt på 63 kg (mod 30-50 kg hos "tidlige Homo'er"), og Homo ergaster var 180 cm høje og slanke og med længere ben end arme, opret gang, udholdende løbere - dvs. alle tegn på, at livet i træer var slut. Et eksempel på Homo ergaster er Turkana-drengen fra for 1,5 mill. år siden. (Tidligere blev Homo ergaster kaldt for Homo erectus - men dette navn bruges nu kun om former i Asien, der er udviklet fra Homo ergaster).
Test dig selv
Du kan tegne abonnement på BioNyt: Videnskabens verden her!
/Immunsystem/Gen/Det korte svar er: I færdigudviklede celler findes der i immunsystemets gener tre typer gener og de mulige konformationer vil kunne give 50 x 4 = 200 forskellige konformationer.
I immunsystemet findes der såkaldte antistoffer. Antistoffer er proteiner, som beskytter os mod forskellige mikroorganismer. Der er flere forskellige mikroorganismer og fremmedstoffer end der er forskellige gener hos mennesket. Man kan så spørge, hvordan der kan laves lige så mange forskellige antistoffer (som altså er proteiner), når der kun er et begrænset antal gener?
I færdigudviklede celler findes der i immunsystemets gener tre typer gener. De kaldes J (Joining) gener, V (Variable) gener og C (Konstante) gener. Der findes 40 forskellige V-gener, fem forskellige J-gener og et enkelt C-gen. De kaldes J-gener, fordi de samler V- og C-generne. De mulige konformationer vil kunne give 50 x 4 = 200 forskellige konformationer. Dette gælder kun for den type af aminosyrekæde på antistofferne, som kaldes κ (kappa) let-kæden. Der findes også en tilsvarende λ(lambda) let-kæde, som vil kunne give 120 forskellige let-kæde konformationer. Ydermere findes der også tunge kæder, som indeholder 27 forskellige D-gener og 51 V-gener og 6 J-gener. Og dermed kan der i alt dannes 27 x 51 x 6 = 8262 forskellige tung-kæde konformationer. Alt i alt kan der med 320 forskellige lette kæder og 8262 forskellige tunge kæder dannes 320 x 8262 = 2.643.840 forskellige antistoffer.
Dog kan der forekomme endnu mere diversitet ved såkaldte somatiske mutationer.
Test dig selv
Du kan tegne abonnement på BioNyt: Videnskabens verden her!
/Kloning/Det korte svar er: En form for ukønnet formering
Kloning er en form for ukønnet formering. Dette sker ved at overføre en kerne (med arvemateriale) fra en celle til en anden (oftest en ægcelle). Kloning blev først et stort emne, da man i 1997 klonede fåret Dolly ved kerneoverførsel.
Kloning er i dag et potentielt redskab til at gøre mange menneskets liv nemmere, bl.a. ved at fremstille forskellige klonede organer eller dele af organer, f.eks. hudstykker osv. Men som ved så mange andre af de bioteknologiske metoder er der også ved kloning en række etiske overvejelser, der skal gøres. I Danmark findes et Etisk Råd, som diskuterer, hvad der bør være lovligt og ikke lovligt. Med hensyn til kloning kan Etisk Råds meninger ses her.
Test dig selv
Du kan tegne abonnement på BioNyt: Videnskabens verden her!
/Landbrug/Det korte svar er: For ca. 10.000 år siden, men forskelligt i forskellige områder af verden
En jagt- og samlerkultur investerer langt mindre ressourcer i at skaffe føde end en landbrugskultur, som skal bearbejde jorden, så, vande, gøde, beskytte området og høste. Dette blev derfor først valgt, når de naturlige ressourcer på stedet ikke mere slog til, og man ikke kunne vandre til et andet område som nomade (ref.9748). De fleste steder kom husdyrhold først 500 år senere end agerbrug (ref.9749). Domesticering svarer til dannelse af nye arter - for husdyrene kan typisk ikke klare sig i naturen. Dyrkede planter ændres også drastisk. Medens vildtvoksende planter kun kan spire fra de frø, som ikke samles af dyr eller mennesker, så har avlede planter størst mulighed for, at netop de indsamlede frø formerer sig. Derfor vil de planter, der danner flest frø, og undlader at kaste dem før mennesket når at høste dem, have størst chance for at bringe arten videre. Græsser med seje aks får en fordel frem for græsser, hvor akset let knækker. Hvis planten har store frø, bliver disse også i højere grad indsamlet af mennesket - det samme gælder, hvis frøene sidder tæt sammen, og hvis der er en tynd frøhvide - der lettere nedbrydes i tarmen (ref.9750). Beskyttelse af (hus) dyr og avl af planter fremkalder altså straks store genetiske ændringer hos disse dyr og planter - uden at mennesket på nogen bevidst måde skal tage stilling til udvælgelsen af de bedst egnede individer for menneskets behov. De første agerbrug blev etableret i Levanten. Det skete for ca. 11.500 år siden (ref.9751). Mejeriprodukter blev en del af kosten for 5-6.000 år siden (ref.9752). I en bosættelse, der blev etableret for ca. 9000 år siden og som fungerede i 800-1000 år (indtil jorden var udpint), var der i perioder op til 10.000 indbyggere (Catal Hüyük i det sydvestlige Asien). Her var også et vidtforgrenet handelsnet. Langs Yangtze-floden i Kina startede risdyrkningen for 9000 år siden, og langs Den Gule Flod i Kina startede dyrkningen af tørketålende hirse for 8500 år siden (ref.9753). Ris leverer i dag stadig en femtedel af menneskehedens kalorier (ref.9754). Kartofler og majs lærte de oprindelige indianere i Amerika at dyrke - foruden at de dyrkede 300 andre plantearter (ref.9755).
Test dig selv
Du kan tegne abonnement på BioNyt: Videnskabens verden her!
/Landbrug/Syndfloden/Det korte svar er: Sortehavet, som for 15.000 år siden var blevet dannet af istidens smeltevand, men som for 7500 år siden gennembrød Bosporus-dæmningen
Myter opstod, som bl.a. myten om Syndfloden. Den baserer sig på Sortehavet, som for 15.000 år siden var blevet dannet af istidens smeltevand, men som for 7500 år siden gennembrød Bosporus-dæmningen, så vandet oversvømmede landbrugsområderne i løbet af få årtier. Den brusende stormflod ved Bosporus har varet mindst 300 dage og man har kunnet høre den 500 km væk (ref.9756). Hele samfund blev opgivet og kysterne ved Sortehavet blev ikke opdyrket igen. Mindet om hændelserne levede videre hos de folk, som flygtede til Sumer, fordi de årlige, naturlige oversvømmelser af Eufrat og Tigris på dette sted mindede folk om "Den Store Oversvømmelse" - som ifølge Syndflod-myten "druknede alle mennesker undtagen en familie, hvorfra vi alle nedstammer" (ref.9757).
Test dig selv
Du kan tegne abonnement på BioNyt: Videnskabens verden her!
/Protein/Det korte svar er: En kæde af aminosyre-enheder
Protein er et græsk ord. Proteiner er afgørende for alt liv. Ordet kommer fra græsk af et ord, som betyder “primært” eller “først”. Proteiner danner øjenlinsen, vedligeholder og beskytter vores DNA, opbygger nerveimpulserne, nedbryder og genbruger molekylerne fra maden, danner byggestene i kroppen, og er motorerne i vores muskler.
Protein opbygges af aminosyrer, 20 forskellige. De består af et skelet af hydrogen, kulstof, oxygen og nitrogen samt R-grupper (radikaler), der også består af disse grundstoffer men for enkelte aminosyrers vedkommende også indeholder svovl. Aminosyrerne sættes sammen - en efter en - under fraspaltning af vand. Aminosyrekæden kan består af fra ganske få aminosyrer til flere tusinde aminosyrer. Sammensætningen af aminosyrer i den korrekte rækkefølge sker i cellens proteinfabrikker, ribosomerne, på baggrund af instruktioner fra RNA, der er kopier af dele af vores DNA.
Kernemagnetisk resonansspektroskopi (NMR Nuclear Magnetic Resonance) er den vigtigste teknik til at bestemme proteiners tredimensionelle struktur
Forskerne ved endnu ikke, hvordan cellen er i stand til at styre, hvilken funktion et ikke-foldet protein skal have - altså f.eks. for prolaktin-receptorens vedkommende, hvordan cellen sikrer, at den ikke giver sig til at vokse, hvis det faktisk er produktion af mælk, der er behov for.
Test dig selv
Du kan tegne abonnement på BioNyt: Videnskabens verden her!
/Protein/3d/Prolaktin/Det korte svar er: Ikke nødvendigvis
Proteiners veldefinerede 3D-struktur har været et dogme, men der er nu sket et dogme-skift om proteiner på dette punkt.
Op mod en tredjedel af menneskets proteiner har nemlig ingen veldefineret tre-dimensionel struktur. Det faktum har rystet og udfordret den naturvidenskabelige verden. Der er tale om et dogme-skifte inden for proteinforskningen.
I vores cellers cellemembran sidder et protein, der er receptor-landingsplads for hormonet prolaktin, som blandt meget andet har betydning for mælkeproduktion i brystkirtlerne. Prolaktinreceptoren sidder i cellemembranen, men den har en ikke-foldet “hale”, der rager ind i cellens indre.
Aktivering af proteinreceptoren kan medføre meget andet end mælkepro-duktion, faktisk måske over 300 for-skellige funktioner i cellen. Hvilken funktion, der bliver resultatet af akti-veringen, skyldes bl.a. denne hale.
Prolaktin-receptoren rager også ud på ydersiden af cellen og (som navnet siger) kan prolaktin-hormonproteinet binde til dette sted.
I halepartiet kan et såkaldt JAK2-protein bindes til receptorens ikke-foldede hale inde i cellen, hvorved der sker ændringer i receptorens struktur. Det ændrer formen på receptorens hale, og dermed synliggøres områder i receptorens hale, så andre proteiner nu kan genkende og binde til dette sted.
Et protein kaldet JAK2 er altid bundet til receptorens “hale”, der altså vender ind i cellen og er ufoldet. Bindingen af JAK2 er i sig selv ikke nok til at medføre nogen funktion i cellen. Men når prolaktin-hormonet (der også er et protein) binder til receptoren på ydersiden af cellen, ændres strukturen af receptoren - også receptordelen inde i cellen - hvilket gør det muligt for forskellige proteiner at genkende og binde til den ikke-foldede del inde i cellen. Det kan medføre, at en hel vifte af forskellige funktioner sættes i gang; f.eks. at cellen får besked om at vokse, at producere mælk, eller at dele sig.
Prolaktin-hormonproteinet og JAK2-proteinet er altså de nøgler, der skal til for at synliggøre én af de mange potentielle former, som receptorens indre hale kan antage.
Hvordan den endelige form kommer til at se ud (og dermed hvilken funktion der igangsættes i cellen), afhænger sandsynligvis af, hvilket protein der dernæst binder til receptorens hale. Hvis cellen skal vokse, er det ét protein, der skal binde til receptorens hale, men hvis cellen skal producere mælk, er det et helt andet protein, der skal binde til receptorens hale.
Proteiner består af lange kæder af aminosyrer. Man har hidtil antaget, at disse kæder umiddelbart efter proteinets dannelse folder sig sammen til en veldefineret garnnøgle-lignende struktur, og at denne foldede, tredimensionelle struktur er altafgørende for proteinets funktion.
Dette kan ikke være den eneste sand-hed, når det nu viser sig, at op mod en tredjedel af alle proteiner i vores krop er ikke-foldede. For de ikke-foldede proteiner varetager nemlig mange vigtige funktioner i cellen, har det vist sig - og altså åbenbart funktioner, som ikke er dikteret af nogen bestemt form.
Ikke-foldede proteiner kan binde til mange forskellige proteinpartnere. Alt efter hvilket protein, der binder til det ikke-foldede protein, kan dette hidtil ikke-foldede protein antage et væld af forskellige former. Dette betyder, at et ikke-foldet protein kan varetage mange forskellige funktioner i cellen.
I 1894 opstillede Emil Fischer en “lås-og-nøgle-teori” for den type proteiner, som har enzymfunktion (enzymer er en gruppe af proteiner, som katalyserer en kemisk reaktion). Teorien går ud på, at strukturen af enzymet ikke ændres, når enzymproteinet binder til dets substrat (ligesom formen på en lås ikke ændres ved at stikke nøglen ind og låse døren op). Det betyder, at strukturen af enzymet bestemmer enzymets funktion – der er kun én nøgle, der kan låse døren op. Ifølge denne teori kan ethvert enzym i princippet kun binde til ét substrat.
Dette meget statiske billede af proteiners samspil med andre molekyler er blevet noget opblødt gennem tiden 30 BioNyt - Videnskabens Verden nr.154, www.bionyt.dk
- men det har vedvarende været et centralt dogme blandt proteinforskere, at et proteins 3D-struktur dikterer dets funktion.
Et ikke-foldet protein er ikke et langt, snorlige molekyle. Det er et meget dynamisk (livligt) molekyle, der hurtigt kan antage mange forskellige udstrakte former - som halen på en hund, der logrer ivrigt. Når et ikke-foldet protein binder til et andet protein, dannes der pludselig struktur i det hidtil ikke-foldede protein: Det antager nu én bestemt form. Hvis det samme ikke-foldede protein i stedet binder til et andet protein, vil det kunne antage en anden, fast form. Dermed kan proteinet få en anden funktion.
Det er pladsbesparende for cellen, at samme protein således kan varetage forskellige former, og dermed forskellige funktioner.
Sommetider vil et protein binde til det ikke-foldede protein, uden at dette medfører nogen funktion i cellen - men bindingen kan danne grundlag for, at andre proteiner nu kan genkende proteinet, binde til det og dermed udføre en bestemt funktion.
Man kan sige, at den første nøgle er med til at forme og dermed synliggøre låsen, sådan at den anden nøgle kan låse op. (Det kan sammenlignes med de hoveddøre, der kræver to nøgler for at åbnes, bortset fra at inde i cellen kommer den anden lås først til syne, når den første nøgle er sat i døren).
Nogle ikke-foldede proteiner kræver mere end to proteiners binding, før den rette funktion kan udføres.
Test dig selv
Du kan tegne abonnement på BioNyt: Videnskabens verden her!
/Sygdom/Sjældne_super/Det korte svar er: Genetiske sygdomme, som kun kendes hos ca. 100 personer i verden eller færre
Sjældne sygdomme skyldes ofte noget genetisk. Definitionen på en sjælden sygdom er, når den rammer færre end 200.000 mennesker verden over. Hvis færre end 100 rammes af sygdommen, kaldes den en “supersjælden sygdom”.
5-årige Marisa Carney på Long Island i USA lider af en supersjælden sygdom. Hun har hjernesygdommen ROHHAD, som giver hurtig fedme og livstruende vejrtrækningsproblemer. Der kendes 75 tilfælde på ver-densplan.
I 2007 beskrev Debra Weese-Mayer og kolleger ved Children Memorial Hospital, Chicago, for første gang ROHHAD (Rapid-debuterende fedme [Obese] med Hypothalamus-dysfunktion, Hypoventilation og Autonom Dysregulering) i tidsskriftet Pediatrics. I 2007 kendtes kun 15 patienter, tre år senere var yderligere 60 patienter med sygdom-me blevet opsporet.
Den amerikanske pige, Marisa, er bundet til en bærbar lungemaskine. Hun kan ikke leve uden. Hun blev født sund, men som 4-årig fik hun ukontrollabel vægtøgning og alvorlige vejrtrækningsproblemer, der med risiko for krampeanfald eller hjertestop tvang hende til at være bundet til en respirator.
Sygdommen rammer hypothalamus, som er to hjerneområder, der regulerer aktiviteterne i det autonome nervesystem, herunder vejrtrækning, kropstemperatur og fordøjelse.
Hendes tilstand blev stabil, så hun som 5-årig kunne være i børnehave, få danseundervisning og lege med venner.
Det specielle ved hende er, at hendes forældre, Bill og Danielle Carney, har indsamlet et stort fundraising-beløb, 220.000 dollar, til støtte for forskning i denne sygdom. Pengene er indsamlet ved lokale bowling-arrangementer og beskedne golfturneringer, men også ved at bede filantropiske organisationer om penge.
Forskere i Chicago har skaffet en liste over patienter med denne sjældne sygdom, og har derved mulighed for at finde det eventuelle defekte gen. Andre forskere i Childrens Hospital i Boston har sammen med forskere fra Harvard universitet startet en genetisk undersøgelse af seks børn med sygdommen. Boston-hospitalet har et Manton Center for Sjældne Sygdomme.
Da sygdommen har med fedme-området at gøre, har den også generel interesse - hvert femte barn i USA er for fed. Selv om fedme naturligvis blot har noget med motion og kost at gøre, poster National Institutes of Health i USA hvert år næsten 1 milliard dollar i fedmeforskning.
De meget sjældne sygdomme kaldes også “forældreløse sygdomme”, fordi der ikke ved normalt salg af medicin vil være penge nok til at støtte forskning i sygdommen. ca. 3 millioner mennesker lider af 7000 sjældne sygdomme(ref.9514).
Medicinalindustrier, som bruger penge på at forske i sjældne sygdom-me, kan få skattefradrag og forlænget monopolret på medicin. Det har givet større interesse, f.eks. har GlaxoSmithKline og Pfizer etableret afdelinger for forskning i sjældne sygdomme. 400 lægemidler mod sjældne sygdomme er nu på vej.
Der er sket hurtige fremskridt i den genetiske videnskab, hvilket har gjort det lettere at udvikle effektive behandlinger, idet de fleste sjældne sygdomme er forårsaget af genetiske mutationer. Det giver hurtigere afkast af investeringer, så der nu er reel mulighed for at gøre noget for disse patienter, hvilket ikke var muligt før de nye DNA-teknikker.
Test dig selv
Du kan tegne abonnement på BioNyt: Videnskabens verden her!

.
Kilde her
Kilde her
(kilde: N.A. Campbell:”Biology – concepts & connections” 4. udg. 2003)










